class: inverse, left, bottom background-image: url("img/back1.jpg") background-size: cover # **Análisis de datos en R** ---- ## **Orlando Joaqui-Barandica** ### Orlando Joaqui-Barandica ### Universidad del Valle | 2022 --- class: inverse, middle, center background-color: #C0392B <br><br> .center[ <img style="border-radius: 50%;" src="img/avatar.png" width="160px" href="https://www.joaquibarandica.com" /> ### [PhD. Student. Orlando Joaqui-Barandica](https://www.joaquibarandica.com) <br/> ### Universidad del Valle ] <br> .center[ *PhD. Student in Engineering with emphasis in Engineering Industrial* *MSc. Applied Economics* *BSc. Statistic* <i class="fas fa-link fa-spin "></i> [www.joaquibarandica.com](https://www.joaquibarandica.com) ] --- class: inverse center middle # PRESENTACIÓN DEL CURSO <img src="https://media.giphy.com/media/Ssltx68WIeX1wA0Mg8/giphy.gif" width="150%"/> .left[Pulsa <kbd-black>O</kbd-black> para ver el panel de diapositivas] .left[Pulsa <kbd-black>H</kbd-black> para ver otros atajos] --- # Objetivos ¡Bienvenidos! Tomen asiento, entren a la secta del **software libre** y dejen a Excel en la entrada.. .pull-left[ El **objetivo** del curso no es ser un/a experimentado/a programador/a de `R`, sino adquirir los suficientes conocimientos como para lograr 5 objetivos: - **Perder el miedo** a programar. - Entender los [**conceptos básicos de R**](https://cran.rstudio.com/), lenguaje estadístico por excelencia, desde cero. - Dotarnos de **autonomía en el análisis** de datos. - Crear programas y flujos de trabajo **reproducibles** y mantenibles. - Adquirir habilidades en la **visualización de datos** en `R` (haciendo uso también de otras herramientas como [**Datawrapper**](https://www.datawrapper.de/)), incluyendo la visualización de datos en **mapas**. ] .pull-right[ <img src="https://media.giphy.com/media/9ADoZQgs0tyww/giphy.gif"/> ] 📚 Estas **diapositivas** han sido elaboradas con el propio `R` haciendo uso del paquete `{xaringan}` y `{xaringanExtra}`. --- class: inverse middle background-color: #C0392B ### 👉 BLOQUE I. Introducción a R desde cero * Instalación de R y RStudio. Primeros pasos. Tipos de datos. * Datos estructurados: matrices y data.frame * Extras: estructuras de control, proyectos ### 👉 BLOQUE II. Introducción a tidyverse * Funciones * Tibbles y tidy data * Introducción a tidyverse. Relacionando datos (joins). ### 👉 BLOQUE III. Introducción al dataviz en R * Dataviz: introducción histórica. * Introducción a ggplot2 * Dataviz: la importancia de visualizar datos. * Profundizando en ggplot2 ### 👉 BLOQUE IV. Comunicando resultados * Introducción a rmarkdown. --- class: inverse center middle # Bloque I: Introducción a R desde cero. .left[ ### 👉 [Instalación y primeros pasos](#instalacion-primeros-pasos) ### 👉 [Celdas: tipos de datos](#tipos-datos) ### 👉 [Columnas: vectores](#vectores) ### 👉 [Tablas: estructuras de datos](#matrices-dataframes) ### 👉 [Extras: estructuras de control](#estructuras) ] --- name: instalacion-primeros-pasos class: center, middle # Instalación y primeros pasos ## **Instalación de R** Programaremos como escribimos un libro: necesitamos una **gramática** (`R`) y un **Word** (`RStudio`). --- # Requisitos del curso Para el presente curso los únicos **requisitos** serán: 1. **Conexión a internet** (para la descarga de algunos datos y paquetes). 2. **Instalar R**: será nuestro **lenguaje**, nuestro **castellano** para poder «comunicarnos con el ordenador. La descarga la haremos (gratuitamente) desde <https://cran.r-project.org/> 3. **Instalar R Studio**. De la misma manera que podemos escribir castellano en un ordenador, en un Word, en un papel o en un tuit, podemos usar distintos IDE (entornos de desarrollo integrados, nuestro Office), para que el trabajo sea más cómodo. Nuestro **Word** para nosotros será **RStudio**. .left[ <img src = "https://raw.githubusercontent.com/dadosdelaplace/slides-ECI-2022/main/img/cran-R.jpg" alt = "cran-R" align = "left" width = "500" style = "margin-top: 5vh"> ] .right[ <img src = "https://raw.githubusercontent.com/dadosdelaplace/slides-ECI-2022/main/img/R-studio.jpg" alt = "RStudio" align = "right" width = "500" style = "margin-top: 5vh;"> ] --- # Instalación de R El **lenguaje de programación R** será nuestra **gramática**, nuestra ortografía y nuestro diccionario <img src = "https://dadosdelaplace.github.io/courses-ECI-2022/img/pantalla1_cran.jpg" alt = "cran-R" align = "right" width = "500" style = "margin-top: 10vh;margin-right: 0.5rem;margin-left: 1rem;"> * **Paso 1**: entra en la [web oficial de R](https://cran.r-project.org/) y selecciona la instalación acorde a tu sistema operativo. * **Paso 2**: para **Mac** basta con que hacer click en el archivo **.pkg**, y abrirlo una vez descargado. Para sistemas **Windows**, debemos clickar en `install R for the first time` y en la siguiente pantalla hacer click en `Download R for Windows`. Una vez descargado, **abrirlo como cualquier archivo de instalación**. * **Paso 3**: abrir el **ejecutable** de tu escritorio o en tu Launchap (en Windows puede que tengas dos ejecutables i386 y x64, la versión de 32 y de 64 bits; haz click preferiblemente en el de x64). --- # Primera operación Para comprobar que se ha instalado correctamente, tras abrir `R`, deberías ver una **pantalla blanca** similar a esta (en realidad se llama **consola**) <img src = "https://dadosdelaplace.github.io/courses-ECI-2022/img/cranR.jpg" alt = "consola" align = "left" width = "350" style = "margin-top: 5vh;margin-right: 3rem;"> Vamos a escribir **nuestra primera operación** en la consola: * Una variable llamada `a` le asignaremos el valor `1`. En `R` asignaremos valores con `<-`, como una flecha: `a <- 1` significa que a una variable que llamamos a le asignamos el valor 1. * Una variable llamada `b` le asignaremos el valor `2`. * **Sumamos las variables** haciendo `a + b`. ```r # Primera operación a <- 1 # Una variable a con valor --> 1 b <- 2 # Una variable b con valor --> 2 *a + b ``` El **resultado** que nos devuelve la consola será 3 (2 + 1). ``` > [1] 3 ``` --- # Instalación de R Studio El **Word** que usaremos para trabajar y escribir en nuestro lenguaje será **RStudio** (lo que se conoce como un IDE: entorno integrado de desarrollo). <img src = "https://dadosdelaplace.github.io/courses-ECI-2022/img/pantalla_rstudio.jpg" alt = "RStudio" align = "right" width = "450" style = "margin-top: 10vh;margin-right: 2rem;"> * **Paso 1**: entra la [web oficial de RStudio](https://www.rstudio.com/products/rstudio/download/#download) y selecciona la descarga gratuita. * **Paso 2**: selecciona el ejecutable que te aparezca acorde a tu sistema operativo. * **Paso 3**: tras descargar el ejecutable, hay que abrirlo como otro cualquier otro ejecutable y dejar que termine la instalación. --- # Organización de RStudio <img src = "https://dadosdelaplace.github.io/courses-ECI-2022/img/inicio_rstudio_2.jpg" alt = "Rstudio" align = "left" width = "333" style = "margin-top: 3vh;margin-right: 2rem;margin-bottom: 1vh;"> <img src = "https://dadosdelaplace.github.io/courses-ECI-2022/img/inicio_rstudio_3.jpg" alt = "Rstudio" align = "left" width = "283" style = "margin-top: 3vh;margin-right: 2rem;margin-bottom: 1vh;"> <img src = "https://dadosdelaplace.github.io/courses-ECI-2022/img/inicio_rstudio_4.jpg" alt = "Rstudio" align = "left" width = "298" style = "margin-top: 3vh;margin-right: 2rem;margin-bottom: 1vh;"> * **Consola**: es el nombre para llamar a la ventana grande que te ocupa buena parte de tu pantalla. Prueba a escribir el mismo código que antes (la suma) en ella. La consola será donde **ejecutaremos órdenes y mostraremos resultados**. * **Environment (entorno de variables)**: la pantalla pequeña (puedes ajustar los márgenes con el ratón a tu gusto) que tenemos en la parte superior derecha. Nos mostrará las **variables que tenemos definidas, el tipo y su valor**. * **Panel multiusos**: la ventana que tenemos en la parte inferior derecha no servirá para buscar **ayuda de funciones**, además de para **visualizar gráficos**. --- # Tips de RStudio: modo oscuro <img src = "https://dadosdelaplace.github.io/courses-ECI-2022/img/menu_1.jpg" alt = "Rstudio" align = "left" width = "300" style = "margin-top: 3vh;margin-right: 2rem;"> <img src = "https://dadosdelaplace.github.io/courses-ECI-2022/img/menu_2.jpg" alt = "Rstudio" align = "left" width = "350" style = "margin-top: 3vh;margin-right: 2rem;"> **Consejo**: cambiar en tu `RStudio` la tonalidad del fondo de tu programa, en tonos oscuros y no blancos. --- # ¿Qué es R? <img src = "https://logos.turbio.repl.co/rlang.svg" alt = "Rstudio" align = "left" width = "350" style = "margin-top: 3vh;margin-right: 2rem;"> `R` es un **lenguaje estadístico**, creado por y para la estadística, con 4 ventajas fundamentales: * **Software libre** (como C++, Python, Fortran, y otros tantos lenguajes). El software libre no solo tiene una ventaja evidente (es gratis, ok) sino que permite **acceder libremente a código ajeno**. * **Lenguaje modular**: en la instalación que hemos realizado no se han instalado todas las funcionalidades, solo el mínimo para poder funcionar, de forma que se ahorra espacio en disco y en memoria. Al ser software libre, existen trozos de **código hechos por otras personas llamados paquetes**, que podemos ir instalando a nuestro gusto según los vayamos necesitando. -- * **Gran comunidad de usuarios**: `R` tiene una comunidad de usuarios gigante para hacer estadística (Python tiene una enorme comunidad pero más enfocada al Machine Learning), con más de 18 000 paquetes. -- * **Lenguaje de alto nivel**. Los lenguajes de alto nivel, como `R` o `Python`, facilitan la programación al usuario, teniendo que preocuparte solo de la tarea de programar. Son lenguajes con una menor curva de aprendizaje aunque suelen ser más lentos en su ejecución en comparación con lenguajes de bajo nivel (`C`, `C++` o `Fortran`). --- class: inverse center middle COMPRAR un libro --> instalar un paquete (una sola vez) `install.packages()` <figure> <img src = "https://cdn.cienradios.com/wp-content/uploads/sites/14/2020/09/Book-Depository-2.jpg" alt = "comprar-libros" align = "middle" width = "480" style = "margin-top: 1vh;"> </figure> SELECCIONAR un libro (ya comprado) --> acceder a un paquete ya instalado (en cada sesión que queramos usarlo) `library()` <figure> <img src = "https://cdn.sincroguia.tv/uploads/programs/l/a/-/la-biblioteca-de-los-libros-rechazados-704306_SPA-77.jpg" alt = "comprar-libros-2" align = "middle" width = "480" style = "margin-top: 1vh;"> </figure> --- # Paquetes en R Como hemos mencionado, existen trozos de **código hechos por otras personas llamados paquetes**, que podemos ir instalando a nuestro gusto según los vayamos necesitando. A lo largo del curso usaremos varios de esos paquetes, pero el más importante para nuestro objetivo es el paquete `{ggplot2}`, un paquete para la **elaboración de visualizaciones de datos**. Vamos a instalarlo (necesitamos internet para ello) con la orden `install.packages("ggplot")` ```r install.packages("ggplot2") ``` La **instalación de un paquete** es el equivalente a comprar a un libro: solo lo debemos hacer **la primera vez** que lo usemos en un ordenador (descargándose los archivos del paquete a nuestro local). Una vez que tenemos comprado nuestro libro, para poder usarlo, simplemente debemos **indicar al programa que nos lo acerque de la estantería** con `library(ggplot2)`. ```r library(ggplot2) ``` --- class: center middle # ¿Por qué no usar Excel?  --- class: inverse center middle # ¿Por qué no usar Excel? <img src = "https://upload.wikimedia.org/wikipedia/commons/9/92/Soup_Spoon.jpg" alt = "cuchara" align = "middle" width = "600" style = "margin-top: 3vh;"> Excel es una excelente cuchara: puedes ser el mejor partiendo un filete con una cuchara, pero seguirás siendo una persona comiendo filete con cuchara. --- # ¿Por qué no usar Excel? Excel es una **hoja de cálculo**, ni más ni menos, y el propio **Microsoft desaconseja el uso de Excel para el análisis de datos**. El Excel es una herramienta maravillosa para ser usada como una sencilla hoja de cálculo: * Llevar las cuentas de tu familia. * Una declaración de Renta sencilla. * Planificar viajes **NO ESTÁ DISEÑADO** para ser una base de datos, y muchos menos pensado para generar un entorno flexible para el análisis estadístico y la visualización de datos, con algunas desventajas: * **Software de pago** (bien por el usuario, bien por la administración o empresa). * **Software cerrado**: solo podemos hacer lo que Excel ha creído que interesante que podamos hacer. Incluso con la programación de macros, las funcionalidades de Excel siguen siendo mucho más limitadas * **Alto consumo de memoria**. * **No es universal**: no solo es de pago sino que además, dependiendo de la versión que tengas de Excel, tendrá un formato distinto para datos como fechas, teniendo incluso extensiones distintas. --- # Epic fails en Excel .pull-left[<img src = "https://dadosdelaplace.github.io/courses-ECI-2022/img/excel_genes.jpg" alt = "Rstudio" align = "left" width = "250"> <img src = "https://dadosdelaplace.github.io/courses-ECI-2022/img/excel_uk.jpg" alt = "Rstudio" align = "left" width = "250"> <img src = "https://dadosdelaplace.github.io/courses-ECI-2022/img/excel_edades.jpg" alt = "Rstudio" align = "left" width = "250">] .pull-right[ ## Problemas de **versiones** ## Problemas de **memoria** ## Problemas de **codificación** ] --- # Primeros pasos en R: calculadora Empecemos por lo sencillo: **¿cómo usar R como una calculadora?** Si escribimos `2 + 1` en la consola y pulsamos ENTER, la consola nos mostrará el resultado de la suma. ```r 2 + 1 ``` ``` > [1] 3 ``` -- Si dicha suma la quisiéramos utilizar para un segundo cálculo: ¿y si la **almacenamos en alguna variable**? Por ejemplo, vamos a guardar la suma en una variable `x` ```r *x <- 2 + 1 ``` -- Si te fijas ahora `x` aparece definida en nuestro **panel `environment`**, y puede ser usada de nuevo ```r x + 3 ``` ``` > [1] 6 ``` <img src = "https://dadosdelaplace.github.io/courses-ECI-2022/img/environment_1.jpg" alt = "environment" align = "right" width = "450"> --- # Primeros pasos en R: calculadora ### Multiplicación ```r x * y ``` ### Elevar al cuadrado ```r x^2 ``` ### Valor absoluto ```r abs(x) ``` -- ## Errores Durante tu aprendizaje va a ser **muy habitual que las cosas no salgan a la primera**, ni siquiera a la décima, apareciendo en consola mensajes en un color rojo. A **programar se aprende programando**, así que haz las pruebas que quieras: lo peor que puede pasar es que tengas que reiniciar `R`. --- # Mensajes de «error» * **Mensajes de ERROR**: irán precedidos de la frase **«Error in…»**, y serán aquellos **fallos que impidan la ejecución del código** (un error muy habitual es intentar acceder a funciones de algún paquete sin tenerlo instalado: estás intentando leerte un libro de tu biblioteca pero ni siquiera has ido a la tienda a «comprarlo»). Veamos un ejemplo intentando **sumar un número a un texto**. ```r "a" + 1 ``` ``` > Error in "a" + 1: argumento no-numérico para operador binario ``` * Mensajes **WARNING**: irán precedidos de la frase **«Warning in…»**, y son los fallos más delicados ya que son posibles incoherencias pero que no van a hacer que tu código deje de ejecutarse. ```r sqrt(-1) ``` ``` > Warning in sqrt(-1): Se han producido NaNs ``` ``` > [1] NaN ``` **¿Ha ejecutado la orden?** Sí, pero te advierte de que el resultado de la operación es un NaN, **Not A Number**, un valor que no existe (al menos dentro de los números reales). --- # ¿Dónde programamos? Script Un **script** será el documento en el que programamos, nuestro equivalente a un archivo `.doc`, pero aquí será un archivo con extensión `.R`, donde escribiremos las órdenes. Es **importante no abusar de la consola**: todo lo que no escribas en un script, cuando cierres `RStudio`, lo habrás perdido (cómo si en lugar de escribir en un Word y guardarlo, nunca guardases el documento). Para **abrir nuestro primero script**, haz click en el menú superior en `File << New File << R Script`. <img src = "https://dadosdelaplace.github.io/courses-ECI-2022/img/inicio_rstudio_5.jpg" alt = "environment" align = "right" width = "450"> --- # Ejecutar nuestros scripts Ahora tenemos una **cuarta ventana**: la ventana donde **escribiremos nuestros códigos** ### **¿Cómo ejecutar nuestro script?** <img src = "https://dadosdelaplace.github.io/courses-ECI-2022/img/inicio_rstudio_6.jpg" alt = "environment" align = "left" width = "500" style = "margin-top: 3vh;margin-right: 2rem;"> 1. Escribimos el código que queremos ejecutar (en este caso, la suma de antes). 2. Guardamos el archivo `.R` haciendo click en el botón de guardar (`Save current document`) 3. El código **no se ejecuta salvo que se lo indiquemos**. Tenemos tres opciones: - **Copiar y pegar en consola** el código. - **Seleccionar las líneas de código** a ejecutar y clickar en `Run` (o con su atajo). - Activar el cuadrado `Source on save` a la **derecha del botón de guardar**: no solo se nos guarda sino que se ejecuta el código completo. --- # Primeros ejercicios: trasteando con la consola .panelset[ .panel[.panel-name[Ejercicios] * 📝 **Ejercicio 1**: el código inferior define una variable `a` al que le asigna el valor `2`. Añade debajo otra línea para definir una variable `b` con el valor `5`. Tras asignarles valores, multiplica los números en consola. ```r a <- 2 ``` * 📝 **Ejercicio 2**: modifica el código inferior para definir dos variables `c` y `d`, con valores 3 y -1. ```r c <- # deberías asignarle el valor 3 d <- # deberías asignarle el valor -1 ``` * 📝 **Ejercicio 3**: con las variables `a` y `b` del ejercicio 1, crea una nueva variable `e` guardando el resultado de su multiplicación `a * b`. Escribe `e` en consola para ver su resultado ] .panel[.panel-name[Solución ej. 1] ```r # Para poner comentarios en el código se usa # # Definición de variables a <- 2 b <- 5 # Multiplicación a * b ``` ``` > [1] 10 ``` ] .panel[.panel-name[Solución ej. 2] ```r # Definición de variables c <- 3 d <- -1 ``` ] .panel[.panel-name[Solución ej. 3] ```r # Variables a <- 2 b <- 5 # Resultado e <- a * b # Muestro en consola e ``` ``` > [1] 10 ``` ] ] --- name: tipos-datos class: center, middle # Tipos de datos ## **De la celda a la tabla** ¿De qué tipo pueden ser los datos que tenemos contenidos en cada celda de una «tabla»? --- # Datos: de la celda a la tabla <img src = "https://raw.githubusercontent.com/dadosdelaplace/slides-ECI-2022/main/img/celdas.jpg" alt = "celdas" align = "center" width = "850" style = "margin-top: 1vh;"> * **Celda**: un **dato individual** de un tipo concreto. * **Variable**: **concatenación de datos** del mismo tipo. * **Matriz**: **concatenación de variables** del **mismo tipo** y longitud. * **Tabla**: **concatenación de variables** de **distinto tipo** pero igual longitud. --- # Celdas: tipos de datos individuales **¿Existen variables más allá de los números?** Piensa por ejemplo en los **datos guardados de una persona**: * La edad o el peso será un **número**. * Su nombre será una cadena de **texto**. * Su fecha de nacimiento será precisamente eso, una **fecha**. * A la pregunta «¿está usted soltero/a?» la respuesta será lo que llamamos una **variable binaria o lógica** (`TRUE` si está soltero/a o `FALSE` en otro caso). <img src = "https://raw.githubusercontent.com/dadosdelaplace/slides-ECI-2022/main/img/celdas.jpg" alt = "celdas" align = "center" width = "600" style = "margin-top: 1vh;"> --- # Variables numéricas El **dato más sencillo**, dato que ya hemos usado en nuestros primeros pasos como calculadora. ```r a <- 1 b <- 2 a + b ``` En el código anterior, tanto `a` como `b`, como la suma `a + b` **son de tipo numérico** (podemos comprobarlo con `class()`) ```r # Clase de las variables *class(a) ``` ``` > [1] "numeric" ``` **Operaciones**: con los datos numéricos podemos realizar todas las **operaciones aritméticas** que se nos ocurriría hacer en una **calculadora**. ```r b <- 5 b^3 ``` ``` > [1] 125 ``` --- # Variables de texto No solo de números viven los datos: imagina que además de la edad de una persona queremos **guardar su nombre**. ```r edad <- 32 *nombre <- "Juanito" ``` La variable `nombre` es de **tipo caracter**: una **cadena de texto** (conocido en otros lenguajes como string o char) que va **SIEMPRE ENTRE comillas**. ```r *class(nombre) ``` ``` > [1] "character" ``` Las cadenas de texto son un **tipo especial de dato** con los que obviamente no podremos hacer operaciones aritméticas, pero si podemos hacer **otras operaciones** como pegar cadenas de texto o localizar patrones de letras dentro de ellas. --- # Nuestra primera función: paste Una **función** es un **trozo de código encapsulado bajo un nombre**, en función de unos **argumentos de entrada**. Nuestra primera función será `paste()`: dadas **dos cadenas de texto como argumento de entrada** nos permite pegarlas, indicándole en el **argumento** `sep = ` el caracter que queremos entre medias. ```r # todo junto, sin espacios, igual a paste0("Juanito", "Perez") paste("Juanito", "Perez", sep = "") ``` ``` > [1] "JuanitoPerez" ``` ```r paste("Juanito", "Perez") # separados por un espacio ``` ``` > [1] "Juanito Perez" ``` ```r paste("Juanito", "Perez", sep = ".") # separados por un punto . ``` ``` > [1] "Juanito.Perez" ``` **Argumentos por defecto**: `paste(nombre, apellido)` es equivalente a `paste(nombre, apellido, sep = " ")`. El argumento `sep` tiene un **valor por defecto** `sep = " "`: si no se le asigna otro, tomará ese por defecto. --- # Nuestro primer paquete: glue Otra forma **más intuitiva de trabajar con textos** es usar el **paquete** `{glue}`. ```r install.packages("glue") # solo la primera vez library(glue) ``` Recuerda que `install.packages()` es solo necesario la primera que «compramos el libro»: cada vez que queramos usarlo bastará con `library()`. Con dicho paquete podemos **usar variables dentro de cadenas de texto**. Por ejemplo, vamos a crear «la edad es de ... años», donde la edad concreta la tenemos guardada en una variable `edad`. ```r edad <- 32 glue("La edad es de {edad} años") ``` ``` > La edad es de 32 años ``` --- # Variables lógicas Una **variable lógica, binaria o booleana** es aquella que **solo puede tomar dos valores** (en realidad pueden tomar un tercer valor, ausente): * `TRUE`, **verdadero**, guardado internamente como un 1. * `FALSE`, **falso**, guardado internamente como un 0. ```r soltero <- TRUE # ¿Es soltero? --> SÍ class(soltero) ``` ``` > [1] "logical" ``` **NO son variables de texto**: "TRUE" es un texto (como rojo o azul), `TRUE` es una valor lógico. ```r TRUE + 1 ``` ``` > [1] 2 ``` ```r "TRUE" + 1 ``` ``` > Error in "TRUE" + 1: argumento no-numérico para operador binario ``` --- # Condiciones lógicas Los valores lógicos suelen ser **resultado de evaluar condiciones lógicas**. Por ejemplo, imaginemos que queremos comprobar si una persona está o no soltero, con el **operador lógico** `==` (¿lo de la izquierda es igual a lo de la derecha) y su opuesto `!=` (distinto de). ```r soltero <- TRUE # persona soltera soltero == TRUE # ¿está soltero? ``` ``` > [1] TRUE ``` ```r soltero != TRUE # ¿no está soltero? ``` ``` > [1] FALSE ``` Es importante **distinguir** una asignación con `<-` (una variable pasará a tener almacenado dicho valor) a una **comparación lógica** con `==` (no estamos asignando nada, estamos preguntando si la parte de la izquierda es igual a la de la derecha). --- # Condiciones lógicas Por el mismo razonamiento podemos **comparar si una variable numérica** cumple una condición. **¿Tiene la persona menos de 32 años?** ```r *edad <- 38 edad < 32 # ¿Es la edad menor de 32 años? ``` ``` > [1] FALSE ``` Además de las comparaciones «igual a» frente «distinto», también comparaciones de orden como `<, <=, > o >=`. ```r edad <= 38 ``` ``` > [1] TRUE ``` ```r edad > 38 ``` ``` > [1] FALSE ``` --- # Combinar condiciones lógicas Las **condiciones lógicas pueden ser combinadas**, principalmente de dos maneras: * **Intersección**: **todas** las condiciones concatenadas se deben cumplir (conjunción y, operador `&`) para devolver un `TRUE`. * **Unión**: basta con que **una** de las condiciones concatenadas se cumpla (conjunción o, operador `|`) para devolver un `TRUE`. Ejemplo: podríamos preguntarnos si la persona tiene menos de 32 años y está soltero (AMBAS deben cumplirse). ```r edad < 32 & soltero ``` ``` > [1] FALSE ``` El resultado es `FALSE` ya que solo se cumple una de las condiciones. Si nos bastase con una («¿está soltero y/o tiene menos de 32 años?»), el valor devuelto sería `TRUE` ```r edad < 32 | soltero ``` ``` > [1] TRUE ``` --- # Variables de tipo fecha Las **variables de tipo fecha** son de un tipo muy especial: no son una simple cadena de texto "2021-04-21", representan un **instante temporal**. ```r # Cadena de texto fecha_char <- "2021-04-21" fecha_char + 1 ``` ``` > Error in fecha_char + 1: argumento no-numérico para operador binario ``` Para **convertirlo a una fecha** podemos usar la función `as.Date()` ```r fecha_date <- as.Date("2021-04-21") fecha_date ``` ``` > [1] "2021-04-21" ``` ```r fecha_date + 1 ``` ``` > [1] "2021-04-22" ``` Dado que internamente es un número, **podemos restar fechas** (días entre ambas), podemos sumar números a fechas (fecha días después), etc. ```r as.Date("2021-04-21") - as.Date("2021-02-15") ``` ``` > Time difference of 65 days ``` --- # Funcionalidades de fechas **¿Cómo obtener automáticamente la fecha de hoy?** ```r Sys.Date() ``` ``` > [1] "2022-04-04" ``` **¿Cómo convertir una cadena de texto a fecha?** Basta usar la función `as.Date()`, pasándole como argumento la fecha y su formato . ```r as.Date("10-03-2020", "%d-%m-%Y") # con día-mes-año (4 cifras) ``` ``` > [1] "2020-03-10" ``` ```r as.Date("10-03-20", "%d-%m-%y") # con día-mes-año (2 cifras) ``` ``` > [1] "2020-03-10" ``` Dentro del **paquete `{lubridate}` tenemos bastantes **funciones útiles** para trabajar con fechas como las siguientes. --- # Paquete lubridate: fechas En `R` tenemos un paquete muy útil para el manejo de fechas, el paquete `{lubridate}` ```r install.packages("lubridate") # solo la primera vez library(lubridate) ``` Con `today()` por ejemplo podemos obtener directamente la fecha actual. ```r today() ``` ``` > [1] "2022-04-04" ``` Con `now()` puedes obtener la fecha y hora actual. ```r now() ``` ``` > [1] "2022-04-04 17:33:38 -05" ``` --- # Paquete lubridate: fechas Con `year()`, `month()` o `day()` podemos extraer el año, mes y día de un dato en tipo fecha. ```r fecha <- today() year(fecha) ``` ``` > [1] 2022 ``` ```r month(fecha) ``` ``` > [1] 4 ``` ```r day(fecha) ``` ``` > [1] 4 ``` También tenemos funciones como `ymd()` o `my()` para **convertir texto o números** a datos de tipo **fecha**. ```r ymd(20170131) ``` ``` > [1] "2017-01-31" ``` ```r my("jan-2021") ``` ``` > [1] "2021-01-01" ``` --- # Paquete lubridate: fechas <div class="figure" style="text-align: center"> <img src="https://raw.githubusercontent.com/rstudio/cheatsheets/main/pngs/thumbnails/lubridate-cheatsheet-thumbs.png" alt="Imagen extraída de https://lubridate.tidyverse.org/" width="97%" /> <p class="caption">Imagen extraída de https://lubridate.tidyverse.org/</p> </div> --- # Paquete stringr El **paquete `{stringr}`** permite un **manejo más complejo de cadenas de texto** (como el uso de **expresiones regulares**). <div class="figure" style="text-align: center"> <img src="https://dadosdelaplace.github.io/courses-ECI-2022/img/stringr.png" alt="Imagen extraída de https://stringr.tidyverse.org/" width="87%" /> <p class="caption">Imagen extraída de https://stringr.tidyverse.org/</p> </div> --- # Tips Si haces click en la consola y pulsas la flecha «arriba» del teclado, te irá apareciendo todo el **historial de órdenes ejecutadas**. Es una manera de ahorrar tiempo para ejecutar órdenes similares a las ya ejecutadas. Si empiezas a escribir el nombre de una variable pero no te acuerdas exactamente de su nombre, pulsando tabulador te autocompletará solo. Siempre que veas el símbolo `>` como última línea en la consola significa que está listo para que le escribamos otra orden. <img src = "https://dadosdelaplace.github.io/courses-ECI-2022/img/tip_consola_lista.jpg" alt = "tip-consola" align = "right" width = "750"> --- # Ejercicios .panelset[ .panel[.panel-name[Ejercicios] * 📝 **Ejercicio 1**: define una variable que guarde tu edad y otra con tu nombre * 📝 **Ejercicio 2**: define otra variable que responda la pregunta «¿tengo hermanos?» y otra con la fecha de tu nacimiento. * 📝 **Ejercicio 3**: define otra variable con tus apellidos. Junta con `paste()` o `glue()` las variables `nombre` y `apellidos` en una sola variable `nombre_completo`. * 📝 **Ejercicio 4**: calcula los días que han pasado desde la fecha de tu nacimiento, haciendo la resta entre la fecha de hoy (`today()`) y la fecha de nacimiento definida en el ejercicio 2. * 📝 **Ejercicio 5**: obtén una variable lógica que nos diga si se cumplen (todas) las condiciones i) menor de 30 años (`edad < 30`); ii) con hermanos (`hermanos == TRUE`); iii) nacido en 1990 o posterior (`fecha_nacimiento >= as.Date("1990-01-01")`). * 📝 **Ejercicio 6**: modifica el código del ejercicio anterior para obtener una variable lógica que nos diga si se cumplen (al menos) ALGUNA de las condiciones. ] .panel[.panel-name[Solución ej. 1] ```r edad <- 32 # tipo numeric class(edad) ``` ``` > [1] "numeric" ``` ```r nombre <- "Juanito" # tipo caracter class(nombre) ``` ``` > [1] "character" ``` ] .panel[.panel-name[Solución ej. 2] ```r hermanos <- TRUE # tipo hermanos class(hermanos) ``` ``` > [1] "logical" ``` ```r fecha_nacimiento <- as.Date("1989-09-10") # tipo fecha class(fecha_nacimiento) ``` ``` > [1] "Date" ``` ] .panel[.panel-name[Solución ej. 3] ```r # Nombre nombre <- "Juanito" # Apellidos apellidos <- "Perez Liébana" # Pegamos library(glue) nombre_completo <- glue("{nombre} {apellidos}") nombre_completo ``` ``` > Juanito Perez Liébana ``` ```r # Otra forma nombre_completo <- paste(nombre, apellidos) nombre_completo ``` ``` > [1] "Juanito Perez Liébana" ``` ] .panel[.panel-name[Solución ej. 4] ```r library(lubridate) fecha_nacimiento <- as.Date("1989-09-10") today() - fecha_nacimiento ``` ``` > Time difference of 11894 days ``` ] .panel[.panel-name[Solución ej. 5] ```r # Se tienen que cumplir todas edad < 30 & fecha_nacimiento >= as.Date("1990-01-01") & hermanos ``` ``` > [1] FALSE ``` ```r # otra forma edad < 30 & fecha_nacimiento >= as.Date("1990-01-01") & hermanos == TRUE ``` ``` > [1] FALSE ``` ] .panel[.panel-name[Solución ej. 6] ```r # Se tienen que cumplir todas edad < 30 | fecha_nacimiento >= as.Date("1990-01-01") | hermanos ``` ``` > [1] TRUE ``` ] ] --- name: vectores class: center, middle # Vectores ## **Variables: colección de datos individuales de igual tipo** ¿Cómo **concatenar** edades de varias personas? ¿Cómo ordenarlas? --- # Vectores: concatenar elementos Los **vectores o arrays** no son más que una **concatenación de elementos del mismo tipo** (de hecho un número individual `x <- 1` es en realidad un vector de longitud uno). Los crearemos con `c()` (c de concatenar), con sus **elementos entre paréntesis y separados por comas**. ```r edades <- c(32, 27, 60, 61) edades ``` ``` > [1] 32 27 60 61 ``` <img src = "https://dadosdelaplace.github.io/courses-ECI-2022/img/vectores_enviroment.jpg" alt = "stringr" align = "right" width = "350"> La **longitud de un vector** se puede calcular con `length()` ```r length(edades) ``` ``` > [1] 4 ``` Dado que un número y un vector es lo mismo (con distinta longitud), podemos **concatenar a vectores uno tras otro**. ```r c(edades, edades, 8) ``` ``` > [1] 32 27 60 61 32 27 60 61 8 ``` --- # Secuencias numéricas Es habitual **crear vectores numéricos con un patrón** repetido. Hay un **atajo**: `seq()` nos permite crear una **secuencia desde un elemento inicial hasta un elemento final**, avanzando de uno en uno. ```r seq(1, 11) # secuencia desde 1 hasta 11 de 1 en 1 (equivalente: 1:11) ``` ``` > [1] 1 2 3 4 5 6 7 8 9 10 11 ``` ```r 11:1 # orden inverso ``` ``` > [1] 11 10 9 8 7 6 5 4 3 2 1 ``` También podemos definir **otro tipo de distancia entre dos elementos consecutivos** ```r seq(1, 7, by = 0.5) # secuencia desde 1 a 7 de 0.5 en 0.5 ``` ``` > [1] 1.0 1.5 2.0 2.5 3.0 3.5 4.0 4.5 5.0 5.5 6.0 6.5 7.0 ``` Otras veces nos interesará definir una secuencia con un **número concreto de elementos**. ```r seq(1, 50, l = 7) # secuencia desde 1 a 50 de longitud 11 ``` ``` > [1] 1.000000 9.166667 17.333333 25.500000 33.666667 41.833333 50.000000 ``` --- # Secuencias numéricas **Vectores repetidos**: otro atajo, para definir vectores de elementos repetidos, es la función `rep()` nos permite repetir un elemento un número fijado de veces. ```r rep(0, 7) # vector de 7 ceros ``` ``` > [1] 0 0 0 0 0 0 0 ``` No solo podemos repetir un número sino que podemos **repetir vectores enteros**. ```r rep(c(0, 1, 2), 4) # repetimos el vector c(0, 1, 2) 4 veces ``` ``` > [1] 0 1 2 0 1 2 0 1 2 0 1 2 ``` Esa repetición además podemos definirla también de **forma intercalada** ```r rep(c(0, 1, 2), each = 4) # cuatro 0, luego cuatro 1, luego cuatro 2 ``` ``` > [1] 0 0 0 0 1 1 1 1 2 2 2 2 ``` --- # Vectores de texto El concepto de vectores **no solo atañe a los números** ```r mi_nombre <- c("Mi", "nombre", "es", "Juanito") mi_nombre ``` ``` > [1] "Mi" "nombre" "es" "Juanito" ``` Cuando tenemos un vector de caracteres podemos colapsarlos con `paste()`, esta vez con `collapse = ...` ```r paste(mi_nombre, collapse = ".") # separados por un punto . ``` ``` > [1] "Mi.nombre.es.Juanito" ``` Podemos también combinar vectores numéricos con caracteres. ```r glue("persona_{1:5}") # separado por una barra baja ``` ``` > persona_1 > persona_2 > persona_3 > persona_4 > persona_5 ``` --- # Vectores lógicos Los **vectores lógicos** suelen aparecer de nuevo como la evaluación de condiciones lógicas. Por ejemplo, si definimos el vector `x <- c(1.5, -1, 2, 4, 3, -4)`, ¿qué numeros de x son menores que 2? ```r x <- c(1.5, -1, 2, 4, 3, -4) x < 2 ``` ``` > [1] TRUE TRUE FALSE FALSE FALSE TRUE ``` El primer, segundo y sexto elemento del vector son los únicos elementos (estrictamente) menores que 2, de ahí que en el primer, segundo y sexto elemento aparezca un TRUE y en el resto un FALSE. Al igual que antes, las condiciones pueden ser **combinadas** ```r x <- c(1.5, -1, 2, 4, 3, -4) x < 3 & x > 0 # Solo los que cumplen ambas condiciones ``` ``` > [1] TRUE FALSE TRUE FALSE FALSE FALSE ``` --- # Datos ausentes * **NA** (not available): valores que no tenemos (por ejemplo, el dato de contagios covid los fines de semana). ```r x <- c(1, NA, 3, NA, NA, 5, 6) # Vector numérico con datos faltante x ``` ``` > [1] 1 NA 3 NA NA 5 6 ``` ```r 2 * x ``` ``` > [1] 2 NA 6 NA NA 10 12 ``` -- * **NaN** (not a number): resultado no permitido ```r sqrt(-1) ``` ``` > [1] NaN ``` ```r x <- c(1, NA, 3, 4, 6, 7, NaN, NA) is.na(x) ``` ``` > [1] FALSE TRUE FALSE FALSE FALSE FALSE TRUE TRUE ``` --- # Operaciones aritméticas con vectores Toda **operación aritmética** que podamos hacer con un número la vamos a poder a hacer con un vector de números: la operación se realizará en **CADA ELEMENTO** del vector (en otros lenguajes no siempre es así). ```r # Multiplicamos por 2 a CADA ELEMENTO del vector z <- c(2, 4, 6) 2 * z ``` ``` > [1] 4 8 12 ``` De la misma manera se pueden definir **sumas** de vectores. ```r x <- c(1, 3, 5) x + z ``` ``` > [1] 3 7 11 ``` -- Dado que los valores lógicos son guardados internamente como `0/1` podemos usar operaciones aritméticas con ellos. ```r x <- c(1, 3, 5) sum(x < 2) # sumamos el vector lógico --> número de valores TRUE ``` ``` > [1] 1 ``` --- # Operaciones estadísticas con vectores También podemos realizar **operaciones estadísticas** como la suma, la media o la mediana. ```r y <- c(1, -4, 5, 0, 7) sum(y) # suma ``` ``` > [1] 9 ``` ```r mean(y) # media ``` ``` > [1] 1.8 ``` **IMPORTANTE**: las operaciones con ausentes darán como resultado ausente (salvo que los eliminemos antes con `na.rm = TRUE`). ```r y <- c(1, 2, NA, -5, 0) sum(y) ``` ``` > [1] NA ``` ```r sum(y, na.rm = TRUE) ``` ``` > [1] -2 ``` --- # Seleccionar elementos ¿Y si del vector original queremos **EXTRAER UN SUBCONJUNTO**? La forma más sencilla es **acceder al elemento i-ésimo** con el operador de selección `[i]`, o en base a una **condición lógica** ```r edades <- c(20, 30, 32, NA, 61) edades[3] # accedemos a la edad de la tercera persona ``` ``` > [1] 32 ``` ```r *edades[c(3, 4)] ``` ``` > [1] 32 NA ``` ```r edades[edades > 30] ``` ``` > [1] 32 NA 61 ``` Otras veces no querremos seleccionar un elemento sino **filtrarlo**, con el operador `[-i]` ```r edades[-1] ``` ``` > [1] 30 32 NA 61 ``` --- # Nombrando vectores `R` nos permite dar **significado léxico a nuestros datos**, pudiendo poner **nombres a los elementos** de un vector. ```r x <- c("edad" = 31, "tlf" = 613910687, "cp" = 33007) # cada número tiene un significado distinto x ``` ``` > edad tlf cp > 31 613910687 33007 ``` Esto es una ventaja ya que nos permite su **selección usando dichos nombres**: los números ya representan algo. ```r x[c("edad", "cp")] # seleccionamos los elementos que tienen ese nombre asignado ``` ``` > edad cp > 31 33007 ``` Con la función `names()` podemos, no solo consultar sino cambiar los nombres. ```r names(x) # Consultamos nombres antiguos ``` ``` > [1] "edad" "tlf" "cp" ``` ```r names(x) <- c("años", "móvil", "dirección") # Cambiamos nombres ``` --- # Ordenar vectores Una acción **muy habitual** es **ordenar los datos**: de menor a mayor edad, datos más recientes vs antiguos, etc. Para ello tenemos la función `sort()`: vamos a ordenar, por ejemplo, una colección de edades. ```r edades <- c(81, 7, 25, 41, 65, 20, 32, 23, 77) sort(edades) # orden de joven a mayor ``` ``` > [1] 7 20 23 25 32 41 65 77 81 ``` Por defecto `sort()` ordena de menor a mayor: con `decreasing = TRUE` podemos ordenar de mayor a menor. ```r sort(edades, decreasing = TRUE) ``` ``` > [1] 81 77 65 41 32 25 23 20 7 ``` Otra forma de ordenar un vector es **pedirle nos devuelva los índices de los elementos ordenados** con `order`, y luego usar dichos índices. ```r x <- c(7, 1, 2, 6) x[order(x)] ``` ``` > [1] 1 2 6 7 ``` --- # Ejercicios con vectores .panelset[ .panel[.panel-name[Ejercicios] * 📝 **Ejercicio 1**: modifica el código inferior para crear un vector de nombre `vector_num` que contenga los números 1, 5 y -7. ```r # Vector de números vector_num <- c(1) ``` * 📝 **Ejercicio 2**: define un vector que contenga los números `1, 10, -1 y 2`, y obtén su longitud. * 📝 **Ejercicio 3**: crea un vector con las palabras "Hola", "me", "llamo" (y tu nombre y apellidos), y pega luego sus elementos de forma que la frase esté correctamente escrita en castellano. Tras hacerlo, añade "y tengo 30 años". * 📝 **Ejercicio 4**: crea una secuencia que empiece en 1 y recorra todos los naturales hasta el 10. Después crea otra secuencia de longitud 7 en la que todos los números sean 3. * 📝 **Ejercicio 5**: crea un vector con las edades de cuatro conocidos o familiares. Tras ello, determina cuáles de ellos tienen menos de 20 años, 30 años o más, menos de 40 años y más de 65 años. ] .panel[.panel-name[Solución ej. 1] ```r # Vector de números vector_num <- c(1, 5, -7) vector_num ``` ``` > [1] 1 5 -7 ``` ] .panel[.panel-name[Solución ej. 2] ```r # Vector de números vector_num <- c(1, 10, -1, 2) length(vector_num) ``` ``` > [1] 4 ``` ] .panel[.panel-name[Solución ej. 3] ```r # Definiendo el vector vector_char <- c("Hola", "me", "llamo", "Juanito", "Perez", "Liébana") # Pegamos frase <- paste(vector_char, collapse = " ") frase ``` ``` > [1] "Hola me llamo Juanito Perez Liébana" ``` ```r # Añadimos frase glue("{frase} y tengo 30 años.") ``` ``` > Hola me llamo Juanito Perez Liébana y tengo 30 años. ``` ] .panel[.panel-name[Solución ej. 4] ```r 1:10 ``` ``` > [1] 1 2 3 4 5 6 7 8 9 10 ``` ```r rep(3, 7) # secuencia repetida de treses ``` ``` > [1] 3 3 3 3 3 3 3 ``` ] .panel[.panel-name[Solución ej. 5] ```r edades <- c(27, 32, 60, 61) # en mi caso, por ejemplo edades < 20 # menos de 20 años ``` ``` > [1] FALSE FALSE FALSE FALSE ``` ```r edades >= 30 # 30 años o más ``` ``` > [1] FALSE TRUE TRUE TRUE ``` ```r edades < 40 # menos de 40 años ``` ``` > [1] TRUE TRUE FALSE FALSE ``` ] ] --- name: matrices-dataframes class: center, middle # Datos estructurados: colección de variables ## **matrices: igual tipo y longitud** ## **data.frame o tabla: distinto tipo pero igual longitud** --- # Datos estructurados: colección de variables <img src = "https://raw.githubusercontent.com/dadosdelaplace/slides-ECI-2022/main/img/celdas.jpg" alt = "celdas" align = "center" width = "580" style = "margin-top: 1vh;"> Ya sabemos: ✅ Los **tipos de datos** que puede contener una **celda**. ✅ Como **concatenar celdas** obteniendo variables (vectores, datos del mismo tipo). Lo que haremos a continuación será **juntar esas variables**: bien del **mismo tipo** (matrices), bien de **distinto tipo** (`data.frame`), pero en cualquier caso de **igual longitud**. --- # Matrices: variables del mismo tipo Cuando analizamos datos solemos tener **varias variables distintas de cada individuo**. Necesitamos una «tabla» que una distintas variables (de IGUAL longitud). Las **matrices** son una **concatenación de variables, del mismo tipo e igual longitud**, dispuestas en columnas (normalmente cada fila representa un individuo y cada columna una variable). La concatenación en columnas la haremos con `cbind()`. ```r estaturas <- c(150, 160, 170, 180) pesos <- c(60, 70, 80, 90) datos_matriz <- cbind(estaturas, pesos) # Construimos la matriz por columnas datos_matriz # nuestra primera matriz ``` ``` > estaturas pesos > [1,] 150 60 > [2,] 160 70 > [3,] 170 80 > [4,] 180 90 ``` | estaturas| pesos| |---------:|-----:| | 150| 60| | 160| 70| | 170| 80| | 180| 90| --- # Matrices También podemos **construir la matriz por filas** con `rbind()`, que nos permite **añadir filas a una matriz o construirla desde cero** (aunque lo habitual es tener cada variable en una columna y cada individuo en una fila). ```r rbind(estaturas, pesos) # Construimos la matriz por filas ``` ``` > [,1] [,2] [,3] [,4] > estaturas 150 160 170 180 > pesos 60 70 80 90 ``` | | | | | | |:---------|---:|---:|---:|---:| |estaturas | 150| 160| 170| 180| |pesos | 60| 70| 80| 90| <img src = "https://dadosdelaplace.github.io/courses-ECI-2022/img/datos_matriz_1.jpg" alt = "course-ECI" align = "right" width = "400" style = "margin-top: 3vh;margin-right: 0.5rem;margin-left: 0.5rem;"> -- Podemos **visualizar la matriz** en un formato «excelizado» con la función `View()` ```r View(datos_matriz) ``` --- # Matrices .panelset[ .panel[.panel-name[Ejemplo] Veamos un ejemplo más jugoso para practicar: una matriz con las edades, teléfonos y códigos postales. ```r edades <- c(14, 56, 31, 20, 68) # vector numérico de longitud 4 tlf <- c(NA, 683839390, 621539732, NA, NA) cp <- c(33007, 28019, 37005, 18003, 28045) # Construimos la matriz por columnas datos_matriz <- cbind(edades, tlf, cp) datos_matriz ``` ``` > edades tlf cp > [1,] 14 NA 33007 > [2,] 56 683839390 28019 > [3,] 31 621539732 37005 > [4,] 20 NA 18003 > [5,] 68 NA 28045 ``` ] .panel[.panel-name[Añadir columna] Con `cbind()` también podemos **añadir una columna** ```r cbind(datos_matriz, "estaturas" = c(160, NA, 181, 165, 173)) ``` ``` > edades tlf cp estaturas > [1,] 14 NA 33007 160 > [2,] 56 683839390 28019 NA > [3,] 31 621539732 37005 181 > [4,] 20 NA 18003 165 > [5,] 68 NA 28045 173 ``` ] .panel[.panel-name[Dimensiones] Y podemos **ver sus dimensiones** con `dim()`, o por separado con `nrow()` y `ncol()` ```r dim(datos_matriz) ``` ``` > [1] 5 3 ``` ```r c(nrow(datos_matriz), ncol(datos_matriz)) ``` ``` > [1] 5 3 ``` Dada una matriz también podemos **«darle vuelta»** (lo que se conoce como **matriz transpuesta**) con `t()`. ```r t(datos_matriz) ``` ``` > [,1] [,2] [,3] [,4] [,5] > edades 14 56 31 20 68 > tlf NA 683839390 621539732 NA NA > cp 33007 28019 37005 18003 28045 ``` ] .panel[.panel-name[matrix()] Igual que a veces es útil generar un vector de elementos repetidos, también podemos definir una **matriz de números repetidos** (por ejemplo, de ceros), con la función `matrix()`, indicándole el número de filas y columnas. ```r matrix(0, nrow = 4, ncol = 2) # 4 filas, 2 columnas, todo 0's ``` ``` > [,1] [,2] > [1,] 0 0 > [2,] 0 0 > [3,] 0 0 > [4,] 0 0 ``` También podemos **definir una matriz a partir de un vector numérico**, reorganizando los valores en forma de matriz, sabiendo que los elementos se van colocando por columnas. ```r z <- matrix(1:15, ncol = 5) # Matriz con el vector 1:5 con 5 columnas (ergo 3 filas) z ``` ``` > [,1] [,2] [,3] [,4] [,5] > [1,] 1 4 7 10 13 > [2,] 2 5 8 11 14 > [3,] 3 6 9 12 15 ``` ] .panel[.panel-name[Operaciones] Con las matrices sucede como con los vectores: cuando **aplicamos una operación aritmética se la estamos aplicando elemento a elemento**, por ejemplo, dividir entre 5 o sumar una constante ```r datos_matriz / 5 ``` ``` > edades tlf cp > [1,] 2.8 NA 6601.4 > [2,] 11.2 136767878 5603.8 > [3,] 6.2 124307946 7401.0 > [4,] 4.0 NA 3600.6 > [5,] 13.6 NA 5609.0 ``` ```r datos_matriz + 3 ``` ``` > edades tlf cp > [1,] 17 NA 33010 > [2,] 59 683839393 28022 > [3,] 34 621539735 37008 > [4,] 23 NA 18006 > [5,] 71 NA 28048 ``` ] .panel[.panel-name[Otras matrices] También podemos **crear matrices de otros tipos de datos**, siempre y cuando las columnas sean del mismo tipo e igual longitud. ```r nombres <- c("Juanito", "Carlos", "María", "Paloma") apellidos <- c("Perez", "García", "Pérez", "Liébana") cbind(nombres, apellidos) ``` ``` > nombres apellidos > [1,] "Juanito" "Perez" > [2,] "Carlos" "García" > [3,] "María" "Pérez" > [4,] "Paloma" "Liébana" ``` ] ] --- # Matrices Las matrices son objetos **bidimensionales**: para acceder a la **fila i-ésima** se usa el operador `[i, ]` (dejando el otro sin rellenar), para acceder a la **columna j-ésima** se usa el operador `[, j]`. Para acceder **conjuntamente al elemento (i, j)** se `[i, j]`. También podemos **acceder por nombres de las columnas**. ```r datos_matriz[2, 3] ``` ``` > cp > 28019 ``` ```r datos_matriz[1, ] # fila 1 ``` ``` > edades tlf cp > 14 NA 33007 ``` ```r datos_matriz[, 3] # columna 3 ``` ``` > [1] 33007 28019 37005 18003 28045 ``` ```r datos_matriz[1, c("edades", "tlf")] # por nombres ``` ``` > edades tlf > 14 NA ``` --- # Ejercicios .panelset[ .panel[.panel-name[Ejercicios] * 📝 **Ejercicio 1**: modifica el código para definir una matriz x de ceros de 3 filas y 7 columnas. ```r # Matriz x <- matrix(0, nrow = 2, ncol = 3) x ``` * 📝 **Ejercicio 2**: a la matriz anterior, suma un 1 a cada número de la matriz y divide el resultado entre 5. * 📝 **Ejercicio 3**: accede a toda la segunda fila de la matriz anterior. Accede a toda la tercera columna de la matriz anterior. Accede al elemento que ocupa la primera fila y la segunda columna. ] .panel[.panel-name[Solución Ej. 1] ```r # Matriz x <- matrix(0, nrow = 3, ncol = 7) x ``` ``` > [,1] [,2] [,3] [,4] [,5] [,6] [,7] > [1,] 0 0 0 0 0 0 0 > [2,] 0 0 0 0 0 0 0 > [3,] 0 0 0 0 0 0 0 ``` ] .panel[.panel-name[Solución Ej. 2] ```r (x + 1) / 5 ``` ``` > [,1] [,2] [,3] [,4] [,5] [,6] [,7] > [1,] 0.2 0.2 0.2 0.2 0.2 0.2 0.2 > [2,] 0.2 0.2 0.2 0.2 0.2 0.2 0.2 > [3,] 0.2 0.2 0.2 0.2 0.2 0.2 0.2 ``` ] .panel[.panel-name[Solución Ej. 3] ```r x[2, ] # toda la segunda fila ``` ``` > [1] 0 0 0 0 0 0 0 ``` ```r x[, 3] # toda la tercera columna ``` ``` > [1] 0 0 0 ``` ```r x[1, 2] # primera fila y segunda columna ``` ``` > [1] 0 ``` ] ] --- # data.frame: tablas **¿Qué sucede si añadimos una columna con los nombres de cada persona en nuestra matriz `datos_matriz`?** En dicha matriz teníamos guardadas las edades, códigos postales y teléfonos de una serie de personas, todas ellas variables numéricas. ```r nombres <- c("Sonia", "Carla", "Pepito", "Carlos") datos_matriz_nueva <- cbind(nombres, datos_matriz) datos_matriz_nueva ``` ``` > nombres edades tlf cp > [1,] "Sonia" "14" NA "33007" > [2,] "Carla" "56" "683839390" "28019" > [3,] "Pepito" "31" "621539732" "37005" > [4,] "Carlos" "20" NA "18003" > [5,] "Sonia" "68" NA "28045" ``` Como una **matriz SOLO puede tener un tipo de dato**, al añadir una variable de tipo texto, `R` se ha visto obligado a convertir los números en texto: hemos **roto la integridad** de los datos. ```r datos_matriz_nueva[, "pesos"] + 1 ``` ``` > Error in datos_matriz_nueva[, "pesos"]: subíndice fuera de los límites ``` --- # data.frame: tablas El formato de tabla de datos en `R` que vamos a empezar a usar se llama `data.frame`, y no es más que una **colección de variables de igual longitud pero cada una puede ser de un tipo distinto**. Para crear un `data.frame` basta con usar la función `data.frame()`. ```r # Nombres nombres <- c("Sonia", "Carla", "Pepito", "Carlos") # Edades edades <- c(45, 67, NA, 31) # Estado civil (no lo sabemos de una persona) casado <- c(TRUE, FALSE, FALSE, NA) # Fecha de creación (fecha en el que esa persona entra en el sistema) # lo convertimos a tipo fecha f_creacion <- as.Date(c("2021-03-04", "2020-10-12", "1990-04-05", "2019-09-10")) # Creamos nuestro primer data.frame tabla <- data.frame(nombres, edades, casado, f_creacion) tabla ``` ``` > nombres edades casado f_creacion > 1 Sonia 45 TRUE 2021-03-04 > 2 Carla 67 FALSE 2020-10-12 > 3 Pepito NA FALSE 1990-04-05 > 4 Carlos 31 NA 2019-09-10 ``` --- # data.frame: tablas Al igual que con las matrices, podemos **crearlos** indicando además el **nombre de las columnas**. ```r tabla <- data.frame("nombre" = nombres, "edad" = edades, "casado" = casado, "fecha_registro" = f_creacion) ``` Si tenemos un `data.frame` ya creado podemos usar `data.frame()` también para añadir una columna. ```r # Añadimos una nueva columna con nº de hermanos/as tabla <- data.frame(tabla, "n_hermanos" = c(0, 0, 1, 5)) ``` Con `View()` podemos **visualizar nuestra tabla**. ```r View(tabla) ``` |nombre | edad|casado |fecha_registro | n_hermanos| |:------|----:|:------|:--------------|----------:| |Sonia | 45|TRUE |2021-03-04 | 0| |Carla | 67|FALSE |2020-10-12 | 0| |Pepito | NA|FALSE |1990-04-05 | 1| |Carlos | 31|NA |2019-09-10 | 5| --- # Selección de columnas y filas Si queremos **acceder a una columna, fila o elemento** en concreto, los `data.frame` tienen las mismas ventajas que una matriz, así que bastaría con usar los mismos operadores. ```r tabla[, 3] # Accedemos a la tercera columna ``` ``` > [1] TRUE FALSE FALSE NA ``` ```r tabla[4, ] # Accedemos a la cuarta fila ``` ``` > nombre edad casado fecha_registro n_hermanos > 4 Carlos 31 NA 2019-09-10 5 ``` No solo tiene las ventajas de una matriz si no que también tiene las ventajas de una **«base» de datos**: podemos **aceder a las variables** por su nombre poniendo el nombre de la tabla más el símbolo `$` y, con el tabulador, nos aparecerá un **menú de columnas** a elegir. <img src = "https://dadosdelaplace.github.io/courses-ECI-2022/img/tabla_dolar.jpg" alt = "course-ECI" align = "right" width = "450" style = "margin-top: 3vh;margin-right: 0.5rem;margin-left: 0.5rem;"> --- # Ejercicios .panelset[ .panel[.panel-name[Ejercicios] * 📝 **Ejercicio 1**: el `data.frame` llamado `airquality`, del paquete `{datasets}`, contiene variables de la calidad del aire de la ciudad de Nueva York desde mayo hasta septiembre de 1973. Obtén el nombre de las variables. * 📝 **Ejercicio 2**: obtén las dimensiones del anterior conjunto de datos. ¿Cuántas variables hay? ¿Cuántos días se han medido? * 📝 **Ejercicio 3**: filtra del conjunto anterior solo los datos del mes de julio. * 📝 **Ejercicio 4**: qúedate solo con las variable Ozone y Temp. * 📝 **Ejercicio 5**: añade a los datos originales una columna con la fecha completa (recuerda que es del año 1973 todas las observaciones. ] .panel[.panel-name[Solución Ej. 1] ```r # install.packages("datasets") si no estuviera ya instalado library(datasets) names(airquality) ``` ``` > [1] "Ozone" "Solar.R" "Wind" "Temp" "Month" "Day" ``` ] .panel[.panel-name[Solución Ej. 2] ```r # Dimensiones dim(airquality) ``` ``` > [1] 153 6 ``` ```r nrow(airquality) ``` ``` > [1] 153 ``` ```r ncol(airquality) ``` ``` > [1] 6 ``` ] .panel[.panel-name[Solución Ej. 3] ```r filtro_filas <- airquality[airquality$Month == 7, ] filtro_filas ``` ``` > Ozone Solar.R Wind Temp Month Day > 62 135 269 4.1 84 7 1 > 63 49 248 9.2 85 7 2 > 64 32 236 9.2 81 7 3 > 65 NA 101 10.9 84 7 4 > 66 64 175 4.6 83 7 5 > 67 40 314 10.9 83 7 6 > 68 77 276 5.1 88 7 7 > 69 97 267 6.3 92 7 8 > 70 97 272 5.7 92 7 9 > 71 85 175 7.4 89 7 10 > 72 NA 139 8.6 82 7 11 > 73 10 264 14.3 73 7 12 > 74 27 175 14.9 81 7 13 > 75 NA 291 14.9 91 7 14 > 76 7 48 14.3 80 7 15 > 77 48 260 6.9 81 7 16 > 78 35 274 10.3 82 7 17 > 79 61 285 6.3 84 7 18 > 80 79 187 5.1 87 7 19 > 81 63 220 11.5 85 7 20 > 82 16 7 6.9 74 7 21 > 83 NA 258 9.7 81 7 22 > 84 NA 295 11.5 82 7 23 > 85 80 294 8.6 86 7 24 > 86 108 223 8.0 85 7 25 > 87 20 81 8.6 82 7 26 > 88 52 82 12.0 86 7 27 > 89 82 213 7.4 88 7 28 > 90 50 275 7.4 86 7 29 > 91 64 253 7.4 83 7 30 > 92 59 254 9.2 81 7 31 ``` ] .panel[.panel-name[Solución Ej. 4] ```r # Filtramos columnas filtro_cols <- airquality[, c("Ozone", "Temp")] filtro_cols ``` ``` > Ozone Temp > 1 41 67 > 2 36 72 > 3 12 74 > 4 18 62 > 5 NA 56 > 6 28 66 > 7 23 65 > 8 19 59 > 9 8 61 > 10 NA 69 > 11 7 74 > 12 16 69 > 13 11 66 > 14 14 68 > 15 18 58 > 16 14 64 > 17 34 66 > 18 6 57 > 19 30 68 > 20 11 62 > 21 1 59 > 22 11 73 > 23 4 61 > 24 32 61 > 25 NA 57 > 26 NA 58 > 27 NA 57 > 28 23 67 > 29 45 81 > 30 115 79 > 31 37 76 > 32 NA 78 > 33 NA 74 > 34 NA 67 > 35 NA 84 > 36 NA 85 > 37 NA 79 > 38 29 82 > 39 NA 87 > 40 71 90 > 41 39 87 > 42 NA 93 > 43 NA 92 > 44 23 82 > 45 NA 80 > 46 NA 79 > 47 21 77 > 48 37 72 > 49 20 65 > 50 12 73 > 51 13 76 > 52 NA 77 > 53 NA 76 > 54 NA 76 > 55 NA 76 > 56 NA 75 > 57 NA 78 > 58 NA 73 > 59 NA 80 > 60 NA 77 > 61 NA 83 > 62 135 84 > 63 49 85 > 64 32 81 > 65 NA 84 > 66 64 83 > 67 40 83 > 68 77 88 > 69 97 92 > 70 97 92 > 71 85 89 > 72 NA 82 > 73 10 73 > 74 27 81 > 75 NA 91 > 76 7 80 > 77 48 81 > 78 35 82 > 79 61 84 > 80 79 87 > 81 63 85 > 82 16 74 > 83 NA 81 > 84 NA 82 > 85 80 86 > 86 108 85 > 87 20 82 > 88 52 86 > 89 82 88 > 90 50 86 > 91 64 83 > 92 59 81 > 93 39 81 > 94 9 81 > 95 16 82 > 96 78 86 > 97 35 85 > 98 66 87 > 99 122 89 > 100 89 90 > 101 110 90 > 102 NA 92 > 103 NA 86 > 104 44 86 > 105 28 82 > 106 65 80 > 107 NA 79 > 108 22 77 > 109 59 79 > 110 23 76 > 111 31 78 > 112 44 78 > 113 21 77 > 114 9 72 > 115 NA 75 > 116 45 79 > 117 168 81 > 118 73 86 > 119 NA 88 > 120 76 97 > 121 118 94 > 122 84 96 > 123 85 94 > 124 96 91 > 125 78 92 > 126 73 93 > 127 91 93 > 128 47 87 > 129 32 84 > 130 20 80 > 131 23 78 > 132 21 75 > 133 24 73 > 134 44 81 > 135 21 76 > 136 28 77 > 137 9 71 > 138 13 71 > 139 46 78 > 140 18 67 > 141 13 76 > 142 24 68 > 143 16 82 > 144 13 64 > 145 23 71 > 146 36 81 > 147 7 69 > 148 14 63 > 149 30 70 > 150 NA 77 > 151 14 75 > 152 18 76 > 153 20 68 ``` ] .panel[.panel-name[Solución Ej. 5] ```r # Construimos las fechas (pegamos año-mes-día con "-") fechas <- as.Date(paste("1973", airquality$Month, airquality$Day, sep = "-")) # Añadimos data.frame(airquality, fechas) ``` ``` > Ozone Solar.R Wind Temp Month Day fechas > 1 41 190 7.4 67 5 1 1973-05-01 > 2 36 118 8.0 72 5 2 1973-05-02 > 3 12 149 12.6 74 5 3 1973-05-03 > 4 18 313 11.5 62 5 4 1973-05-04 > 5 NA NA 14.3 56 5 5 1973-05-05 > 6 28 NA 14.9 66 5 6 1973-05-06 > 7 23 299 8.6 65 5 7 1973-05-07 > 8 19 99 13.8 59 5 8 1973-05-08 > 9 8 19 20.1 61 5 9 1973-05-09 > 10 NA 194 8.6 69 5 10 1973-05-10 > 11 7 NA 6.9 74 5 11 1973-05-11 > 12 16 256 9.7 69 5 12 1973-05-12 > 13 11 290 9.2 66 5 13 1973-05-13 > 14 14 274 10.9 68 5 14 1973-05-14 > 15 18 65 13.2 58 5 15 1973-05-15 > 16 14 334 11.5 64 5 16 1973-05-16 > 17 34 307 12.0 66 5 17 1973-05-17 > 18 6 78 18.4 57 5 18 1973-05-18 > 19 30 322 11.5 68 5 19 1973-05-19 > 20 11 44 9.7 62 5 20 1973-05-20 > 21 1 8 9.7 59 5 21 1973-05-21 > 22 11 320 16.6 73 5 22 1973-05-22 > 23 4 25 9.7 61 5 23 1973-05-23 > 24 32 92 12.0 61 5 24 1973-05-24 > 25 NA 66 16.6 57 5 25 1973-05-25 > 26 NA 266 14.9 58 5 26 1973-05-26 > 27 NA NA 8.0 57 5 27 1973-05-27 > 28 23 13 12.0 67 5 28 1973-05-28 > 29 45 252 14.9 81 5 29 1973-05-29 > 30 115 223 5.7 79 5 30 1973-05-30 > 31 37 279 7.4 76 5 31 1973-05-31 > 32 NA 286 8.6 78 6 1 1973-06-01 > 33 NA 287 9.7 74 6 2 1973-06-02 > 34 NA 242 16.1 67 6 3 1973-06-03 > 35 NA 186 9.2 84 6 4 1973-06-04 > 36 NA 220 8.6 85 6 5 1973-06-05 > 37 NA 264 14.3 79 6 6 1973-06-06 > 38 29 127 9.7 82 6 7 1973-06-07 > 39 NA 273 6.9 87 6 8 1973-06-08 > 40 71 291 13.8 90 6 9 1973-06-09 > 41 39 323 11.5 87 6 10 1973-06-10 > 42 NA 259 10.9 93 6 11 1973-06-11 > 43 NA 250 9.2 92 6 12 1973-06-12 > 44 23 148 8.0 82 6 13 1973-06-13 > 45 NA 332 13.8 80 6 14 1973-06-14 > 46 NA 322 11.5 79 6 15 1973-06-15 > 47 21 191 14.9 77 6 16 1973-06-16 > 48 37 284 20.7 72 6 17 1973-06-17 > 49 20 37 9.2 65 6 18 1973-06-18 > 50 12 120 11.5 73 6 19 1973-06-19 > 51 13 137 10.3 76 6 20 1973-06-20 > 52 NA 150 6.3 77 6 21 1973-06-21 > 53 NA 59 1.7 76 6 22 1973-06-22 > 54 NA 91 4.6 76 6 23 1973-06-23 > 55 NA 250 6.3 76 6 24 1973-06-24 > 56 NA 135 8.0 75 6 25 1973-06-25 > 57 NA 127 8.0 78 6 26 1973-06-26 > 58 NA 47 10.3 73 6 27 1973-06-27 > 59 NA 98 11.5 80 6 28 1973-06-28 > 60 NA 31 14.9 77 6 29 1973-06-29 > 61 NA 138 8.0 83 6 30 1973-06-30 > 62 135 269 4.1 84 7 1 1973-07-01 > 63 49 248 9.2 85 7 2 1973-07-02 > 64 32 236 9.2 81 7 3 1973-07-03 > 65 NA 101 10.9 84 7 4 1973-07-04 > 66 64 175 4.6 83 7 5 1973-07-05 > 67 40 314 10.9 83 7 6 1973-07-06 > 68 77 276 5.1 88 7 7 1973-07-07 > 69 97 267 6.3 92 7 8 1973-07-08 > 70 97 272 5.7 92 7 9 1973-07-09 > 71 85 175 7.4 89 7 10 1973-07-10 > 72 NA 139 8.6 82 7 11 1973-07-11 > 73 10 264 14.3 73 7 12 1973-07-12 > 74 27 175 14.9 81 7 13 1973-07-13 > 75 NA 291 14.9 91 7 14 1973-07-14 > 76 7 48 14.3 80 7 15 1973-07-15 > 77 48 260 6.9 81 7 16 1973-07-16 > 78 35 274 10.3 82 7 17 1973-07-17 > 79 61 285 6.3 84 7 18 1973-07-18 > 80 79 187 5.1 87 7 19 1973-07-19 > 81 63 220 11.5 85 7 20 1973-07-20 > 82 16 7 6.9 74 7 21 1973-07-21 > 83 NA 258 9.7 81 7 22 1973-07-22 > 84 NA 295 11.5 82 7 23 1973-07-23 > 85 80 294 8.6 86 7 24 1973-07-24 > 86 108 223 8.0 85 7 25 1973-07-25 > 87 20 81 8.6 82 7 26 1973-07-26 > 88 52 82 12.0 86 7 27 1973-07-27 > 89 82 213 7.4 88 7 28 1973-07-28 > 90 50 275 7.4 86 7 29 1973-07-29 > 91 64 253 7.4 83 7 30 1973-07-30 > 92 59 254 9.2 81 7 31 1973-07-31 > 93 39 83 6.9 81 8 1 1973-08-01 > 94 9 24 13.8 81 8 2 1973-08-02 > 95 16 77 7.4 82 8 3 1973-08-03 > 96 78 NA 6.9 86 8 4 1973-08-04 > 97 35 NA 7.4 85 8 5 1973-08-05 > 98 66 NA 4.6 87 8 6 1973-08-06 > 99 122 255 4.0 89 8 7 1973-08-07 > 100 89 229 10.3 90 8 8 1973-08-08 > 101 110 207 8.0 90 8 9 1973-08-09 > 102 NA 222 8.6 92 8 10 1973-08-10 > 103 NA 137 11.5 86 8 11 1973-08-11 > 104 44 192 11.5 86 8 12 1973-08-12 > 105 28 273 11.5 82 8 13 1973-08-13 > 106 65 157 9.7 80 8 14 1973-08-14 > 107 NA 64 11.5 79 8 15 1973-08-15 > 108 22 71 10.3 77 8 16 1973-08-16 > 109 59 51 6.3 79 8 17 1973-08-17 > 110 23 115 7.4 76 8 18 1973-08-18 > 111 31 244 10.9 78 8 19 1973-08-19 > 112 44 190 10.3 78 8 20 1973-08-20 > 113 21 259 15.5 77 8 21 1973-08-21 > 114 9 36 14.3 72 8 22 1973-08-22 > 115 NA 255 12.6 75 8 23 1973-08-23 > 116 45 212 9.7 79 8 24 1973-08-24 > 117 168 238 3.4 81 8 25 1973-08-25 > 118 73 215 8.0 86 8 26 1973-08-26 > 119 NA 153 5.7 88 8 27 1973-08-27 > 120 76 203 9.7 97 8 28 1973-08-28 > 121 118 225 2.3 94 8 29 1973-08-29 > 122 84 237 6.3 96 8 30 1973-08-30 > 123 85 188 6.3 94 8 31 1973-08-31 > 124 96 167 6.9 91 9 1 1973-09-01 > 125 78 197 5.1 92 9 2 1973-09-02 > 126 73 183 2.8 93 9 3 1973-09-03 > 127 91 189 4.6 93 9 4 1973-09-04 > 128 47 95 7.4 87 9 5 1973-09-05 > 129 32 92 15.5 84 9 6 1973-09-06 > 130 20 252 10.9 80 9 7 1973-09-07 > 131 23 220 10.3 78 9 8 1973-09-08 > 132 21 230 10.9 75 9 9 1973-09-09 > 133 24 259 9.7 73 9 10 1973-09-10 > 134 44 236 14.9 81 9 11 1973-09-11 > 135 21 259 15.5 76 9 12 1973-09-12 > 136 28 238 6.3 77 9 13 1973-09-13 > 137 9 24 10.9 71 9 14 1973-09-14 > 138 13 112 11.5 71 9 15 1973-09-15 > 139 46 237 6.9 78 9 16 1973-09-16 > 140 18 224 13.8 67 9 17 1973-09-17 > 141 13 27 10.3 76 9 18 1973-09-18 > 142 24 238 10.3 68 9 19 1973-09-19 > 143 16 201 8.0 82 9 20 1973-09-20 > 144 13 238 12.6 64 9 21 1973-09-21 > 145 23 14 9.2 71 9 22 1973-09-22 > 146 36 139 10.3 81 9 23 1973-09-23 > 147 7 49 10.3 69 9 24 1973-09-24 > 148 14 20 16.6 63 9 25 1973-09-25 > 149 30 193 6.9 70 9 26 1973-09-26 > 150 NA 145 13.2 77 9 27 1973-09-27 > 151 14 191 14.3 75 9 28 1973-09-28 > 152 18 131 8.0 76 9 29 1973-09-29 > 153 20 223 11.5 68 9 30 1973-09-30 ``` ] ] --- name: estructuras class: center, middle # Extra: estructuras de control ## **if-else y bucles** A veces necesitaremos algunas estructuras de control para guiar nuestro código --- # Estructuras if-else `if-else` es una de las **estructuras de control** más famosas: **SI** las condiciones impuestas se cumplen (`TRUE`), ejecuta las **órdenes que tengamos dentro del `if {}`. ```r edades <- c(14, 17, 24, 56, 31, 20, 87, 73) if (any(edades < 18)) { # TRUE si al menos una persona mayor de edad print("existe alguna persona mayor de edad") } ``` ``` > [1] "existe alguna persona mayor de edad" ``` ```r if (all(edades >= 18)) { # TRUE si TODOS son mayores de edad print("todas las personas son mayores de edad") } ``` Si no se cumple no hace nada salvo que añadamos un `else {}` (con lo que sucede cuando no sucede) ```r if (all(edades >= 18)) { # TRUE si TODOS son mayores de edad print("todas las personas son mayores de edad") } else { # si hay alguno menor de edad print("existe alguna persona menor de edad") } ``` ``` > [1] "existe alguna persona menor de edad" ``` --- # Estructuras if-else Con `ifelse` podemos plantear una **estructura condicional en formato vectorial**, en una sola línea de código, pasándole tres argumentos: * la condición a cumplir * lo que asignamos cuando se cumple * lo que asignamos cuando no se cumple ```r edades <- c(14, 17, 24, 56, 31, 20, 87, 73) edades_nuevas <- ifelse(edades <= 18, "menor", "mayor") edades_nuevas ``` ``` > [1] "menor" "menor" "mayor" "mayor" "mayor" "mayor" "mayor" "mayor" ``` --- # Bucles (a evitar) * `for`: bucle que permite **repetir** el mismo código un **número fijo** de veces. ```r library(glue) nombres <- c("Javi", "Laura") edades <- c(32, 51) # Recorremos cada uno de los nombres e imprimimos un mensaje que depende de i *for (i in 1:length(nombres)) { print(glue("{nombres[i]} tiene {edades[i]} años")) } ``` ``` > Javi tiene 32 años > Laura tiene 51 años ``` * `while`: **repetir** un **número indeterminado** de veces hasta que se **deje de cumplir una condición**. ```r ciclos <- 1 *while(ciclos <= 3) { print(paste("Todavía no, vamos por el ciclo ", ciclos)) ciclos <- ciclos + 1 } ``` ``` > [1] "Todavía no, vamos por el ciclo 1" > [1] "Todavía no, vamos por el ciclo 2" > [1] "Todavía no, vamos por el ciclo 3" ``` --- # Bucles (a evitar) Los **bucles** en `R` suelen ser **bastante ineficiente** y solemos tener** estructuras más eficientes**. Por ejemplo, imagina que queremos calcular el cuadrado de todos los números desde el 1 hasta el 500. * **Opción sin bucle**: definir la secuencia `1:500` y elevarla al cuadrado con `(1:500)^2` * **Opción con bucle** ```r for (i in 1:1:500) { i^2 } ``` Para comprobar la **eficiencia en tiempo** podemos usar el paquete `{microbenchmark}`: le pasamos las dos órdenes a comparar, y el número de veces `times` que ejecutaremos cada una para calcular tiempos medios. ```r # install.packages("microbenchmark") library(microbenchmark) microbenchmark((1:500)^2, for (i in 1:1:500) {i^2}, times = 300) ``` ``` > Unit: microseconds > expr min lq mean median uq > (1:500)^2 1.7 2.6 7.481333 5.75 8.60 > for (i in 1:1:500) { i^2 } 2035.6 2447.7 4276.133333 3271.40 4916.85 > max neval > 48.1 300 > 64772.5 300 ``` --- class: inverse center middle # Bloque II: Introducción al manejo de datos. .left[ ### 👉 [Funciones](#proyecto-funciones) ### 👉 [Datos tibble](#tibble) ### 👉 [Tidy data: ordenando los datos](#tidy-data) ### 👉 [Introducción a Tidyverse](#tidyverse) ### 👉 [Introducción a purrr (listas)](#purrr) ### 👉 [Relacionando datos: joins](#joins) ] --- name: repaso-1 class: center, middle # Repaso Previously on Breaking Bad...  --- # Repaso: tipos de datos <img src = "https://miro.medium.com/max/727/0*MgEQsDgZZ7fJcdhZ.png" alt = "course-ECI" align = "left" width = "990" style = "margin-top: -5vh;margin-right: 0.5rem;margin-left: 0.5rem;"> https://medium.com/@tiwarigaurav2512 --- class: center, middle # **¿SEGUIMOS?**  --- name: proyecto-funciones class: center, middle # Uso de funciones ## **encapsulando y ordenando nuestro código** Las funciones nos servirán para **encapsular nuestro código** de forma que podamos usarlo muchas veces sin necesidad de escribirlo de nuevo. --- # Funciones No solo podemos usar las funciones predeterminadas, como `sum()` o `paste()`, sino que además podemos **crear nuestras propias funciones**, para automatizar **tareas que vayamos a repetir**, y evitarnos líneas y líneas de código, horas y horas de programación. **¿Cómo crear nuestra propia función?** Veamos su **esquema básico**: * Un **nombre**, por ejemplo `nombre_funcion`. * A dicho nombre le asignamos `<-` la palabra reservada `function()`. * Dentro de `function()` definimos los **argumentos de entrada**. * Dentro de `{}` incluiremos las órdenes. * Finalizaremos la función con `return()` indicando lo que queremos **devolver**. ```r nombre_funcion <- function(argumento_1, argumento_2, ... ) { # Código que queramos ejecutar en la función código # Salida return(variable_salida) } ``` **IMPORTANTE**: las variables que definamos dentro de la función son **locales, solo existirán dentro de la función** salvo que especifiquemos lo contrario. --- # Funciones Un **ejemplo muy simple**: una función para **calcular el área de un rectángulo**. Los **argumentos de entrada** serán los **lados** y el valor a **devolver** será el área (el producto de los lados). ```r # Definición del nombre de función y argumentos de entrada calcular_area <- function(lado_1, lado_2) { # Resultado que devolvemos return(lado_1 * lado_2) } ``` **¿Cómo aplicar la función?** ```r # Aplicación de la función con los parámetros por defecto calcular_area(5, 3) # área de un rectángulo 5 x 3 ``` ``` > [1] 15 ``` --- # Funciones Imagina que nos damos cuenta que el **90% de las veces el área que nos toca calcular fuese la de un cuadrado**, es decir, solo necesitamos un argumento, un lado: `R` nos permite **definir argumentos por defecto** en la función (tomarán dicho valor salvo que le asignemos otro). **¿Por qué no asignar `lado_2 = lado_1` por defecto?** ```r # Definición del nombre de función y argumentos de entrada calcular_area <- function(lado_1, lado_2 = lado_1) { # Cuerpo de la función area <- lado_1 * lado_2 # Resultado que devolvemos return(area) } ``` Ahora, **si no indicamos nada por defecto el segundo lado será igual al primero** (un cuadrado). ```r calcular_area(lado_1 = 5) ``` ``` > [1] 25 ``` --- # Funciones **Compliquemos un poco la función** y añadamos en la salida los valores de cada lado. ```r calcular_area <- function(lado_1, lado_2 = lado_1) { # Cuerpo de la función area <- lado_1 * lado_2 # Resultado return(c("area" = area, "lado_1" = lado_1, "lado_2" = lado_2)) } salida <- calcular_area(lado_1 = 5, lado_2 = 3) salida ``` ``` > area lado_1 lado_2 > 15 5 3 ``` ```r salida["area"] ``` ``` > area > 15 ``` --- # Ejercicios de funciones .panelset[ .panel[.panel-name[Ejercicios] * 📝 **Ejercicio 1**: modifica el código inferior para definir una función llamada `funcion_suma`, de forma que dados dos elementos, devuelve su suma. ```r # Definimos función nombre <- function(x, y) { # Sumamos suma <- # código a ejecutar # ¿Qué devolvemos? return() } ``` * 📝 **Ejercicio 2**: modifica el código para definir una función llamada `funcion_producto`, de forma que dados dos elementos, devuelve su producto. * 📝 **Ejercicio 3**: modifica el código para definir una función llamada `funcion_producto`, pero que por defecto calcule el cuadrado. * 📝 **Ejercicio 4**: define una función llamada igualdad_nombres que, dados dos `nombres persona_1` e `persona_2`, nos diga si son iguales o no. Hazlo considerando importantes las mayúsculas, y sin que importen las mayúsculas. Recuerda que con `toupper()` podemos pasar todo un texto a mayúscula. ] .panel[.panel-name[Solución Ej. 1] ```r # Definimos función funcion_suma <- function(x, y) { # Sumamos suma <- x + y # Devolvemos la salida return(suma) } # Aplicamos la función funcion_suma(3, 7) ``` ``` > [1] 10 ``` ] .panel[.panel-name[Solución Ej. 2] ```r # Definimos función funcion_producto <- function(x, y) { # Multiplicamos producto <- x * y # Devolvemos la salida return(producto) } # Aplicamos la función funcion_producto(3, -7) ``` ``` > [1] -21 ``` ] .panel[.panel-name[Solución Ej. 3] ```r # Definimos función funcion_producto <- function(x, y = x) { # Multiplicamos producto <- x * y # Devolvemos la salida return(producto) } # Aplicamos la función funcion_producto(3) # por defecto x = 3, y = 3 ``` ``` > [1] 9 ``` ```r funcion_producto(3, -7) ``` ``` > [1] -21 ``` ] .panel[.panel-name[Solución Ej. 4] ```r # Distinguiendo mayúsculas igualdad_nombres <- function(persona_1, persona_2) { return(persona_1 == persona_2) } igualdad_nombres("Javi", "javi") ``` ``` > [1] FALSE ``` ```r igualdad_nombres("Javi", "Lucía") ``` ``` > [1] FALSE ``` ```r # Sin importar mayúsculas igualdad_nombres <- function(persona_1, persona_2) { return(toupper(persona_1) == toupper(persona_2)) } igualdad_nombres("Javi", "javi") ``` ``` > [1] TRUE ``` ```r igualdad_nombres("Javi", "Lucía") ``` ``` > [1] FALSE ``` ] ] --- name: tibble class: center, middle # Mejorando los datos: tibble  ## **mejorando los data.frame** Un **dato de tipo tibble** será un `data.frame` mejorado: más ágil, más rápido y más cómodo. --- # Tibble Los **datos en formato tibble** (del paquete `{tibble}` incluido ya en `{tidyverse}`) son un tipo de `data.frame` mejorado, para una **gestión más ágil, eficiente y coherente**. Las tablas en formato `tibble` tienen **4 ventajas principales**: * Imprime **mayor información de las variables**, y solo **imprime por defecto las primeras filas**. ```r library(tibble) # tibble tabla_tb <- tibble("x" = 1:30, "y" = rep(c("a", "b", "c"), 10), "z" = 31:60, "logica" = rep(c(TRUE, TRUE, FALSE), 10)) tabla_tb ``` ``` > # A tibble: 30 x 4 > x y z logica > <int> <chr> <int> <lgl> > 1 1 a 31 TRUE > 2 2 b 32 TRUE > 3 3 c 33 FALSE > 4 4 a 34 TRUE > 5 5 b 35 TRUE > 6 6 c 36 FALSE > 7 7 a 37 TRUE > 8 8 b 38 TRUE > 9 9 c 39 FALSE > 10 10 a 40 TRUE > # ... with 20 more rows ``` --- # Tibble Los **datos en formato tibble** (del paquete `{tibble}`) son un tipo de `data.frame` mejorado, para una **gestión más ágil, eficiente y coherente**. Las tablas en formato `tibble` tienen **4 ventajas principales**: * Mantiene la **integridad de los datos** (no cambia los tipos de las variables y hace una carga de datos inteligente). ```r tibble("fecha" = as.Date(c("1989-01-01", "1989-02-01", "1989-03-01")), "valores" = 1:3) ``` ``` > # A tibble: 3 x 2 > fecha valores > <date> <int> > 1 1989-01-01 1 > 2 1989-02-01 2 > 3 1989-03-01 3 ``` --- # Tibble Los **datos en formato tibble** (del paquete `{tibble}`) son un tipo de `data.frame` mejorado, para una **gestión más ágil, eficiente y coherente**. Las tablas en formato `tibble` tienen **4 ventajas principales**: * La función `tibble()` **construye las variables secuencialmente**. ```r # data.frame data.frame("x" = 1:3, "z" = 11:13, "x*z" = x * z) ``` ``` > Error in x * z: arreglos de dimensón no compatibles ``` ```r # tibble tabla_tb <- tibble("x" = 1:3, "z" = 11:13, "x*z" = x * z) tabla_tb ``` ``` > # A tibble: 3 x 3 > x z `x*z` > <int> <int> <int> > 1 1 11 11 > 2 2 12 24 > 3 3 13 39 ``` --- # Tibble Puedes consultar **más funcionalidades** de dichos datos en <https://tibble.tidyverse.org/>. Ahora además de poder ver una cabecera de las filas con `head()` tenemos la función `glimpse()`, que nos permite **obtener el resumen de columnas** ```r glimpse(tabla_tb) ``` ``` > Rows: 3 > Columns: 3 > $ x <int> 1, 2, 3 > $ z <int> 11, 12, 13 > $ `x*z` <int> 11, 24, 39 ``` Si ya tienes un `data.frame` es altamente recomendable **convertirlo a tibble** con `as_tibble()`. Prueba además el paquete `{datapasta}`, que nos permite **copiar y pegar tablas de páginas web**, conviertiéndolas en formato `tribble` (tablas `tibble` fila a fila). ```r datos <- tribble( ~colA, ~colB, "a", 1, "b", 2, ) datos ``` ``` > # A tibble: 2 x 2 > colA colB > <chr> <dbl> > 1 a 1 > 2 b 2 ``` --- name: tidy-data class: center, middle # Importando datos ## **Cargando datos** En `{tidyverse}` no solo tenemos distintas funciones para cargar archivos (los paquetes `{readr}`, `{readxl}`, `{haven}`, `{rvest}` o `{googlesheets4}` como ejemplos) sino que disponemos de distintos paquetes para la carga de datos desde API como las del **AEMET, Our World in Data, Eurostat, Twitter o Spotify**. --- # Importando archivos El paquete `{readr}` dentro del entorno `{tidyverse}` contiene distintas funciones útiles para la carga de **datos rectangulares** (sin formatear). * `read_csv()`: archivos `.csv` cuyo separador sea la coma `,` * `read_csv2()`: archivos `.csv` cuyo separado sea la coma `;` * `read_tsv()`: archivos cuyos valores estén separados por un tabulador. * `read_delim()`: función general para leer archivos delimitados por caracteres. Todos necesitan como argumento la **ruta del archivo**, amén de otros argumentos opcionales (saltar o no cabecera, si los decimales son con punto o coma, etc) <div class="figure" style="text-align: center"> <img src="https://github.com/rstudio/cheatsheets/raw/main/pngs/thumbnails/data-import-cheatsheet-thumbs.png" alt="Resumen del paquete `{readr}`. Ver más en https://readr.tidyverse.org/" width="65%" /> <p class="caption">Resumen del paquete `{readr}`. Ver más en https://readr.tidyverse.org/</p> </div> --- # Importando archivos El paquete `{readxl}` contiene distintas funciones útiles para la carga de **archivos formateados por Excel** * `read_excel()`: para archivos Excel en general. * `read_xls()`: para archivos `.xls` en particular * `read_xlsx()`: para archivos `.xlsx` en particular El paquete `{haven}` contiene distintas funciones útiles para la carga de **archivos de SAS, SPSS y Stata**. En todos los paquetes mencionados, podemos ejecutar funciones equivalentes `write_...()` para exportar dichos datos y guardarlos en los formatos mencionados. El paquete `{rvest}` contiene distintas funciones útiles para screpear web sencillas como wikipedia. Puedes ver más información en <https://rvest.tidyverse.org/> --- name: tidy-data class: center, middle # Tidy data ## **Ordenando los datos** > Tidy datasets are all alike, but every messy dataset is messy in its own way (Hadley Wickham, Chief Scientist en RStudio). --- # Tidy data: filosofía de trabajo Hasta ahora solo le hemos dado importancia al «qué» pero no al **«cómo» manejamos los datos**. <div class="figure" style="text-align: center"> <img src="./img/flujo_tidy_data.jpg" alt="Flujo deseable de datos según Hadley Wickham, extraída de https://r4ds.had.co.nz/wrangle-intro.html" width="60%" /> <p class="caption">Flujo deseable de datos según Hadley Wickham, extraída de https://r4ds.had.co.nz/wrangle-intro.html</p> </div> La **organización de nuestros datos** es fundamental para que su preparación y explotación sea lo más eficiente posible. El concepto **tidy data** fue introducido por **Hadley Wickham** como el primer paso a realizar del entorno de paquetes que posteriormente se fueron desarrollando bajo el nombre de `{tidyverse}`, desarrollando un flujo de trabajo desde la carga hasta la visualización --- # Tidy data Los **conjuntos tidy o datos ordenados** tienen tres objetivos principales: 1. **Estandarización** en su estructura. 2. **Sencillez** en su manipulación. 3. Listos para ser **modelizados y visualizados**. Para ello, los **datos ordenados o tidy data** deben cumplir: * Cada **variable en una columna**. * Cada **observación/registro/individuo en una fila** diferente. * Cada **celda con un único valor**. * Cada **conjunto** o unidad observacional conforma una **tabla**. <div class="figure" style="text-align: center"> <img src="./img/tidy_data.png" alt="Infografía con datos ordenados (tidy data) extraída de https://r4ds.had.co.nz/tidy-data.html" width="67%" /> <p class="caption">Infografía con datos ordenados (tidy data) extraída de https://r4ds.had.co.nz/tidy-data.html</p> </div> Lo contrario lo llamaremos **datos desordenados** o _messy data_. --- # Messy data: valores en el nombre Veamos un primer ejemplo con el conjunto `table4a` del paquete `{tidyr}`. ```r library(tidyr) table4a ``` ``` > # A tibble: 3 x 3 > country `1999` `2000` > * <chr> <int> <int> > 1 Afghanistan 745 2666 > 2 Brazil 37737 80488 > 3 China 212258 213766 ``` En este ejemplo de 3 filas y 3 columnas, tenemos dos columnas `1999` y `2000` que **no están representando cada una a una sola variable**: ambas son la misma variable, solo que medida en años distintos, **cada fila está representando dos observaciones (1999, 2000) en lugar de un solo registro**. Lo que haremos será **incluir una nueva columna llamada `year`** que nos marque el año y otra `values` que nos diga el valor de la variable de interés en cada uno de esos años. --- # pivot_longer: pivotando tablas .pull-left[ Con la función `pivot_longer` del mencionado paquete le indicaremos lo siguiente: * `cols`: **nombre de las columnas a pivotar**. * `names_to`: nombre de la columna a la que vamos a mandar los valores que figuran ahora en los nombres de las columnas. * `values_to`: nombre de la columna a la que vamos a mandar los valores. ```r table4a %>% pivot_longer(cols = c("1999", "2000"), names_to = "year", * values_to = "values") ``` ``` > # A tibble: 6 x 3 > country year values > <chr> <chr> <int> > 1 Afghanistan 1999 745 > 2 Afghanistan 2000 2666 > 3 Brazil 1999 37737 > 4 Brazil 2000 80488 > 5 China 1999 212258 > 6 China 2000 213766 ``` ] .pull-right[ <div class="figure" style="text-align: center"> <img src="./img/tidy1.jpg" alt="Imagen extraída de https://github.com/rstudio/cheatsheets/blob/main/tidyr.pdf" width="99%" /> <p class="caption">Imagen extraída de https://github.com/rstudio/cheatsheets/blob/main/tidyr.pdf</p> </div> ] --- # Segundo ejemplo: relig_income ```r relig_income[1:3, ] ``` ``` > # A tibble: 3 x 11 > religion `<$10k` `$10-20k` `$20-30k` `$30-40k` `$40-50k` `$50-75k` `$75-100k` > <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> > 1 Agnostic 27 34 60 81 76 137 122 > 2 Atheist 12 27 37 52 35 70 73 > 3 Buddhist 27 21 30 34 33 58 62 > # ... with 3 more variables: `$100-150k` <dbl>, `>150k` <dbl>, > # `Don't know/refused` <dbl> ``` Salvo la primera, el **resto de columnas tienen como nombre los valores de una variable en sí misma (ingresos)**. Ahora en lugar de indicarle el nombre de todas vamos a indicarle **la columna que NO queremos pivotar**. ```r # No necesitamos comillas en el nombre salvo que tengan caracteres que no sean letras *relig_income %>% pivot_longer(-religion, names_to = "ingresos", values_to = "frec") ``` ``` > # A tibble: 180 x 3 > religion ingresos frec > <chr> <chr> <dbl> > 1 Agnostic <$10k 27 > 2 Agnostic $10-20k 34 > 3 Agnostic $20-30k 60 > 4 Agnostic $30-40k 81 > 5 Agnostic $40-50k 76 > 6 Agnostic $50-75k 137 > 7 Agnostic $75-100k 122 > 8 Agnostic $100-150k 109 > 9 Agnostic >150k 84 > 10 Agnostic Don't know/refused 96 > # ... with 170 more rows ``` --- # Messy data: un registro en varias filas .pull-left[ El **caso inverso**: tener un mismo registro (datos de un mismo individuo) pero dividido en varias filas con `pivot_wider()` **«ampliaremos» la tabla a lo ancho** ```r table2[1:3, ] ``` ``` > # A tibble: 3 x 4 > country year type count > <chr> <int> <chr> <int> > 1 Afghanistan 1999 cases 745 > 2 Afghanistan 1999 population 19987071 > 3 Afghanistan 2000 cases 2666 ``` ```r table2 %>% pivot_wider(names_from = type, * values_from = count) ``` ``` > # A tibble: 6 x 4 > country year cases population > <chr> <int> <int> <int> > 1 Afghanistan 1999 745 19987071 > 2 Afghanistan 2000 2666 20595360 > 3 Brazil 1999 37737 172006362 > 4 Brazil 2000 80488 174504898 > 5 China 1999 212258 1272915272 > 6 China 2000 213766 1280428583 ``` ] .pull-right[ <div class="figure" style="text-align: center"> <img src="./img/tidy2.jpg" alt="Imagen extraída de https://github.com/rstudio/cheatsheets/blob/main/tidyr.pdf" width="99%" /> <p class="caption">Imagen extraída de https://github.com/rstudio/cheatsheets/blob/main/tidyr.pdf</p> </div> ] --- # Messy data: múltiples valores en celda .pull-left[ ```r table3 ``` ``` > # A tibble: 4 x 3 > country year rate > <chr> <int> <chr> > 1 Afghanistan 1999 745/19987071 > 2 Afghanistan 2000 2666/20595360 > 3 Brazil 1999 37737/172006362 > 4 Brazil 2000 80488/174504898 ``` En la columna `rate` hay **guardados dos valores, separados por /**, en una **celda no hay un único valor**. La función `separate()` nos permitirá **separar los múltiples valores** ```r table3 %>% separate(rate, into = c("cases", * "population")) ``` ``` > # A tibble: 6 x 4 > country year cases population > <chr> <int> <chr> <chr> > 1 Afghanistan 1999 745 19987071 > 2 Afghanistan 2000 2666 20595360 > 3 Brazil 1999 37737 172006362 > 4 Brazil 2000 80488 174504898 > 5 China 1999 212258 1272915272 > 6 China 2000 213766 1280428583 ``` ] .pull-right[ <div class="figure" style="text-align: center"> <img src="./img/tidy3.jpg" alt="Imagen extraída de https://github.com/rstudio/cheatsheets/blob/main/tidyr.pdf" width="99%" /> <p class="caption">Imagen extraída de https://github.com/rstudio/cheatsheets/blob/main/tidyr.pdf</p> </div> ] --- # Messy data: múltiples valores en celda Si **queremos un caracter concreto para dividir** podemos indicárselo explícitamente ```r table3 %>% separate(rate, into = c("cases", "population"), sep = "/") ``` ``` > # A tibble: 6 x 4 > country year cases population > <chr> <int> <chr> <chr> > 1 Afghanistan 1999 745 19987071 > 2 Afghanistan 2000 2666 20595360 > 3 Brazil 1999 37737 172006362 > 4 Brazil 2000 80488 174504898 > 5 China 1999 212258 1272915272 > 6 China 2000 213766 1280428583 ``` --- # Messy data: unite() .pull-left[ De la misma manera que **podemos separar columnas también podemos unirlas**. Para ello vamos a usar la tabla `table5`: con la función `unite()` vamos a **unir el siglo (en century) y el año (en year)**. ```r table5 %>% * unite(date, century, year) ``` ``` > # A tibble: 6 x 3 > country date rate > <chr> <chr> <chr> > 1 Afghanistan 19_99 745/19987071 > 2 Afghanistan 20_00 2666/20595360 > 3 Brazil 19_99 37737/172006362 > 4 Brazil 20_00 80488/174504898 > 5 China 19_99 212258/1272915272 > 6 China 20_00 213766/1280428583 ``` ] .pull-right[ <div class="figure" style="text-align: center"> <img src="./img/tidy4.jpg" alt="Imagen extraída de https://github.com/rstudio/cheatsheets/blob/main/tidyr.pdf" width="99%" /> <p class="caption">Imagen extraída de https://github.com/rstudio/cheatsheets/blob/main/tidyr.pdf</p> </div> ] --- # Convertir al tipo adecuado Una opción muy útil que podemos usar al **aplicar la separación de los múltiples valores** es **convertir los datos al tipo adecuado**. Los datos unidos en `rate` eran **caracteres** (no podían ser numéricos). Al separarlos, por defecto, aunque ahora ya son solo números, **los separa como si fueran textos**. Con `convert = TRUE` podemos indicarle que identifique el tipo de dato y lo convierta. ```r table3 %>% separate(rate, into = c("cases", "population"), convert = TRUE) ``` ``` > # A tibble: 6 x 4 > country year cases population > <chr> <int> <int> <int> > 1 Afghanistan 1999 745 19987071 > 2 Afghanistan 2000 2666 20595360 > 3 Brazil 1999 37737 172006362 > 4 Brazil 2000 80488 174504898 > 5 China 1999 212258 1272915272 > 6 China 2000 213766 1280428583 ``` --- # Ejercicios .panelset[ .panel[.panel-name[Ejercicios] * 📝 **Ejercicio 1**: convierte en _tidy data_ el siguiente _data.frame_. ```r tabla_tb <- tibble("trimestre" = c("T1", "T2", "T3"), "2020" = c(10, 12, 7.5), "2021" = c(8, 0, 9)) ``` * 📝 **Ejercicio 2**: convierte en _tidy data_ el siguiente _data.frame_. ```r tabla_tb <- tibble("año" = c(2019, 2019, 2020, 2020, 2021, 2021), "variable" = c("A", "B", "A", "B", "A", "B"), "valor" = c(10, 9383, 7.58, 10839, 9, 32949)) ``` * 📝 **Ejercicio 3**: convierte en _tidydata_ la tabla `table5` del paquete `{tidyr}`. ] .panel[.panel-name[Solución Ej. 1] El problema es que las dos columnas con nombres de año son en realidad valores que deberían pasar a ser variables, así que deberíamos disminuir aplicar `pivot_longer()`. ```r library(tidyr) tabla_tb <- tibble("trimestre" = c("T1", "T2", "T3"), "2020" = c(10, 12, 7.5), "2021" = c(8, 0, 9)) # Aplicamos pivot_longer tabla_tb %>% pivot_longer(cols = c("2020", "2021"), names_to = "año", values_to = "valores") ``` ``` > # A tibble: 6 x 3 > trimestre año valores > <chr> <chr> <dbl> > 1 T1 2020 10 > 2 T1 2021 8 > 3 T2 2020 12 > 4 T2 2021 0 > 5 T3 2020 7.5 > 6 T3 2021 9 ``` ] .panel[.panel-name[Solución Ej. 2] El problema es que las filas que comparten año son el mismo registro (pero con dos características que tenemos divididas en dos filas), así que deberíamos disminuir aplicar `pivot_wider()`. ```r tabla_tb <- tibble("año" = c(2019, 2019, 2020, 2020, 2021, 2021), "variable" = c("A", "B", "A", "B", "A", "B"), "valor" = c(10, 9383, 7.58, 10839, 9, 32949)) # Aplicamos pivot_wider tabla_tb %>% pivot_wider(names_from = "variable", values_from = "valor") ``` ``` > # A tibble: 3 x 3 > año A B > <dbl> <dbl> <dbl> > 1 2019 10 9383 > 2 2020 7.58 10839 > 3 2021 9 32949 ``` ] .panel[.panel-name[Solución Ej. 3] Primero uniremos el siglo y las dos últimas cifras del año para obtener el año completo (guardado en año). Tras ello deberemos separar el valor del ratio en denominador y numerador (ya que ahora hay dos valores en una celda), y convertiremos el tipo de dato en la salida para que sea número. ```r table5 %>% unite(año, century, year, sep = "") %>% separate(rate, c("numerador", "denominador"), convert = TRUE) ``` ``` > # A tibble: 6 x 4 > country año numerador denominador > <chr> <chr> <int> <int> > 1 Afghanistan 1999 745 19987071 > 2 Afghanistan 2000 2666 20595360 > 3 Brazil 1999 37737 172006362 > 4 Brazil 2000 80488 174504898 > 5 China 1999 212258 1272915272 > 6 China 2000 213766 1280428583 ``` ] ] --- name: tidyverse class: center, middle # Introducción a tidyverse ## **Manipulando (para bien) los datos** El conjunto de paquetes de `{tidyverse}` supuso un antes y un después en el preprocesamiento de los datos en `R` --- # Entorno tidyverse Aunque conocemos ya un formato «excelizado» de almacenar los datos, muchas veces los **datos no los tenemos en el formato deseado**, o directamente queremos realizar algunas **transformaciones en los mismos**. Para trabajar con los datos vamos a cargar `{tidyverse}`, un **entorno de paquetes para el manejo de datos**. ```r install.packages("tidyverse") # SOLO la primera vez library(tidyverse) ``` <div class="figure" style="text-align: center"> <img src="https://dadosdelaplace.github.io/courses-ECI-2022/img/flujo_tidyverse.png" alt="Imagen extraída de https://sporella.github.io/datos_espaciales_presentacion/#30" width="60%" /> <p class="caption">Imagen extraída de https://sporella.github.io/datos_espaciales_presentacion/#30</p> </div> --- # Entorno tidyverse El **entorno tidyverse** es una de las **herramientas más importantes en el manejo de datos en R**, una **colección de paquetes** pensada para el manejo, la exploración, el análisis y la visualización de datos, **compartiendo una misma filosofía y gramática**. <div class="figure" style="text-align: center"> <img src="https://dadosdelaplace.github.io/courses-ECI-2022/img/flujo_tidyverse_1.png" alt="Imagen extraída de https://www.storybench.org/getting-started-with-tidyverse-in-r/" width="90%" /> <p class="caption">Imagen extraída de https://www.storybench.org/getting-started-with-tidyverse-in-r/</p> </div> --- # Paquetes de tidyverse * `{tidyr}`: para **adecuar los datos a tidy data** * `{tibble}`: **mejorando los data.frame** para un manejo más eficiente. * Paquetes `{readr}` para una **carga rápida y eficaz de datos rectangulares** (formatos .csv, .tsv, etc). Paquete `{readxl}` para **importar archivos .xls y .xlsx**. Paquete `{haven}` para importar archivos desde **SPSS, Stata y SAS**. Paquete `{httr}` para **importar desde web**. Paquete `{rvest}` para **web scraping**. * `{dplyr}`: una **gramática de manipulación de datos** para facilitar su procesamiento. * `{ggplot2}`: una **gramática para la visualización** de datos. * Paquete `{stringr}` para un manejo sencillo de **cadenas de texto**. Paquete `{forcast}` para un manejo de **variables cualitativas** (en R conocidas como factores). * `{purrr}`: manejo de **listas**. * `{lubridate}` para el **manejo de fechas**. Puedes ver su **documentación completa** en <https://www.tidyverse.org/>. --- # Intro a tidyverse: conjunto starwars Para nuestra **introducción de tidyverse** vamos a trastear con el conjunto de datos `starwars` del paquete `{dplyr}` (incluido en `{tidyverse}`). .pull-left[ <div class="figure" style="text-align: left"> <img src="https://rstudio-education.github.io/bootcamper/slides/02-visualize-data/img/luke-skywalker.png" alt="Imagen extraída de https://rstudio-education.github.io/" width="90%" /> <p class="caption">Imagen extraída de https://rstudio-education.github.io/</p> </div> ] .pull-right[ ```r starwars ``` ``` > # A tibble: 87 x 14 > name height mass hair_color skin_color eye_color birth_year sex gender > <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr> > 1 Luke Sk~ 172 77 blond fair blue 19 male mascu~ > 2 C-3PO 167 75 <NA> gold yellow 112 none mascu~ > 3 R2-D2 96 32 <NA> white, bl~ red 33 none mascu~ > 4 Darth V~ 202 136 none white yellow 41.9 male mascu~ > 5 Leia Or~ 150 49 brown light brown 19 fema~ femin~ > 6 Owen La~ 178 120 brown, gr~ light blue 52 male mascu~ > 7 Beru Wh~ 165 75 brown light blue 47 fema~ femin~ > 8 R5-D4 97 32 <NA> white, red red NA none mascu~ > 9 Biggs D~ 183 84 black light brown 24 male mascu~ > 10 Obi-Wan~ 182 77 auburn, w~ fair blue-gray 57 male mascu~ > # ... with 77 more rows, and 5 more variables: homeworld <chr>, species <chr>, > # films <list>, vehicles <list>, starships <list> ``` ] --- # Intro a tidyverse: conjunto starwars ```r *glimpse(starwars) ``` ``` > Rows: 87 > Columns: 14 > $ name <chr> "Luke Skywalker", "C-3PO", "R2-D2", "Darth Vader", "Leia Or~ > $ height <int> 172, 167, 96, 202, 150, 178, 165, 97, 183, 182, 188, 180, 2~ > $ mass <dbl> 77.0, 75.0, 32.0, 136.0, 49.0, 120.0, 75.0, 32.0, 84.0, 77.~ > $ hair_color <chr> "blond", NA, NA, "none", "brown", "brown, grey", "brown", N~ > $ skin_color <chr> "fair", "gold", "white, blue", "white", "light", "light", "~ > $ eye_color <chr> "blue", "yellow", "red", "yellow", "brown", "blue", "blue",~ > $ birth_year <dbl> 19.0, 112.0, 33.0, 41.9, 19.0, 52.0, 47.0, NA, 24.0, 57.0, ~ > $ sex <chr> "male", "none", "none", "male", "female", "male", "female",~ > $ gender <chr> "masculine", "masculine", "masculine", "masculine", "femini~ > $ homeworld <chr> "Tatooine", "Tatooine", "Naboo", "Tatooine", "Alderaan", "T~ > $ species <chr> "Human", "Droid", "Droid", "Human", "Human", "Human", "Huma~ > $ films <list> <"The Empire Strikes Back", "Revenge of the Sith", "Return~ > $ vehicles <list> <"Snowspeeder", "Imperial Speeder Bike">, <>, <>, <>, "Imp~ > $ starships <list> <"X-wing", "Imperial shuttle">, <>, <>, "TIE Advanced x1",~ ``` Dicho conjunto de datos, extraído de la [Star Wars API](https://swapi.dev/), recopila **14 variables para 87 personajes de Star Wars**. ```r dim(starwars) ``` ``` > [1] 87 14 ``` --- # Intro a tidyverse: conjunto starwars ```r print(starwars, n = 3, width = Inf) ``` ``` > # A tibble: 87 x 14 > name height mass hair_color skin_color eye_color birth_year sex > <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> > 1 Luke Skywalker 172 77 blond fair blue 19 male > 2 C-3PO 167 75 <NA> gold yellow 112 none > 3 R2-D2 96 32 <NA> white, blue red 33 none > gender homeworld species films vehicles starships > <chr> <chr> <chr> <list> <list> <list> > 1 masculine Tatooine Human <chr [5]> <chr [2]> <chr [2]> > 2 masculine Tatooine Droid <chr [6]> <chr [0]> <chr [0]> > 3 masculine Naboo Droid <chr [7]> <chr [0]> <chr [0]> > # ... with 84 more rows ``` * `int`: números enteros (`height`). * `dbl`: números reales (`mass`, `birth_year`). * `chr`: cadenas de texto (`name`, `hair_color`, `skin_color`, `eye_color`, `sex`, `gender`, `homeworld`, `species`). * `list`: listas (`films`, `vehicles`, `starships`). --- # Intro a tidyverse: conjunto starwars Los **datos de tipo lista** (aparecen con un icono distinto en el menú de variables) son el dato más flexible de `R`: permiten **concatenar datos de cualquier tipo PERO también de cualquier longitud**. Por ejemplo en `starwars` tenemos guardados las películas de cada personaje en modo lista en la variable `starwars$films`. Por ejemplo, podemos extraer las películas en las que aparecen los tres primeros personajes, basta con ejecutar (del conjunto starwars –> accedemos a la variable films con `$`) ```r starwars$films[1:2] ``` ``` > [[1]] > [1] "The Empire Strikes Back" "Revenge of the Sith" > [3] "Return of the Jedi" "A New Hope" > [5] "The Force Awakens" > > [[2]] > [1] "The Empire Strikes Back" "Attack of the Clones" > [3] "The Phantom Menace" "Revenge of the Sith" > [5] "Return of the Jedi" "A New Hope" ``` El paquete `{purrr}` contienen multitud de herramientas para **aplicar de forma sencilla funciones a cada elemento de una lista**. --- # dplyr: manipulando datos <div class="figure" style="text-align: center"> <img src="https://dadosdelaplace.github.io/courses-ECI-2022/img/dplyr.png" alt="Cheet sheet de las opciones del paquete dplyr para la manipulación de datos extraída de https://github.com/rstudio/cheatsheets/blob/master/data-transformation.pdf" width="63%" /> <p class="caption">Cheet sheet de las opciones del paquete dplyr para la manipulación de datos extraída de https://github.com/rstudio/cheatsheets/blob/master/data-transformation.pdf</p> </div> --- # Pipeline (tuberías) %>% En este entorno de trabajo tendremos un **operador clave**: el **operador pipeline (%>%)**, el cual lo debemos interpretar como una **tubería conectada a los datos por el que pasan operaciones**, de una forma legible. Con el **pipeline %>%** podremos escribir (y leer) la concetanción de órdenes de forma sencilla y **léxicamente interpertable** ```r # Idea datos %>% filtro(...) %>% ordeno(...) %>% selecciono(...) %>% modifico(...) %>% agrupo(...) %>% resumo(...) %>% visualizo(...) ``` Dicho operador depende del paquete `{magrittr}`: para evitar esta dependencia (cuantos menos paquetes, mejor), desde la **versión 4.1.0 de R** disponemos de un operador nativo de R, el **operador |>** (disponible además fuera de tidyverse). --- # Visualizar el flujo de datos Existe una **reciente herramienta** que nos va a permitir **entender mejor y visualizar el flujo de trabajo** en `{tidyverse}`: <https://tidydatatutor.com/>. Basta con poner el código que queremos ejecutar, y nos **muestra visualmente las operaciones en los datos**. <img src = "https://dadosdelaplace.github.io/courses-ECI-2022/img/tidytutor3.jpg" alt = "course-ECI" align = "right" width = "450" style = "margin-top: 1vh;margin-right: 0.5rem;margin-left: 0.5rem;"> <img src = "https://dadosdelaplace.github.io/courses-ECI-2022/img/tidytutor4.jpg" alt = "course-ECI" align = "right" width = "450" style = "margin-top: 1vh;margin-right: 0.5rem;margin-left: 0.5rem;"> <img src = "https://dadosdelaplace.github.io/courses-ECI-2022/img/tidytutor5.jpg" alt = "course-ECI" align = "right" width = "400" style = "margin-top: 1vh;margin-right: 0.5rem;margin-left: 0.5rem;"> --- # Filtrar filas: filter() .pull-left[ Una de las **operaciones más comunes** es **filtrar registros en base a alguna condición**: con `filter()` se seleccionarán solo individuos que cumplan ciertas condiciones. ```r # Idea starwars %>% filtro(cond1, cond2, ...) ``` Por ejemplo, vamos a **filtrar aquellos personajes con ojos marrones** (cumpliendo la condición `eye_color == "brown"`) ```r starwars %>% * filter(eye_color == "brown") ``` ``` > # A tibble: 21 x 14 > name height mass hair_color skin_color eye_color birth_year sex gender > <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr> > 1 Leia Or~ 150 49 brown light brown 19 fema~ femin~ > 2 Biggs D~ 183 84 black light brown 24 male mascu~ > 3 Han Solo 180 80 brown fair brown 29 male mascu~ > 4 Yoda 66 17 white green brown 896 male mascu~ > 5 Boba Fe~ 183 78.2 black fair brown 31.5 male mascu~ > 6 Lando C~ 177 79 black dark brown 31 male mascu~ > 7 Arvel C~ NA NA brown fair brown NA male mascu~ > 8 Wicket ~ 88 20 brown brown brown 8 male mascu~ > 9 Quarsh ~ 183 NA black dark brown 62 <NA> <NA> > 10 Shmi Sk~ 163 NA black fair brown 72 fema~ femin~ > # ... with 11 more rows, and 5 more variables: homeworld <chr>, species <chr>, > # films <list>, vehicles <list>, starships <list> ``` ] .pull-right[ <div class="figure" style="text-align: center"> <img src="./img/filter1.jpg" alt="Flujo de https://tidydatatutor.com/" width="150%" /> <p class="caption">Flujo de https://tidydatatutor.com/</p> </div> ] --- # Filtrar filas: filter() .pull-left[ El operador de comparación `==` puede ser cambiado por otros: * `!=`: parte de la izq distinta la dcha. * `<`, `>`: menor, mayor que... * `<=`, `>=`: menor o igual, mayor igual que... * `%in%`: si los valores pertenecen a una lista finita de opciones permitidas. * `between(variable, val1, val2)`: si los valores (normalmente continuos) son mayores que `val1` y menores que `val2` ```r starwars %>% filter(eye_color != "brown") ``` ``` > # A tibble: 66 x 14 > name height mass hair_color skin_color eye_color birth_year sex gender > <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr> > 1 Luke Sk~ 172 77 blond fair blue 19 male mascu~ > 2 C-3PO 167 75 <NA> gold yellow 112 none mascu~ > 3 R2-D2 96 32 <NA> white, bl~ red 33 none mascu~ > 4 Darth V~ 202 136 none white yellow 41.9 male mascu~ > 5 Owen La~ 178 120 brown, gr~ light blue 52 male mascu~ > 6 Beru Wh~ 165 75 brown light blue 47 fema~ femin~ > 7 R5-D4 97 32 <NA> white, red red NA none mascu~ > 8 Obi-Wan~ 182 77 auburn, w~ fair blue-gray 57 male mascu~ > 9 Anakin ~ 188 84 blond fair blue 41.9 male mascu~ > 10 Wilhuff~ 180 NA auburn, g~ fair blue 64 male mascu~ > # ... with 56 more rows, and 5 more variables: homeworld <chr>, species <chr>, > # films <list>, vehicles <list>, starships <list> ``` ] .pull-right[ <div class="figure" style="text-align: center"> <img src="./img/filter2.jpg" alt="Flujo de https://tidydatatutor.com/" width="150%" /> <p class="caption">Flujo de https://tidydatatutor.com/</p> </div> ] --- # Filtrar filas: filter() .pull-left[ ```r # con ojos marrones o azules o rojos starwars %>% filter(eye_color %in% c("brown", "blue")) ``` ``` > # A tibble: 5 x 4 > name height mass eye_color > <chr> <int> <dbl> <chr> > 1 Luke Skywalker 172 77 blue > 2 Leia Organa 150 49 brown > 3 Owen Lars 178 120 blue > 4 Beru Whitesun lars 165 75 blue > 5 Biggs Darklighter 183 84 brown ``` ```r # con estatura entre 120 y 160 cm starwars %>% filter(between(height, 120, 160)) ``` ``` > # A tibble: 5 x 4 > name height mass eye_color > <chr> <int> <dbl> <chr> > 1 Leia Organa 150 49 brown > 2 Mon Mothma 150 NA blue > 3 Nien Nunb 160 68 black > 4 Watto 137 NA yellow > 5 Gasgano 122 NA black ``` ] .pull-right[ <div class="figure" style="text-align: center"> <img src="./img/filter3.jpg" alt="Flujo de https://tidydatatutor.com/" width="150%" /> <p class="caption">Flujo de https://tidydatatutor.com/</p> </div> ] --- # Filtrar filas: filter() .pull-left[ Las condiciones se pueden **concatenar**, pudiendo en pocas líneas realizar un filtro complejo * `a & b`: solo devolverá `TRUE` cuando se cumpla tanto `a` como `b`. * `a | b`: devolverá `TRUE` cuando se cumpla al menos una de ellas (a y no b, b y no a, o ambas). Por ejemplo, seleccionamos los **personajes con ojos marrones Y ADEMÁS NO humanos**, o los personajes con más de 60 años. ```r starwars %>% filter((eye_color == "brown" & species != "Human") | birth_year > 60) ``` ``` > # A tibble: 18 x 14 > name height mass hair_color skin_color eye_color birth_year sex gender > <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr> > 1 C-3PO 167 75 <NA> gold yellow 112 none mascu~ > 2 Wilhuff~ 180 NA auburn, g~ fair blue 64 male mascu~ > 3 Chewbac~ 228 112 brown unknown blue 200 male mascu~ > 4 Jabba D~ 175 1358 <NA> green-tan~ orange 600 herm~ mascu~ > 5 Yoda 66 17 white green brown 896 male mascu~ > 6 Palpati~ 170 75 grey pale yellow 82 male mascu~ > 7 Wicket ~ 88 20 brown brown brown 8 male mascu~ > 8 Qui-Gon~ 193 89 brown fair blue 92 male mascu~ > 9 Finis V~ 170 NA blond fair blue 91 male mascu~ > 10 Quarsh ~ 183 NA black dark brown 62 <NA> <NA> > 11 Shmi Sk~ 163 NA black fair brown 72 fema~ femin~ > 12 Mace Wi~ 188 84 none dark brown 72 male mascu~ > 13 Ki-Adi-~ 198 82 white pale yellow 92 male mascu~ > 14 Eeth Ko~ 171 NA black brown brown NA male mascu~ > 15 Cliegg ~ 183 NA brown fair blue 82 male mascu~ > 16 Dooku 193 80 white fair brown 102 male mascu~ > 17 Bail Pr~ 191 NA black tan brown 67 male mascu~ > 18 Jango F~ 183 79 black tan brown 66 male mascu~ > # ... with 5 more variables: homeworld <chr>, species <chr>, films <list>, > # vehicles <list>, starships <list> ``` ] .pull-right[ <div class="figure" style="text-align: center"> <img src="./img/filter5.jpg" alt="Flujo de https://tidydatatutor.com/" width="150%" /> <p class="caption">Flujo de https://tidydatatutor.com/</p> </div> ] --- # Ejercicios con filter() .panelset[ .panel[.panel-name[Ejercicios] * 📝 **Ejercicio 1**: selecciona del conjunto de `starwars` solo los personajes que sean humanos (`species == "Human"`) * 📝 **Ejercicio 2**: selecciona del conjunto de `starwars` solo los personajes cuyo peso esté entre 65 y 90 kg. * 📝 **Ejercicio 3**: selecciona del conjunto de `starwars` los personajes con ojos marrones o rojos. * 📝 **Ejercicio 4**: selecciona del conjunto de `starwars` los personajes no humanos, hombres y que midan más de 170 cm, o los personajes con ojos marrones o rojos. ] .panel[.panel-name[Sol. Ej. 1] ```r starwars %>% filter(species == "Human") ``` ``` > # A tibble: 35 x 14 > name height mass hair_color skin_color eye_color birth_year sex gender > <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr> > 1 Luke Sk~ 172 77 blond fair blue 19 male mascu~ > 2 Darth V~ 202 136 none white yellow 41.9 male mascu~ > 3 Leia Or~ 150 49 brown light brown 19 fema~ femin~ > 4 Owen La~ 178 120 brown, gr~ light blue 52 male mascu~ > 5 Beru Wh~ 165 75 brown light blue 47 fema~ femin~ > 6 Biggs D~ 183 84 black light brown 24 male mascu~ > 7 Obi-Wan~ 182 77 auburn, w~ fair blue-gray 57 male mascu~ > 8 Anakin ~ 188 84 blond fair blue 41.9 male mascu~ > 9 Wilhuff~ 180 NA auburn, g~ fair blue 64 male mascu~ > 10 Han Solo 180 80 brown fair brown 29 male mascu~ > # ... with 25 more rows, and 5 more variables: homeworld <chr>, species <chr>, > # films <list>, vehicles <list>, starships <list> ``` ] .panel[.panel-name[Sol. Ej. 2] ```r starwars %>% filter(between(mass, 65, 90)) ``` ``` > # A tibble: 32 x 14 > name height mass hair_color skin_color eye_color birth_year sex gender > <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr> > 1 Luke Sk~ 172 77 blond fair blue 19 male mascu~ > 2 C-3PO 167 75 <NA> gold yellow 112 none mascu~ > 3 Beru Wh~ 165 75 brown light blue 47 fema~ femin~ > 4 Biggs D~ 183 84 black light brown 24 male mascu~ > 5 Obi-Wan~ 182 77 auburn, w~ fair blue-gray 57 male mascu~ > 6 Anakin ~ 188 84 blond fair blue 41.9 male mascu~ > 7 Han Solo 180 80 brown fair brown 29 male mascu~ > 8 Greedo 173 74 <NA> green black 44 male mascu~ > 9 Wedge A~ 170 77 brown fair hazel 21 male mascu~ > 10 Palpati~ 170 75 grey pale yellow 82 male mascu~ > # ... with 22 more rows, and 5 more variables: homeworld <chr>, species <chr>, > # films <list>, vehicles <list>, starships <list> ``` ] .panel[.panel-name[Sol. Ej. 3] ```r starwars %>% filter(eye_color %in% c("brown", "red")) ``` ``` > # A tibble: 26 x 14 > name height mass hair_color skin_color eye_color birth_year sex gender > <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr> > 1 R2-D2 96 32 <NA> white, bl~ red 33 none mascu~ > 2 Leia Or~ 150 49 brown light brown 19 fema~ femin~ > 3 R5-D4 97 32 <NA> white, red red NA none mascu~ > 4 Biggs D~ 183 84 black light brown 24 male mascu~ > 5 Han Solo 180 80 brown fair brown 29 male mascu~ > 6 Yoda 66 17 white green brown 896 male mascu~ > 7 Boba Fe~ 183 78.2 black fair brown 31.5 male mascu~ > 8 IG-88 200 140 none metal red 15 none mascu~ > 9 Bossk 190 113 none green red 53 male mascu~ > 10 Lando C~ 177 79 black dark brown 31 male mascu~ > # ... with 16 more rows, and 5 more variables: homeworld <chr>, species <chr>, > # films <list>, vehicles <list>, starships <list> ``` ] .panel[.panel-name[Sol. Ej. 4] ```r starwars %>% filter((species != "Human" & sex == "Male" & height > 170) | eye_color %in% c("brown", "red")) ``` ``` > # A tibble: 26 x 14 > name height mass hair_color skin_color eye_color birth_year sex gender > <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr> > 1 R2-D2 96 32 <NA> white, bl~ red 33 none mascu~ > 2 Leia Or~ 150 49 brown light brown 19 fema~ femin~ > 3 R5-D4 97 32 <NA> white, red red NA none mascu~ > 4 Biggs D~ 183 84 black light brown 24 male mascu~ > 5 Han Solo 180 80 brown fair brown 29 male mascu~ > 6 Yoda 66 17 white green brown 896 male mascu~ > 7 Boba Fe~ 183 78.2 black fair brown 31.5 male mascu~ > 8 IG-88 200 140 none metal red 15 none mascu~ > 9 Bossk 190 113 none green red 53 male mascu~ > 10 Lando C~ 177 79 black dark brown 31 male mascu~ > # ... with 16 more rows, and 5 more variables: homeworld <chr>, species <chr>, > # films <list>, vehicles <list>, starships <list> ``` ] ] --- # Rebanadas de los datos: slice() También podemos **seleccionar filas por su posición** con `slice()`. ```r # slice: extraemos filas por índice de fila. *starwars %>% slice(1) ``` ``` > # A tibble: 1 x 14 > name height mass hair_color skin_color eye_color birth_year sex gender > <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr> > 1 Luke Sky~ 172 77 blond fair blue 19 male mascu~ > # ... with 5 more variables: homeworld <chr>, species <chr>, films <list>, > # vehicles <list>, starships <list> ``` Podemos **extraer varias a la vez**, incluso **usar una secuencia de índices a extraer**, por ejemplo solo las filas pares. ```r # filas pares hasta la octava starwars %>% slice(seq(2, 8, by = 2)) ``` ``` > # A tibble: 4 x 14 > name height mass hair_color skin_color eye_color birth_year sex gender > <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr> > 1 C-3PO 167 75 <NA> gold yellow 112 none mascu~ > 2 Darth Va~ 202 136 none white yellow 41.9 male mascu~ > 3 Owen Lars 178 120 brown, gr~ light blue 52 male mascu~ > 4 R5-D4 97 32 <NA> white, red red NA none mascu~ > # ... with 5 more variables: homeworld <chr>, species <chr>, films <list>, > # vehicles <list>, starships <list> ``` --- # Rebanadas de los datos: slice() .pull-left[ Disponemos además de opciones por defecto * `slice_head(n = ...)`: extraer las n primeras filas. * `slice_tail(n = ...)`: extraer las n últimas filas. * `slice_sample(n = ...)`: extrae n filas elegidas aleatoriamente. * `slice_min(var, n = ...)` y `slice_max(var, n = ...)`: **extrae las n filas con menor/mayor de una variable** (si hay empate, mostrará todas salvo que `with_ties = FALSE`). ```r # los 3 más bajitos starwars %>% slice_min(height, n = 3) ``` ``` > # A tibble: 3 x 14 > name height mass hair_color skin_color eye_color birth_year sex gender > <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr> > 1 Yoda 66 17 white green brown 896 male mascu~ > 2 Ratts Ty~ 79 15 none grey, blue unknown NA male mascu~ > 3 Wicket S~ 88 20 brown brown brown 8 male mascu~ > # ... with 5 more variables: homeworld <chr>, species <chr>, films <list>, > # vehicles <list>, starships <list> ``` ] .pull-right[ <div class="figure" style="text-align: center"> <img src="./img/filter6.jpg" alt="Flujo de https://tidydatatutor.com/" width="150%" /> <p class="caption">Flujo de https://tidydatatutor.com/</p> </div> ] --- # Reordenar filas: rearrange() Otra operación habitual en las filas es **ordenar las filas en función del valor de alguna de las variables** con `arrange()`, pasándole como argumento el nombre de la variable que usaremos para la ordenación. Por defecto la ordenación es de menor a mayor pero podemos invertirlo usando `desc()`. ```r datos %>% filtro(cond1, cond2) %>% * ordeno(var1, desc(var2)) ``` Por ejemplo, vamos a **ordenar** los personajes por altura, de bajitos a altos, y en caso de empate, por peso (pero al revés, de pesados a ligeros). ```r starwars %>% * arrange(height, desc(mass)) ``` ``` > # A tibble: 87 x 14 > name height mass hair_color skin_color eye_color birth_year sex gender > <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr> > 1 Yoda 66 17 white green brown 896 male mascu~ > 2 Ratts T~ 79 15 none grey, blue unknown NA male mascu~ > 3 Wicket ~ 88 20 brown brown brown 8 male mascu~ > 4 Dud Bolt 94 45 none blue, grey yellow NA male mascu~ > 5 R2-D2 96 32 <NA> white, bl~ red 33 none mascu~ > 6 R4-P17 96 NA none silver, r~ red, blue NA none femin~ > 7 R5-D4 97 32 <NA> white, red red NA none mascu~ > 8 Sebulba 112 40 none grey, red orange NA male mascu~ > 9 Gasgano 122 NA none white, bl~ black NA male mascu~ > 10 Watto 137 NA black blue, grey yellow NA male mascu~ > # ... with 77 more rows, and 5 more variables: homeworld <chr>, species <chr>, > # films <list>, vehicles <list>, starships <list> ``` --- # Eliminar filas La misma lógica que hemos usado para seleccionar filas podemos usarla para **eliminar filas**: * `filter(!condicion)`: filtramos aquellas filas que NO CUMPLEN la condición (eliminando las que sí). * `slice(-indices)`: eliminamos las filas que ocupan los indices. Por ejemplo, vamos a eliminar las 80 primeras filas. ```r # eliminamos las 80 primeras filas starwars %>% slice(-(1:80)) ``` ``` > # A tibble: 7 x 14 > name height mass hair_color skin_color eye_color birth_year sex gender > <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr> > 1 Tion Med~ 206 80 none grey black NA male mascu~ > 2 Finn NA NA black dark dark NA male mascu~ > 3 Rey NA NA brown light hazel NA fema~ femin~ > 4 Poe Dame~ NA NA brown light brown NA male mascu~ > 5 BB8 NA NA none none black NA none mascu~ > 6 Captain ~ NA NA unknown unknown unknown NA <NA> <NA> > 7 Padmé Am~ 165 45 brown light brown 46 fema~ femin~ > # ... with 5 more variables: homeworld <chr>, species <chr>, films <list>, > # vehicles <list>, starships <list> ``` --- # Eliminar datos ausentes (NA) Podemos también **eliminar los registros ausentes** en alguna de sus variables con `drop_na()`. Si no especificamos variables, elimina todos los registros que tenga alguno de sus campos ausente. ```r starwars %>% drop_na() ``` ``` > # A tibble: 29 x 14 > name height mass hair_color skin_color eye_color birth_year sex gender > <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr> > 1 Luke Sk~ 172 77 blond fair blue 19 male mascu~ > 2 Darth V~ 202 136 none white yellow 41.9 male mascu~ > 3 Leia Or~ 150 49 brown light brown 19 fema~ femin~ > 4 Owen La~ 178 120 brown, gr~ light blue 52 male mascu~ > 5 Beru Wh~ 165 75 brown light blue 47 fema~ femin~ > 6 Biggs D~ 183 84 black light brown 24 male mascu~ > 7 Obi-Wan~ 182 77 auburn, w~ fair blue-gray 57 male mascu~ > 8 Anakin ~ 188 84 blond fair blue 41.9 male mascu~ > 9 Chewbac~ 228 112 brown unknown blue 200 male mascu~ > 10 Han Solo 180 80 brown fair brown 29 male mascu~ > # ... with 19 more rows, and 5 more variables: homeworld <chr>, species <chr>, > # films <list>, vehicles <list>, starships <list> ``` Podemos indicarle que nos elimine filas con datos ausentes fijándonos solo en **algunos campos en concreto**. ```r starwars %>% drop_na(mass, height, sex, gender, birth_year) ``` --- # Eliminar duplicados: distinct() Otra opción es **eliminar filas duplicadas** con `distinct()`, pasándole como argumentos el nombre de las variables que usaremos para quitar duplicados, por ejemplo, personajes con igual par de color de pelo y ojos. Por defecto, solo extrae las columnas en base a las cuales hemos eliminado duplicados. Si queremos que nos mantenga todas deberemos explicitarlo con `.keep_all = TRUE`. ```r # Elimina filas con igual (color_pelo, color_ojos) starwars %>% distinct(hair_color, eye_color) ``` ``` > # A tibble: 35 x 2 > hair_color eye_color > <chr> <chr> > 1 blond blue > 2 <NA> yellow > 3 <NA> red > 4 none yellow > 5 brown brown > 6 brown, grey blue > 7 brown blue > 8 black brown > 9 auburn, white blue-gray > 10 auburn, grey blue > # ... with 25 more rows ``` --- # Añadir filas: bind_rows() Si quisiéramos añadir un nuevo registro manualmente, podremos hacerlo con `bind_rows()`, asegurándonos que las **variables en el nuevo registro son las mismas que en el original**. Por ejemplo, vamos a añadir al fichero original los 3 primeros registros (al final). ```r starwars_nuevo <- * bind_rows(starwars, starwars[1:3, ]) dim(starwars) ``` ``` > [1] 87 14 ``` ```r dim(starwars_nuevo) ``` ``` > [1] 90 14 ``` --- # Ejercicios .panelset[ .panel[.panel-name[Ejercicios] * 📝 **Ejercicio 1**: selecciona solo los personajes que sean humanos y de ojos marrones, para después ordernarlos en altura descendente y peso ascendente. * 📝 **Ejercicio 2**: extrae 3 registros aleatoriamente. Vuelve a hacerlo para comprobar que salen diferentes. * 📝 **Ejercicio 3**: extrae el 10% de los registros aleatoriamente, llamando a `slice_sample()` asignando valor al argumento `prop` * 📝 **Ejercicio 4**: selecciona los 3 personajes más mayores y los 3 personajes más bajitos. * 📝 **Ejercicio 5**: para saber que valores únicos hay en el color de pelo, elimina duplicados por dicha variable `hair_color`, eliminando antes los ausentes de dicha variable ] .panel[.panel-name[Sol. Ej. 1] ```r # Podemos combinar varias acciones en pocas líneas starwars %>% filter(eye_color == "brown", species == "Human") %>% arrange(height, desc(mass)) ``` ``` > # A tibble: 17 x 14 > name height mass hair_color skin_color eye_color birth_year sex gender > <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr> > 1 Leia Or~ 150 49 brown light brown 19 fema~ femin~ > 2 Cordé 157 NA brown light brown NA fema~ femin~ > 3 Shmi Sk~ 163 NA black fair brown 72 fema~ femin~ > 4 Padmé A~ 165 45 brown light brown 46 fema~ femin~ > 5 Dormé 165 NA brown light brown NA fema~ femin~ > 6 Lando C~ 177 79 black dark brown 31 male mascu~ > 7 Han Solo 180 80 brown fair brown 29 male mascu~ > 8 Biggs D~ 183 84 black light brown 24 male mascu~ > 9 Jango F~ 183 79 black tan brown 66 male mascu~ > 10 Boba Fe~ 183 78.2 black fair brown 31.5 male mascu~ > 11 Gregar ~ 185 85 black dark brown NA male mascu~ > 12 Mace Wi~ 188 84 none dark brown 72 male mascu~ > 13 Raymus ~ 188 79 brown light brown NA male mascu~ > 14 Bail Pr~ 191 NA black tan brown 67 male mascu~ > 15 Dooku 193 80 white fair brown 102 male mascu~ > 16 Arvel C~ NA NA brown fair brown NA male mascu~ > 17 Poe Dam~ NA NA brown light brown NA male mascu~ > # ... with 5 more variables: homeworld <chr>, species <chr>, films <list>, > # vehicles <list>, starships <list> ``` ] .panel[.panel-name[Sol. Ej. 2] ```r starwars %>% slice_sample(n = 3) ``` ``` > # A tibble: 3 x 14 > name height mass hair_color skin_color eye_color birth_year sex gender > <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr> > 1 Darth Ma~ 175 80 none red yellow 54 male mascu~ > 2 Bib Fort~ 180 NA none pale pink NA male mascu~ > 3 Shmi Sky~ 163 NA black fair brown 72 fema~ femin~ > # ... with 5 more variables: homeworld <chr>, species <chr>, films <list>, > # vehicles <list>, starships <list> ``` ```r starwars %>% slice_sample(n = 3) ``` ``` > # A tibble: 3 x 14 > name height mass hair_color skin_color eye_color birth_year sex gender > <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr> > 1 Zam Wese~ 168 55 blonde fair, gre~ yellow NA fema~ femin~ > 2 Sebulba 112 40 none grey, red orange NA male mascu~ > 3 Jango Fe~ 183 79 black tan brown 66 male mascu~ > # ... with 5 more variables: homeworld <chr>, species <chr>, films <list>, > # vehicles <list>, starships <list> ``` ] .panel[.panel-name[Sol. Ej. 3] ```r starwars %>% slice_sample(prop = 0.1) ``` ``` > # A tibble: 8 x 14 > name height mass hair_color skin_color eye_color birth_year sex gender > <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr> > 1 Han Solo 180 80 brown fair brown 29 male mascu~ > 2 C-3PO 167 75 <NA> gold yellow 112 none mascu~ > 3 Obi-Wan ~ 182 77 auburn, w~ fair blue-gray 57 male mascu~ > 4 Mace Win~ 188 84 none dark brown 72 male mascu~ > 5 Luminara~ 170 56.2 black yellow blue 58 fema~ femin~ > 6 BB8 NA NA none none black NA none mascu~ > 7 Lama Su 229 88 none grey black NA male mascu~ > 8 Owen Lars 178 120 brown, gr~ light blue 52 male mascu~ > # ... with 5 more variables: homeworld <chr>, species <chr>, films <list>, > # vehicles <list>, starships <list> ``` ] .panel[.panel-name[Sol. Ej. 4] ```r starwars %>% slice_max(birth_year, n = 3) ``` ``` > # A tibble: 3 x 14 > name height mass hair_color skin_color eye_color birth_year sex gender > <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr> > 1 Yoda 66 17 white green brown 896 male mascu~ > 2 Jabba De~ 175 1358 <NA> green-tan~ orange 600 herm~ mascu~ > 3 Chewbacca 228 112 brown unknown blue 200 male mascu~ > # ... with 5 more variables: homeworld <chr>, species <chr>, films <list>, > # vehicles <list>, starships <list> ``` ```r starwars %>% slice_min(height, n = 3) ``` ``` > # A tibble: 3 x 14 > name height mass hair_color skin_color eye_color birth_year sex gender > <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr> > 1 Yoda 66 17 white green brown 896 male mascu~ > 2 Ratts Ty~ 79 15 none grey, blue unknown NA male mascu~ > 3 Wicket S~ 88 20 brown brown brown 8 male mascu~ > # ... with 5 more variables: homeworld <chr>, species <chr>, films <list>, > # vehicles <list>, starships <list> ``` ] .panel[.panel-name[Sol. Ej. 5] ```r starwars %>% drop_na(hair_color) %>% distinct(hair_color) ``` ``` > # A tibble: 12 x 1 > hair_color > <chr> > 1 blond > 2 none > 3 brown > 4 brown, grey > 5 black > 6 auburn, white > 7 auburn, grey > 8 white > 9 grey > 10 auburn > 11 blonde > 12 unknown ``` ] ] --- # Seleccionar columnas: select() .pull-left[ La opción más sencilla para **seleccionar variables** es `select()`, con argumentos los nombres de columnas (¡SIN COMILLAS!). ```r # Idea datos %>% filtro(cond1, cond2) %>% ordeno(var1, desc(var2)) %>% selecciono(col1, col2, ...) ``` Seleccionamos por ejemplo la columna `hair_color` ```r # seleccionamos 1 columna starwars %>% * select(hair_color) ``` ``` > # A tibble: 87 x 1 > hair_color > <chr> > 1 blond > 2 <NA> > 3 <NA> > 4 none > 5 brown > 6 brown, grey > 7 brown > 8 <NA> > 9 black > 10 auburn, white > # ... with 77 more rows ``` ] .pull-right[ <div class="figure" style="text-align: center"> <img src="./img/select1.jpg" alt="Flujo de https://tidydatatutor.com/" width="150%" /> <p class="caption">Flujo de https://tidydatatutor.com/</p> </div> ] --- # Seleccionar columnas: select() .pull-left[ Como sucedía al filtrar, la función `select()` es bastante versatil y nos permite: * Seleccionar **varias variables a la vez** (concatenando sus nombres) * **Deseleccionar** columnas con `-` * Seleccionar columnas que **comiencen por un prefijo** (`starts_with()`), **terminen** con un sufijo (`ends_with()`), **contengan** un texto (`contains()`) o cumplan una **expresión regular** (`matches()`) * Seleccionar columnas de **un tipo** haciendo uso de `where(is.numeric)`, etc. ```r # color de piel y de pelo (en ese orden) starwars %>% select(c(skin_color, hair_color)) ``` ``` > # A tibble: 87 x 2 > skin_color hair_color > <chr> <chr> > 1 fair blond > 2 gold <NA> > 3 white, blue <NA> > 4 white none > 5 light brown > 6 light brown, grey > 7 light brown > 8 white, red <NA> > 9 light black > 10 fair auburn, white > # ... with 77 more rows ``` ] .pull-right[ <div class="figure" style="text-align: center"> <img src="./img/select2.jpg" alt="Flujo de https://tidydatatutor.com/" width="150%" /> <p class="caption">Flujo de https://tidydatatutor.com/</p> </div> ] --- # Seleccionar columnas: select() .pull-left[ ```r # desde name hasta hair_color starwars %>% select(c(name:hair_color)) ``` ``` > # A tibble: 5 x 4 > name height mass hair_color > <chr> <int> <dbl> <chr> > 1 Luke Skywalker 172 77 blond > 2 C-3PO 167 75 <NA> > 3 R2-D2 96 32 <NA> > 4 Darth Vader 202 136 none > 5 Leia Organa 150 49 brown ``` ```r # nombre acaba en "color" starwars %>% select(ends_with("color")) ``` ``` > # A tibble: 5 x 3 > hair_color skin_color eye_color > <chr> <chr> <chr> > 1 blond fair blue > 2 <NA> gold yellow > 3 <NA> white, blue red > 4 none white yellow > 5 brown light brown ``` ] .pull-right[ ```r # empiezan por h starwars %>% select(starts_with("h")) ``` ``` > # A tibble: 5 x 3 > height hair_color homeworld > <int> <chr> <chr> > 1 172 blond Tatooine > 2 167 <NA> Tatooine > 3 96 <NA> Naboo > 4 202 none Tatooine > 5 150 brown Alderaan ``` ```r # Solo columnas numéricas starwars %>% select(where(is.numeric)) ``` ``` > # A tibble: 5 x 3 > height mass birth_year > <int> <dbl> <dbl> > 1 172 77 19 > 2 167 75 112 > 3 96 32 33 > 4 202 136 41.9 > 5 150 49 19 ``` ] --- # Recolocar columnas: relocate() Fíjate que con `select()` podrías además recolocar columnas, indícandole el orden, ayudándote también del selector `everything()` ```r starwars %>% select(c(species, name, birth_year, everything())) ``` Para facilitar la recolocación tenemos una función para ello, `relocate()`, indicándole en `.after` o `.before` detrás o delante de qué columnas queremos moverlas. ```r starwars %>% relocate(species, .before = name) ``` ``` > # A tibble: 87 x 14 > species name height mass hair_color skin_color eye_color birth_year sex > <chr> <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> > 1 Human Luke S~ 172 77 blond fair blue 19 male > 2 Droid C-3PO 167 75 <NA> gold yellow 112 none > 3 Droid R2-D2 96 32 <NA> white, bl~ red 33 none > 4 Human Darth ~ 202 136 none white yellow 41.9 male > 5 Human Leia O~ 150 49 brown light brown 19 fema~ > 6 Human Owen L~ 178 120 brown, gr~ light blue 52 male > 7 Human Beru W~ 165 75 brown light blue 47 fema~ > 8 Droid R5-D4 97 32 <NA> white, red red NA none > 9 Human Biggs ~ 183 84 black light brown 24 male > 10 Human Obi-Wa~ 182 77 auburn, w~ fair blue-gray 57 male > # ... with 77 more rows, and 5 more variables: gender <chr>, homeworld <chr>, > # films <list>, vehicles <list>, starships <list> ``` --- # Extraer columnas: pull() .pull-left[ Si observas la salida de los `select()`, sigue siendo una tabla `tibble`, nos preserva la naturaleza de nuestros datos. ```r starwars %>% select(name) ``` ``` > # A tibble: 87 x 1 > name > <chr> > 1 Luke Skywalker > 2 C-3PO > 3 R2-D2 > 4 Darth Vader > 5 Leia Organa > 6 Owen Lars > 7 Beru Whitesun lars > 8 R5-D4 > 9 Biggs Darklighter > 10 Obi-Wan Kenobi > # ... with 77 more rows ``` ] .pull-right[ A veces no querremos dicha estructura sino **extraer literalmente la columna en un vector**, algo que podemos hacer con `pull()` ```r starwars %>% pull(name) ``` ``` > [1] "Luke Skywalker" "C-3PO" "R2-D2" > [4] "Darth Vader" "Leia Organa" "Owen Lars" > [7] "Beru Whitesun lars" "R5-D4" "Biggs Darklighter" > [10] "Obi-Wan Kenobi" "Anakin Skywalker" "Wilhuff Tarkin" > [13] "Chewbacca" "Han Solo" "Greedo" > [16] "Jabba Desilijic Tiure" "Wedge Antilles" "Jek Tono Porkins" > [19] "Yoda" "Palpatine" "Boba Fett" > [22] "IG-88" "Bossk" "Lando Calrissian" > [25] "Lobot" "Ackbar" "Mon Mothma" > [28] "Arvel Crynyd" "Wicket Systri Warrick" "Nien Nunb" > [31] "Qui-Gon Jinn" "Nute Gunray" "Finis Valorum" > [34] "Jar Jar Binks" "Roos Tarpals" "Rugor Nass" > [37] "Ric Olié" "Watto" "Sebulba" > [40] "Quarsh Panaka" "Shmi Skywalker" "Darth Maul" > [43] "Bib Fortuna" "Ayla Secura" "Dud Bolt" > [46] "Gasgano" "Ben Quadinaros" "Mace Windu" > [49] "Ki-Adi-Mundi" "Kit Fisto" "Eeth Koth" > [52] "Adi Gallia" "Saesee Tiin" "Yarael Poof" > [55] "Plo Koon" "Mas Amedda" "Gregar Typho" > [58] "Cordé" "Cliegg Lars" "Poggle the Lesser" > [61] "Luminara Unduli" "Barriss Offee" "Dormé" > [64] "Dooku" "Bail Prestor Organa" "Jango Fett" > [67] "Zam Wesell" "Dexter Jettster" "Lama Su" > [70] "Taun We" "Jocasta Nu" "Ratts Tyerell" > [73] "R4-P17" "Wat Tambor" "San Hill" > [76] "Shaak Ti" "Grievous" "Tarfful" > [79] "Raymus Antilles" "Sly Moore" "Tion Medon" > [82] "Finn" "Rey" "Poe Dameron" > [85] "BB8" "Captain Phasma" "Padmé Amidala" ``` ] --- # Renombrar columnas: rename() .pull-left[ A veces también podemos querer **modificar la «metainformación» de los datos**, renombrando las columnas. Para ello usaremos la función `rename()` poniendo **primero el nombre nuevo y luego el antiguo**. Como ejemplo, vamos a traducir el nombre de las columnas `name, height, mass` a castellano. ```r starwars %>% rename(nombre = name, altura = height, peso = mass) ``` ``` > # A tibble: 87 x 14 > nombre altura peso hair_color skin_color eye_color birth_year sex gender > <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr> > 1 Luke Sk~ 172 77 blond fair blue 19 male mascu~ > 2 C-3PO 167 75 <NA> gold yellow 112 none mascu~ > 3 R2-D2 96 32 <NA> white, bl~ red 33 none mascu~ > 4 Darth V~ 202 136 none white yellow 41.9 male mascu~ > 5 Leia Or~ 150 49 brown light brown 19 fema~ femin~ > 6 Owen La~ 178 120 brown, gr~ light blue 52 male mascu~ > 7 Beru Wh~ 165 75 brown light blue 47 fema~ femin~ > 8 R5-D4 97 32 <NA> white, red red NA none mascu~ > 9 Biggs D~ 183 84 black light brown 24 male mascu~ > 10 Obi-Wan~ 182 77 auburn, w~ fair blue-gray 57 male mascu~ > # ... with 77 more rows, and 5 more variables: homeworld <chr>, species <chr>, > # films <list>, vehicles <list>, starships <list> ``` ] .pull-right[ <div class="figure" style="text-align: center"> <img src="./img/rename1.jpg" alt="Flujo de https://tidydatatutor.com/" width="150%" /> <p class="caption">Flujo de https://tidydatatutor.com/</p> </div> ] --- # Modificadores: at, if Muchas de las funciones vistas pueden ir acompañadas de `at` o `if`, como `rename_if`, que nos permite hacer operaciones más personalizadas. Por ejemplo, vamos a renombrar solo las columnas que sean de tipo numérico, y además les aplicaremos la función `toupper()`, pasándolas a mayúsculas. ```r starwars %>% rename_if(is.numeric, toupper) ``` ``` > # A tibble: 87 x 14 > name HEIGHT MASS hair_color skin_color eye_color BIRTH_YEAR sex gender > <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr> > 1 Luke Sk~ 172 77 blond fair blue 19 male mascu~ > 2 C-3PO 167 75 <NA> gold yellow 112 none mascu~ > 3 R2-D2 96 32 <NA> white, bl~ red 33 none mascu~ > 4 Darth V~ 202 136 none white yellow 41.9 male mascu~ > 5 Leia Or~ 150 49 brown light brown 19 fema~ femin~ > 6 Owen La~ 178 120 brown, gr~ light blue 52 male mascu~ > 7 Beru Wh~ 165 75 brown light blue 47 fema~ femin~ > 8 R5-D4 97 32 <NA> white, red red NA none mascu~ > 9 Biggs D~ 183 84 black light brown 24 male mascu~ > 10 Obi-Wan~ 182 77 auburn, w~ fair blue-gray 57 male mascu~ > # ... with 77 more rows, and 5 more variables: homeworld <chr>, species <chr>, > # films <list>, vehicles <list>, starships <list> ``` --- # Ejercicios: select() y rename() .panelset[ .panel[.panel-name[Ejercicios] * 📝 **Ejercicio 1**: filtra el conjunto de personajes y quédate solo con aquellos que en la variable `height` no tengan un dato ausente. * 📝 **Ejercicio 2**: con los datos obtenidos del filtro anterior, selecciona solo las variables `name`, `height`, así como todas aquellas variables que CONTENGAN la palabra `color` en su nombre. * 📝 **Ejercicio 3**: con los datos obtenidos del ejercicio, traduce el nombre de las columnas a castellano * 📝 **Ejercicio 4**: con los datos obtenidos del ejercicio, coloca la variable de color de pelo justo detrás de la variable de nombres. * 📝 **Ejercicio 5**: con los datos obtenidos del ejercicio, comprueba cuántas modalidades únicas hay en la variable de color de pelo. ] .panel[.panel-name[Sol. Ej. 1] **IMPORTANTE**: todo lo que hagas en la tabla original, si el resultado final no se lo asignas `<-` a otra variable, lo verás en consola pero no se guardará en ningún sitio. Lo que no guardes, no existe. ```r starwars_NA <- starwars %>% drop_na(height) starwars_NA ``` ``` > # A tibble: 81 x 14 > name height mass hair_color skin_color eye_color birth_year sex gender > <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr> > 1 Luke Sk~ 172 77 blond fair blue 19 male mascu~ > 2 C-3PO 167 75 <NA> gold yellow 112 none mascu~ > 3 R2-D2 96 32 <NA> white, bl~ red 33 none mascu~ > 4 Darth V~ 202 136 none white yellow 41.9 male mascu~ > 5 Leia Or~ 150 49 brown light brown 19 fema~ femin~ > 6 Owen La~ 178 120 brown, gr~ light blue 52 male mascu~ > 7 Beru Wh~ 165 75 brown light blue 47 fema~ femin~ > 8 R5-D4 97 32 <NA> white, red red NA none mascu~ > 9 Biggs D~ 183 84 black light brown 24 male mascu~ > 10 Obi-Wan~ 182 77 auburn, w~ fair blue-gray 57 male mascu~ > # ... with 71 more rows, and 5 more variables: homeworld <chr>, species <chr>, > # films <list>, vehicles <list>, starships <list> ``` ] .panel[.panel-name[Sol. Ej. 2] ```r starwars %>% drop_na(height) %>% select(c(name, height, contains("color"))) ``` ``` > # A tibble: 81 x 5 > name height hair_color skin_color eye_color > <chr> <int> <chr> <chr> <chr> > 1 Luke Skywalker 172 blond fair blue > 2 C-3PO 167 <NA> gold yellow > 3 R2-D2 96 <NA> white, blue red > 4 Darth Vader 202 none white yellow > 5 Leia Organa 150 brown light brown > 6 Owen Lars 178 brown, grey light blue > 7 Beru Whitesun lars 165 brown light blue > 8 R5-D4 97 <NA> white, red red > 9 Biggs Darklighter 183 black light brown > 10 Obi-Wan Kenobi 182 auburn, white fair blue-gray > # ... with 71 more rows ``` ] .panel[.panel-name[Sol. Ej. 3] ```r starwars %>% drop_na(height) %>% select(c(name, height, contains("color"))) %>% rename(nombre = name, altura = height, color_pelo = hair_color, color_piel = skin_color, color_ojos = eye_color) ``` ``` > # A tibble: 81 x 5 > nombre altura color_pelo color_piel color_ojos > <chr> <int> <chr> <chr> <chr> > 1 Luke Skywalker 172 blond fair blue > 2 C-3PO 167 <NA> gold yellow > 3 R2-D2 96 <NA> white, blue red > 4 Darth Vader 202 none white yellow > 5 Leia Organa 150 brown light brown > 6 Owen Lars 178 brown, grey light blue > 7 Beru Whitesun lars 165 brown light blue > 8 R5-D4 97 <NA> white, red red > 9 Biggs Darklighter 183 black light brown > 10 Obi-Wan Kenobi 182 auburn, white fair blue-gray > # ... with 71 more rows ``` ] .panel[.panel-name[Sol. Ej. 4] ```r starwars %>% drop_na(height) %>% select(c(name, height, contains("color"))) %>% rename(nombre = name, altura = height, color_pelo = hair_color, color_piel = skin_color, color_ojos = eye_color) %>% relocate(color_pelo, .after = nombre) ``` ``` > # A tibble: 81 x 5 > nombre color_pelo altura color_piel color_ojos > <chr> <chr> <int> <chr> <chr> > 1 Luke Skywalker blond 172 fair blue > 2 C-3PO <NA> 167 gold yellow > 3 R2-D2 <NA> 96 white, blue red > 4 Darth Vader none 202 white yellow > 5 Leia Organa brown 150 light brown > 6 Owen Lars brown, grey 178 light blue > 7 Beru Whitesun lars brown 165 light blue > 8 R5-D4 <NA> 97 white, red red > 9 Biggs Darklighter black 183 light brown > 10 Obi-Wan Kenobi auburn, white 182 fair blue-gray > # ... with 71 more rows ``` ] .panel[.panel-name[Sol. Ej. 5] ```r starwars %>% drop_na(height) %>% select(c(name, height, contains("color"))) %>% rename(nombre = name, altura = height, color_pelo = hair_color, color_piel = skin_color, color_ojos = eye_color) %>% relocate(color_pelo, .after = nombre) %>% distinct(color_pelo) ``` ``` > # A tibble: 12 x 1 > color_pelo > <chr> > 1 blond > 2 <NA> > 3 none > 4 brown > 5 brown, grey > 6 black > 7 auburn, white > 8 auburn, grey > 9 white > 10 grey > 11 auburn > 12 blonde ``` **IMPORTANTE**: recuerda que `distinct()` de mantener todas las columnas añadiendo `.keep_all = TRUE`. ] ] --- # Modificar/crear variables: mutate() .pull-left[ A veces queremos **modificar o crear variables**. Para ello tenemos la función `mutate()`. ```r # Idea datos %>% filtro(cond1, cond2) %>% ordeno(var1, desc(var2)) %>% selecciono(col1, col2, ...) %>% modifico(var_nueva = funcion(var_existentes)) ``` Para empezar, vamos a crear una **nueva variable** `height_m` con la altura en centímetros. ```r # altura en metros starwars %>% mutate(height_m = height / 100) ``` ``` > # A tibble: 87 x 4 > name mass height height_m > <chr> <dbl> <int> <dbl> > 1 Luke Skywalker 77 172 1.72 > 2 C-3PO 75 167 1.67 > 3 R2-D2 32 96 0.96 > 4 Darth Vader 136 202 2.02 > 5 Leia Organa 49 150 1.5 > 6 Owen Lars 120 178 1.78 > 7 Beru Whitesun lars 75 165 1.65 > 8 R5-D4 32 97 0.97 > 9 Biggs Darklighter 84 183 1.83 > 10 Obi-Wan Kenobi 77 182 1.82 > # ... with 77 more rows ``` ] .pull-right[ <div class="figure" style="text-align: center"> <img src="./img/mutate1.jpg" alt="Flujo de https://tidydatatutor.com/" width="150%" /> <p class="caption">Flujo de https://tidydatatutor.com/</p> </div> ] --- # Modificar/crear variables: mutate() Otra opción es **quedarnos solo con las columnas nuevas** creadas con `transmute()` ```r starwars %>% transmute(height_m = height / 100) ``` ``` > # A tibble: 5 x 1 > height_m > <dbl> > 1 1.72 > 2 1.67 > 3 0.96 > 4 2.02 > 5 1.5 ``` También se pueden aplicar **funciones más complejas** como `if_else()` o `case_when()` ```r starwars %>% mutate(humano = if_else(species == "Human", "humano", "no humano")) ``` ``` > # A tibble: 87 x 5 > name height mass species humano > <chr> <int> <dbl> <chr> <chr> > 1 Luke Skywalker 172 77 Human humano > 2 C-3PO 167 75 Droid no humano > 3 R2-D2 96 32 Droid no humano > 4 Darth Vader 202 136 Human humano > 5 Leia Organa 150 49 Human humano > 6 Owen Lars 178 120 Human humano > 7 Beru Whitesun lars 165 75 Human humano > 8 R5-D4 97 32 Droid no humano > 9 Biggs Darklighter 183 84 Human humano > 10 Obi-Wan Kenobi 182 77 Human humano > # ... with 77 more rows ``` --- # Recategorizar: case_when() Una operación también muy habitual es querer **recategorizar nuestras variables**. Supongamos por ejemplo que queremos crear una **categoría en función de su altura**. Para ello podemos podemos usar `case_when`, en el que le podemos indicar que valores asignamos en función de condiciones. Vamos a crear una variable nueva de altura * Si `height > 180` –> serán `"altos"`. * Si `height <= 180` y `height > 120` –> serán `"bajos"` * Si `height <= 120` y `height > 0` –> serán `"enanos"` * Si no se cumple lo anterior –> serán `"ausentes"` ```r starwars %>% mutate(altura = case_when(height > 180 ~ "altos", height > 120 ~ "bajos", height > 0 ~ "enanos", TRUE ~ "ausentes")) ``` ``` > # A tibble: 87 x 4 > name height mass altura > <chr> <int> <dbl> <chr> > 1 Luke Skywalker 172 77 bajos > 2 C-3PO 167 75 bajos > 3 R2-D2 96 32 enanos > 4 Darth Vader 202 136 altos > 5 Leia Organa 150 49 bajos > 6 Owen Lars 178 120 bajos > 7 Beru Whitesun lars 165 75 bajos > 8 R5-D4 97 32 enanos > 9 Biggs Darklighter 183 84 altos > 10 Obi-Wan Kenobi 182 77 altos > # ... with 77 more rows ``` --- # Ejercicios .panelset[ .panel[.panel-name[Ejercicios] * 📝 **Ejercicio 1**: define una función llamada `IMC()`, que dados dos argumentos `peso` y `estatura` (en metros), nos devuelve el índice de masa corporal ($IMC = \frac{peso}{altura^2}$). * 📝 **Ejercicio 2**: con esa función definida, añade a `starwars` una nueva columna que indique el IMC de cada personaje. * 📝 **Ejercicio 3**: selecciona los 3 personajes con mayor IMC y los 3 con menos. * 📝 **Ejercicio 4**: define una nueva variable llamada `edad` que nos recategorice la variable `birth_year`: `"menor"` para los que tengan menos de 18 años; `"adulto"` entre 18 y 65 años (ambos inclusive); `"mayor"` de 66 a 99 años; y `"centenario"` de 100 en adelante. ] .panel[.panel-name[Sol. 1] Primero definimos la función `IMC` (calcula el IMC dada estatura y peso). ```r # Suponiendo a la altura ya en metros IMC <- function(peso, estatura) { IMC <- peso / estatura^2 return(IMC) } IMC(90, 1.60) ``` ``` > [1] 35.15625 ``` ```r IMC(71, 1.73) ``` ``` > [1] 23.72281 ``` ] .panel[.panel-name[Sol. 2] La función definida como `IMC()` podemos ahora aplicarla dentro del `mutate()`. ```r starwars %>% mutate(height_m = height / 100, IMC = IMC(mass, height_m)) %>% # Las movemos al inicio (por defecto las mete al final) relocate(height_m, IMC, .after = name) ``` ``` > # A tibble: 87 x 16 > name height_m IMC height mass hair_color skin_color eye_color birth_year > <chr> <dbl> <dbl> <int> <dbl> <chr> <chr> <chr> <dbl> > 1 Luke ~ 1.72 26.0 172 77 blond fair blue 19 > 2 C-3PO 1.67 26.9 167 75 <NA> gold yellow 112 > 3 R2-D2 0.96 34.7 96 32 <NA> white, bl~ red 33 > 4 Darth~ 2.02 33.3 202 136 none white yellow 41.9 > 5 Leia ~ 1.5 21.8 150 49 brown light brown 19 > 6 Owen ~ 1.78 37.9 178 120 brown, gr~ light blue 52 > 7 Beru ~ 1.65 27.5 165 75 brown light blue 47 > 8 R5-D4 0.97 34.0 97 32 <NA> white, red red NA > 9 Biggs~ 1.83 25.1 183 84 black light brown 24 > 10 Obi-W~ 1.82 23.2 182 77 auburn, w~ fair blue-gray 57 > # ... with 77 more rows, and 7 more variables: sex <chr>, gender <chr>, > # homeworld <chr>, species <chr>, films <list>, vehicles <list>, > # starships <list> ``` ] .panel[.panel-name[Sol. 3] ```r starwars_IMC <- starwars %>% mutate(height_m = height / 100, IMC = IMC(mass, height_m)) starwars_IMC %>% slice_max(IMC, n = 3) ``` ``` > # A tibble: 3 x 16 > name height mass hair_color skin_color eye_color birth_year sex gender > <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr> > 1 Jabba De~ 175 1358 <NA> green-tan~ orange 600 herm~ mascu~ > 2 Dud Bolt 94 45 none blue, grey yellow NA male mascu~ > 3 Yoda 66 17 white green brown 896 male mascu~ > # ... with 7 more variables: homeworld <chr>, species <chr>, films <list>, > # vehicles <list>, starships <list>, height_m <dbl>, IMC <dbl> ``` ```r starwars_IMC %>% slice_min(IMC, n = 3) ``` ``` > # A tibble: 3 x 16 > name height mass hair_color skin_color eye_color birth_year sex gender > <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr> > 1 Wat Tamb~ 193 48 none green, gr~ unknown NA male mascu~ > 2 Adi Gall~ 184 50 none dark blue NA fema~ femin~ > 3 Sly Moore 178 48 none pale white NA <NA> <NA> > # ... with 7 more variables: homeworld <chr>, species <chr>, films <list>, > # vehicles <list>, starships <list>, height_m <dbl>, IMC <dbl> ``` ] .panel[.panel-name[Sol. 4] ```r starwars %>% mutate(edad = case_when(birth_year < 18 ~ "menor", between(birth_year, 18, 65) ~ "adulto", between(birth_year, 66, 99) ~ "mayor", TRUE ~ "centenario")) ``` ``` > # A tibble: 87 x 15 > name height mass hair_color skin_color eye_color birth_year sex gender > <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr> > 1 Luke Sk~ 172 77 blond fair blue 19 male mascu~ > 2 C-3PO 167 75 <NA> gold yellow 112 none mascu~ > 3 R2-D2 96 32 <NA> white, bl~ red 33 none mascu~ > 4 Darth V~ 202 136 none white yellow 41.9 male mascu~ > 5 Leia Or~ 150 49 brown light brown 19 fema~ femin~ > 6 Owen La~ 178 120 brown, gr~ light blue 52 male mascu~ > 7 Beru Wh~ 165 75 brown light blue 47 fema~ femin~ > 8 R5-D4 97 32 <NA> white, red red NA none mascu~ > 9 Biggs D~ 183 84 black light brown 24 male mascu~ > 10 Obi-Wan~ 182 77 auburn, w~ fair blue-gray 57 male mascu~ > # ... with 77 more rows, and 6 more variables: homeworld <chr>, species <chr>, > # films <list>, vehicles <list>, starships <list>, edad <chr> ``` ] ] --- # Contar: count() Tenemos además la función `count()` que nos permitirá contar los datos. ```r # Idea datos %>% filtro(cond1, cond2) %>% ordeno(var1, desc(var2)) %>% selecciono(col1, col2, ...) %>% modifico(var_nueva = funcion(var_existentes)) %>% cuento(var1, var2) ``` Cuando lo usamos en solitario, `count()` nos devolverá simplemente el **número de registros** ```r starwars %>% count() ``` ``` > # A tibble: 1 x 1 > n > <int> > 1 87 ``` --- # Contar: count() Cuando lo usamos pasándole como argumento una o varias variables, `count()` nos cuenta lo que se conoce en estadística como **frecuencias absolutas**: el número de elementos pertenecientes a cada una de las **modalidades**. En el caso por ejemplo del fichero de `starwars` `sex` tiene 4 modalidades `female, hermaphroditic, male, none`. ```r starwars %>% count(sex) ``` ``` > # A tibble: 5 x 2 > sex n > <chr> <int> > 1 female 16 > 2 hermaphroditic 1 > 3 male 60 > 4 none 6 > 5 <NA> 4 ``` Si pasamos varias variables nos calcula las **frecuencias absolutas n-dimensionales** ```r starwars %>% count(sex, gender) ``` ``` > # A tibble: 6 x 3 > sex gender n > <chr> <chr> <int> > 1 female feminine 16 > 2 hermaphroditic masculine 1 > 3 male masculine 60 > 4 none feminine 1 > 5 none masculine 5 > 6 <NA> <NA> 4 ``` --- # Paréntesis: estadística descriptiva En estadística, como en probabilidad, podemos distinguir las variables en función de las modalidades permitidas en dos grandes categorías: * **Variables categóricas (nominales o cualitativas)**. Son variables que representan categorías o cualidades. Ejemplos: color, forma, estado civil, religión, etc. Estas variables las podemos **subdividir en función de si admiten o no un orden**: - **Cualitativas ordinales**: aunque representen cualidades, tienen una jerarquía de orden. Ejemplos: suspenso-aprobado-notable, sano - herido leve - grave, etc. - **Cualitativas nominales**: no admiten (salvo problemas nuestros) una jerarquía de orden. Ejemplos: ateo-católico-musulmán, soltero-casado, homber-mujer, etc. * **Variables cuantitativas**: representan una **cantidad numérica** medible, una característica cuantificable matemáticamente. A su vez se pueden subdividir en dos grupos. - **Cuantitativas discretas**: se pueden **contar** y **enumerar (aunque sean infinitos)**, detrás de un valor puedo saber cuál viene después (personas, granos de arena, etc). - **Cuantitativas continuas**: no solo toman infinitos valores sino que entre dos valores cualesquiera, también hay infinitos términos, no se puede determinar el siguiente valor a uno dado (estaturas, pesos, temperatura, etc). --- # Paréntesis: estadística descriptiva En estadística llamamos **medidas de centralización** a aquellas medidas que nos indican en torno a qué **valores se concentran los datos** * **Media**: medida de centralización basada en el valor que nos **minimiza el promedio de desviaciones al cuadrado**. La media es el valor que está más «cerca» de todos los puntos a la vez, pero **SOLO se puede calcular para variables cuantitativas**: solo podemos calcular medias de números. * **Mediana**: medida de centralización definida como el **valor que ocupa el centro de los datos** cuando los **ordenamos de menor a mayor**, un valor que nos deja por debajo al menos el 50% y por encima al menos el 50%, y **SOLO se puede calcular para variables cuantitativas** o **variables cualitatives ordinales** (solo podemos calcular el valor de en medio en datos que permitan ser ordenados). * **Moda**: medida de centralización definida como el **valor más repetido**. Al contrario que la media y la mediana, la moda **no tiene porque ser única**: podemos tener un empate. Si todos se repiten por igual, al no destacar ninguno, se dice que es **amodal**. Se puede **calcular para todo tipo de variables**. --- # Paréntesis: estadística descriptiva .pull-left[ Es importante saber las **diferencias entre las medidas de centralización** respecto a lo que en estadística se llama **robustez** * La **media es muy poco robusta**: valores extremos la perturban muy fácil. Piensa en una baraja de cartas: si extraemos 10 ases, la media será 1. Si cambiamos uno por un 10, solo por cambiar el 10% de los datos la media casi se duplica. ```r mean(c(1, 1, 1, 1, 1, 1, 1, 1, 1, 10)) ``` ``` > [1] 1.9 ``` * La **mediana es algo más robusta** pero la **moda es la más robusta**: para que cambie en el ejemplo se necesitarían cambiar más de la mitad de los datos Y ADEMÁS que los nuevos sean todos repetidos. ] .pull-right[ 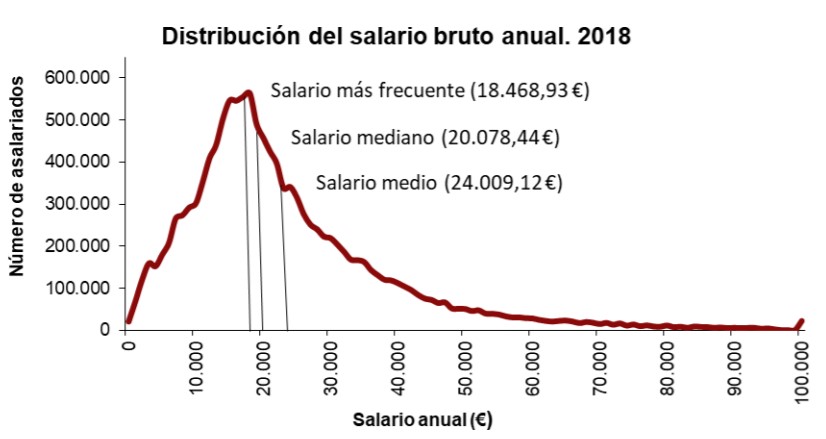 La **falta de robustez** de la media es la razón por la que, a la hora de analizar por ejemplo datos tan desiguales como los salarios en España, el **salario medio** no nos aporta realmente información útil sobre la centralidad de los datos, ya que personas extremadamente ricas hacen que la media sea bastante más elevada que el **salario más frecuente**. ] --- # Paréntesis: estadística descriptiva De la misma manera podemos pedirle que nos calcule **medidas de dispersión** (varianza, desv. típica y cv), que nos indican cómo de **dispersos están los datos respecto a un centro**, normalmente la media, y **medidas de localización (percentiles)**, valores que nos parten los datos en trozos iguales) El **coeficiente de variación (CV)** es una medida de dispersión adimensional: nos permita comparar entre variables para saber cuál es más dispersa. --- # Agrupar: group_by() Una de las funciones más potentes es `group_by()` que nos permitirá **agrupar nuestros registros** ```r # Idea datos %>% filtro() %>% ordeno() %>% selecciono() %>% modifico() %>% agrupo(var1, var2, ...) %>% cuento(var) %>% * desagrupo() ``` Cuando aplicamos `group_by()` es importante entender que **NO MODIFICA los datos**: nos crea una variable de grupo que **modificará las acciones futuras que apliquemos**. Internamente, es como generar **múltiples subtablas**, y las operaciones aplicadas después se **aplicarán a cada una por separado**. --- # Agrupar: group_by() .pull-left[ Por ejemplo, vamos a **agrupar por las variables `sex` y `gender`**, y después aplicaremos `count()`: ya no realiza el conteo de filas en general, sino que cuenta los registros para cada par sexo-género distinto (realiza la acción en cada subtabla). ```r starwars %>% * group_by(sex, gender) %>% count() %>% * ungroup() ``` ``` > # A tibble: 6 x 3 > sex gender n > <chr> <chr> <int> > 1 female feminine 16 > 2 hermaphroditic masculine 1 > 3 male masculine 60 > 4 none feminine 1 > 5 none masculine 5 > 6 <NA> <NA> 4 ``` **IMPORTANTE**: siempre que agrupes, acuérdate de desagrupar con `ungroup()`. ] .pull-right[ <div class="figure" style="text-align: center"> <img src="./img/group_1.jpg" alt="Flujo de https://tidydatatutor.com/" width="150%" /> <p class="caption">Flujo de https://tidydatatutor.com/</p> </div> <div class="figure" style="text-align: center"> <img src="./img/group_count.jpg" alt="Flujo de https://tidydatatutor.com/" width="150%" /> <p class="caption">Flujo de https://tidydatatutor.com/</p> </div> ] --- # Resumir: summarise() La función `summarise()` unida a `group_by()` es de las más útiles de `{tidyverse}`: nos permite **sumarizar o resumir nuestros datos**. ```r # Idea datos %>% filtro() %>% selecciono() %>% * agrupo(var1, var2, ...) %>% resumo() %>% * desagrupo() ``` Un ejemplo: vamos a calcular la **media de las alturas**. Si lo hacemos sin `group_by()` se hará de todos los personajes. ```r starwars %>% drop_na(height) %>% * summarise(media_altura = mean(height)) ``` ``` > # A tibble: 1 x 1 > media_altura > <dbl> > 1 174. ``` --- # Resumir: summarise() .pull-left[ ```r # Idea datos %>% filtro() %>% selecciono() %>% * agrupo(var1, var2, ...) %>% resumo() %>% * desagrupo() ``` Si la misma acción la realizamos con un `group_by()` previo, la media de las alturas se hará ahora **de forma independiente, en cada subtabla por grupo creada**. ```r starwars %>% drop_na(height) %>% group_by(sex) %>% summarise(media = mean(height)) %>% ungroup() ``` ``` > # A tibble: 5 x 2 > sex media > <chr> <dbl> > 1 female 169. > 2 hermaphroditic 175 > 3 male 179. > 4 none 131. > 5 <NA> 181. ``` ] .pull-right[ <div class="figure" style="text-align: center"> <img src="./img/group_summarise.jpg" alt="Flujo de https://tidydatatutor.com/" width="150%" /> <p class="caption">Flujo de https://tidydatatutor.com/</p> </div> ] --- # Resumir: summarise() Podemos **resumir varias variables a la vez**, haciendo uso de la función `across()` * primero indicamos **las variables a recorrer** (en este caso `height:mass`) * después indicamos la función a aplicar (en este caso, la media `mean()` * por último indicamos argumentos extras si fuesen necesarios (en este caso, `na.rm = TRUE` para eliminar los ausentes en la media) ```r starwars %>% * group_by(sex) %>% * summarise(medias = across(height:mass, mean, na.rm = TRUE)) %>% ungroup() ``` ``` > # A tibble: 5 x 2 > sex medias$height $mass > <chr> <dbl> <dbl> > 1 female 169. 54.7 > 2 hermaphroditic 175 1358 > 3 male 179. 81.0 > 4 none 131. 69.8 > 5 <NA> 181. 48 ``` --- # Resumir: summarise() Además la función `across()` podemos combinarla con `where()`, para que resuma las variables de un mismo tipo, por ejemplo `where(is.numeric)` selecciona solo las numéricas. ```r starwars %>% drop_na(height, mass) %>% group_by(sex, gender) %>% summarise(across(where(is.numeric), mean)) %>% ungroup() ``` ``` > # A tibble: 5 x 5 > sex gender height mass birth_year > <chr> <chr> <dbl> <dbl> <dbl> > 1 female feminine 169. 54.7 NA > 2 hermaphroditic masculine 175 1358 600 > 3 male masculine 178. 81.0 NA > 4 none masculine 140 69.8 NA > 5 <NA> <NA> 178 48 NA ``` Algunas de las funciones más útiles dentro de `summarise()`: * `sum()`, `mean()`, `median()` * `min()`, `which.min()`, `max()`, `which.max()` * `n()` (número de registros), `n_distinct()` (número de registros únicos). --- # Resumir: skim() En el paquete `{skimr}` tenemos una función `skim()` que noes permite **resumir rápidamente nuestras variables**, proporcionándonos los principales parámetros de centralización, dispersión y localización. ```r library(skimr) starwars %>% select(name:gender) %>% skim() ``` --- # Operaciones por filas: rowwise() Una opción muy útil usada antes de una operación también es `rowwise()`: toda operación que venga después se aplicará **en cada fila por separado**. Por ejemplo, vamos a definir un conjunto dummy de notas. ```r notas <- tibble("mates" = sample(0:10, size = 4, replace = TRUE), "lengua" = sample(0:10, size = 4, replace = TRUE)) ``` Si aplicamos la media directamente, en cada fila el valor `media_curso` es **idéntico ya que nos ha hecho la media global**. En realidad a nosotros nos gustaría **sacar una media por registro, por alumno**. Para eso usaremos `rowwise()` antes del cálculo. ```r # Aplicado por fila notas %>% rowwise() %>% mutate(media_curso = mean(c(mates, lengua))) ``` ``` > # A tibble: 4 x 3 > # Rowwise: > mates lengua media_curso > <int> <int> <dbl> > 1 5 0 2.5 > 2 3 8 5.5 > 3 3 9 6 > 4 10 8 9 ``` --- # Ejercicios .panelset[ .panel[.panel-name[Ejercicios] * 📝 **Ejercicio 1**: con `group_by()` y `count()`, extrae cuántas personajes a cada especie de `starwars` * 📝 **Ejercicio 2**: determina con `summarise()` y `n_distinct()` el número de especies distintas * 📝 **Ejercicio 3**: tras eliminar ausentes de peso y altura, añade una nueva variable que nos calcule el IMC de cada personaje, y determina el IMC medio de nuestros datos desagregados por sexo. * 📝 **Ejercicio 4**: obtén la edad mínima y máxima de cada sexo. ] .panel[.panel-name[Sol. Ej. 1] ```r starwars %>% group_by(species) %>% count() %>% ungroup() ``` ``` > # A tibble: 38 x 2 > species n > <chr> <int> > 1 Aleena 1 > 2 Besalisk 1 > 3 Cerean 1 > 4 Chagrian 1 > 5 Clawdite 1 > 6 Droid 6 > 7 Dug 1 > 8 Ewok 1 > 9 Geonosian 1 > 10 Gungan 3 > # ... with 28 more rows ``` ] .panel[.panel-name[Sol. Ej. 2] ```r starwars %>% summarise(especies_distintas = n_distinct(species)) ``` ``` > # A tibble: 1 x 1 > especies_distintas > <int> > 1 38 ``` ] .panel[.panel-name[Sol. Ej. 3] ```r starwars %>% drop_na(height, mass) %>% mutate(IMC = mass / (height/100)^2) %>% group_by(sex) %>% summarise(media_IMC = mean(IMC)) %>% ungroup() ``` ``` > # A tibble: 5 x 2 > sex media_IMC > <chr> <dbl> > 1 female 19.2 > 2 hermaphroditic 443. > 3 male 25.7 > 4 none 32.7 > 5 <NA> 15.1 ``` ] .panel[.panel-name[Sol. Ej. 4] ```r starwars %>% drop_na(birth_year) %>% group_by(sex) %>% summarise(edad_min = min(birth_year), edad_max = max(birth_year)) ``` ``` > # A tibble: 5 x 3 > sex edad_min edad_max > <chr> <dbl> <dbl> > 1 female 19 72 > 2 hermaphroditic 600 600 > 3 male 8 896 > 4 none 15 112 > 5 <NA> 62 62 ``` ] ] --- name: purr class: center, middle # Introducción al manejo de listas ## **Concatenando cualquier objeto** Las listas son un tipo de objeto que permite incluir cualquier tipo de dato y longitud, y el paquete `{purrr}` de `{tidyverse}` permite su manejo como si fuesen vectores. --- # Purrr: listas Las **listas** son el tipo de dato **más flexible** ya que permite **concatenar variables de diferente tipo pero también de diferente longitud**, con estructuras totalmente heterógeneas. Un ejemplo: vamos a crear una lista con un vector de textos (nombre de padres), un valor lógico (si están casado o no) y un vector numérico (edades de los miembros que viven con nosotros) ```r var1 <- c("Paloma", "Gregorio") var2 <- TRUE var3 <- c(61, 62, 28) ``` Para reunirlas en un solo objeto de tipo lista basta con usar `list()`, indicándole si queremos el nombre de cada elemento. ```r lista <- list("nombres_padres" = var1, "casados" = var2, "edades_familia" = var3) lista ``` ``` > $nombres_padres > [1] "Paloma" "Gregorio" > > $casados > [1] TRUE > > $edades_familia > [1] 61 62 28 ``` --- # Purrr: listas * **Longitud**: las variables de tipo lista solo tienen una dimensión, como un vector (solo que en cada elemento hay a su vez otro objeto) ```r length(lista) ``` ``` > [1] 3 ``` Si los juntásemos con un data.frame o tibble, al tener distinta longitud, obtendríamos un error: ```r data.frame("nombres_padres" = var1, "casados" = var2, "edades_familia" = var3) ``` ``` > Error in data.frame(nombres_padres = var1, casados = var2, edades_familia = var3): arguments imply differing number of rows: 2, 1, 3 ``` --- # Purrr: listas Para **acceder a un elemento de la lista** tenemos dos opciones: * **Acceder por índice**: con el operador `[[i]]` accedemos al elemento i-ésimo de la lista (con el corchete doble, en contraposición con el corchete simple que nos permite acceder a varios elementos a la vez) * **Acceder por nombre**: con el operador `$` accedemos al elemento por su nombre ```r # Accedemos por índice lista[[1]] ``` ``` > [1] "Paloma" "Gregorio" ``` ```r lista$progenitores ``` ``` > NULL ``` ```r lista[1:2] ``` ``` > $nombres_padres > [1] "Paloma" "Gregorio" > > $casados > [1] TRUE ``` --- # Purrr: listas Una lista no se puede vectorizar de forma inmediata, por lo cualquier operación aritmética aplicada a una lista dará error. ```r datos <- list("a" = 1:5, "b" = 10:20) datos ``` ``` > $a > [1] 1 2 3 4 5 > > $b > [1] 10 11 12 13 14 15 16 17 18 19 20 ``` ```r datos / 2 ``` ``` > Error in datos/2: argumento no-numérico para operador binario ``` Para ello están disponibles las funciones del paquete `{purrr}`. --- # Purrr: listas La más sencilla es `map()`, que **recorre cada elemento de la lista** y nos aplica la función que le indiquemos. ```r datos %>% map(mean) ``` ``` > $a > [1] 3 > > $b > [1] 15 ``` Puede ser una función predefinida o una propia ```r datos %>% map(function(x) { x / 2}) ``` ``` > $a > [1] 0.5 1.0 1.5 2.0 2.5 > > $b > [1] 5.0 5.5 6.0 6.5 7.0 7.5 8.0 8.5 9.0 9.5 10.0 ``` --- # Purrr: listas Si te fijas por defecto `map()` devuelve a su vez una lista ```r datos %>% map(mean) ``` ``` > $a > [1] 3 > > $b > [1] 15 ``` ¿Cómo indicarle que **queremos que nos devuelva un vector numérico** con las medias? Tenemos a nuestra disposición distintas funciones `map_xxx()`, como `map_dbl()`, para indicarle el formato de salida ```r datos %>% map_dbl(mean) ``` ``` > a b > 3 15 ``` --- name: joins class: center, middle # Relacionando datos ## **Cruzando tablas con joins** No siempre tenemos la información en una sola tabla y a veces nos interesará cruzar las tablas por algún identificador como un DNI o un id. --- # Relacionando datos: join Un clásico de todo lenguaje que maneja datos son los famosos **join**: siempre que veamos un join, lo debemos imaginar es que estamos **cruzando una o variables tablas**, haciendo uso de una columna identificadora de cada una de ellas (por ejemplo, imagina que cruzamos datos de hacienda y de antecedentes penales, haciendo _join_ por la columna `DNI`). <div class="figure" style="text-align: center"> <img src="https://dadosdelaplace.github.io/courses-ECI-2022/img/sql-joins.jpg" alt="Esquema con los principales tipos de join, extraído de https://estradawebgroup.com/Post/-Que-es-y-para-que-sirve-SQL-Joins-/4278" width="60%" /> <p class="caption">Esquema con los principales tipos de join, extraído de https://estradawebgroup.com/Post/-Que-es-y-para-que-sirve-SQL-Joins-/4278</p> </div> --- # Relacionando datos: join .pull-left[ ```r tabla_1 %>% xxx_join(tabla_2) ``` Disponemos de distintos tipos de join: * `inner_join()`: solo **sobreviven los registros cuyos id esté en ambas** tablas. * `left_join()`: **mantiene todos los registros de la primera tabla**, y busca cuales tienen id también en la segunda (en caso de no tenerlo, se rellena con ausente los campos de la segunda tabla). * `right_join()`: **mantiene todos los registros de la segunda tabla**, y busca cuales tienen id también en la primera (en caso de no tenerlo, se rellena con ausente los campos de la primera tabla). * `anti_join()`: nos devuelve los **registros que estén en la primera tabla y no en la segunda**. ] .pull-right[ <div class="figure" style="text-align: center"> <img src="https://dadosdelaplace.github.io/courses-ECI-2022/img/left_join.jpg" alt="Esquema del left join" width="50%" /> <p class="caption">Esquema del left join</p> </div> <div class="figure" style="text-align: center"> <img src="https://dadosdelaplace.github.io/courses-ECI-2022/img/right_join.jpg" alt="Esquema del right join" width="50%" /> <p class="caption">Esquema del right join</p> </div> <div class="figure" style="text-align: center"> <img src="https://dadosdelaplace.github.io/courses-ECI-2022/img/full_join.jpg" alt="Esquema del full join, extraído de https://r4ds.had.co.nz/relational-data.html#mutating-joins" width="50%" /> <p class="caption">Esquema del full join, extraído de https://r4ds.had.co.nz/relational-data.html#mutating-joins</p> </div> ] --- # Relacionando datos: join Para los ejemolos usaremos las tablas disponibles en el paquete `{nycflights13}` ```r library(nycflights13) ``` * airlines: nombre de aerolíneas (con su abreviatura). * airports: datos de aeropuertos (nombres, longitud, latitud, altitud, etc). * flights: datos de vuelos. * planes: datos de los aviones. * weather: datos meteorológicos horarios de las estaciones LGA, JFK y EWR. ```r # Seleccionamos antes columnas para que sea más corto flights_filtrada <- flights %>% select(year:day, arr_time, carrier:dest) flights_filtrada ``` ``` > # A tibble: 336,776 x 9 > year month day arr_time carrier flight tailnum origin dest > <int> <int> <int> <int> <chr> <int> <chr> <chr> <chr> > 1 2013 1 1 830 UA 1545 N14228 EWR IAH > 2 2013 1 1 850 UA 1714 N24211 LGA IAH > 3 2013 1 1 923 AA 1141 N619AA JFK MIA > 4 2013 1 1 1004 B6 725 N804JB JFK BQN > 5 2013 1 1 812 DL 461 N668DN LGA ATL > 6 2013 1 1 740 UA 1696 N39463 EWR ORD > 7 2013 1 1 913 B6 507 N516JB EWR FLL > 8 2013 1 1 709 EV 5708 N829AS LGA IAD > 9 2013 1 1 838 B6 79 N593JB JFK MCO > 10 2013 1 1 753 AA 301 N3ALAA LGA ORD > # ... with 336,766 more rows ``` --- # Left join .pull-left[ * `left_join()`: **mantiene todos los registros de la primera tabla**, y busca cuales tienen id también en la segunda (en caso de no tenerlo, se rellena con ausente los campos de la segunda tabla). ```r flights_filtrada %>% left_join(airlines) ``` ] .pull-right[ <div class="figure" style="text-align: center"> <img src="https://dadosdelaplace.github.io/courses-ECI-2022/img/left_join.jpg" alt="Esquema del left join" width="60%" /> <p class="caption">Esquema del left join</p> </div> ] Queremos **TODAS las filas de los vuelos** con la información extra que tenemos de la aerolínea que opere los vuelos. El campo común que nos permite cruzarla, la **clave (key)**, es el código abreviado de las aerolíneas (`by = carrier`). ```r flights_filtrada %>% left_join(airlines, by = "carrier") ``` ``` > # A tibble: 336,776 x 10 > year month day arr_time carrier flight tailnum origin dest name > <int> <int> <int> <int> <chr> <int> <chr> <chr> <chr> <chr> > 1 2013 1 1 830 UA 1545 N14228 EWR IAH United Air Li~ > 2 2013 1 1 850 UA 1714 N24211 LGA IAH United Air Li~ > 3 2013 1 1 923 AA 1141 N619AA JFK MIA American Airl~ > 4 2013 1 1 1004 B6 725 N804JB JFK BQN JetBlue Airwa~ > 5 2013 1 1 812 DL 461 N668DN LGA ATL Delta Air Lin~ > 6 2013 1 1 740 UA 1696 N39463 EWR ORD United Air Li~ > 7 2013 1 1 913 B6 507 N516JB EWR FLL JetBlue Airwa~ > 8 2013 1 1 709 EV 5708 N829AS LGA IAD ExpressJet Ai~ > 9 2013 1 1 838 B6 79 N593JB JFK MCO JetBlue Airwa~ > 10 2013 1 1 753 AA 301 N3ALAA LGA ORD American Airl~ > # ... with 336,766 more rows ``` --- # Left join La tabla `airlines` tenía 2 columnas, una la clave y otra con el nombre `name` de la aerolínea. ¿Y si en flights hay algún vuelo operado por alguna aerolínea que no estuviese en esa segunda tabla `airlines`? Al estar haciendo `left_join()`, nos mantendrá todos los registros de la primera tabla pero si no los encuentra en la segunda, imputará ausente. ```r # Eliminamos una aerolínea airlines_sin_UA <- airlines %>% filter(!(carrier %in% c("UA"))) flights_filtrada %>% left_join(airlines_sin_UA, by = "carrier") ``` ``` > # A tibble: 336,776 x 10 > year month day arr_time carrier flight tailnum origin dest name > <int> <int> <int> <int> <chr> <int> <chr> <chr> <chr> <chr> > 1 2013 1 1 830 UA 1545 N14228 EWR IAH <NA> > 2 2013 1 1 850 UA 1714 N24211 LGA IAH <NA> > 3 2013 1 1 923 AA 1141 N619AA JFK MIA American Airl~ > 4 2013 1 1 1004 B6 725 N804JB JFK BQN JetBlue Airwa~ > 5 2013 1 1 812 DL 461 N668DN LGA ATL Delta Air Lin~ > 6 2013 1 1 740 UA 1696 N39463 EWR ORD <NA> > 7 2013 1 1 913 B6 507 N516JB EWR FLL JetBlue Airwa~ > 8 2013 1 1 709 EV 5708 N829AS LGA IAD ExpressJet Ai~ > 9 2013 1 1 838 B6 79 N593JB JFK MCO JetBlue Airwa~ > 10 2013 1 1 753 AA 301 N3ALAA LGA ORD American Airl~ > # ... with 336,766 more rows ``` --- # Right join .pull-left[ * `right_join()`: **mantiene todos los registros de la segunda tabla**, y busca cuales id en la primera. ```r tabla_1 %>% right_join(tabla_2) ``` ] .pull-right[ <div class="figure" style="text-align: center"> <img src="https://dadosdelaplace.github.io/courses-ECI-2022/img/right_join.jpg" alt="Esquema del right join" width="60%" /> <p class="caption">Esquema del right join</p> </div> ] ```r tabla_1 <- tibble("key_1" = 1:5, "valor" = paste0("x", 1:5)) tabla_2 <- tibble("key_2" = 3:7, "valor" = paste0("x", 3:7)) ``` .pull-left[ ```r # Left tabla_1 %>% left_join(tabla_2, by = c("key_1" = "key_2")) ``` ``` > # A tibble: 5 x 3 > key_1 valor.x valor.y > <int> <chr> <chr> > 1 1 x1 <NA> > 2 2 x2 <NA> > 3 3 x3 x3 > 4 4 x4 x4 > 5 5 x5 x5 ``` ] .pull-right[ ```r # Left tabla_1 %>% right_join(tabla_2, by = c("key_1" = "key_2")) ``` ``` > # A tibble: 5 x 3 > key_1 valor.x valor.y > <int> <chr> <chr> > 1 3 x3 x3 > 2 4 x4 x4 > 3 5 x5 x5 > 4 6 <NA> x6 > 5 7 <NA> x7 ``` ] --- # Claves y sufijos * `by = c("var_t1" = "var_t2")`: le indicaremos en qué columna de cada tabla están las claves por las que vamos a cruzar (si se llaman igual, basta con `by = "var_t1"`). .pull-left[ ```r # Left tabla_1 %>% left_join(tabla_2, by = c("key_1" = "key_2")) ``` ``` > # A tibble: 5 x 3 > key_1 valor.x valor.y > <int> <chr> <chr> > 1 1 x1 <NA> > 2 2 x2 <NA> > 3 3 x3 x3 > 4 4 x4 x4 > 5 5 x5 x5 ``` ] .pull-right[ ```r # Left tabla_1 %>% right_join(tabla_2, by = c("key_1" = "key_2")) ``` ``` > # A tibble: 5 x 3 > key_1 valor.x valor.y > <int> <chr> <chr> > 1 3 x3 x3 > 2 4 x4 x4 > 3 5 x5 x5 > 4 6 <NA> x6 > 5 7 <NA> x7 ``` ] Además podemos **cruzar por varias columnas a la vez** (interpretará como igual registro aquel que tenga el conjunto de claves igual), con `by = c("var1_t1" = "var1_t2", "var2_t1" = "var2_t2", ...)` --- # Claves y sufijos * `suffix`: si al unir dos tablas, hay columnas que se llaman igual, por defecto nos añade los sufijos `_x` y `_y`. ```r # Left tabla_1 %>% left_join(tabla_2, by = c("key_1" = "key_2")) ``` ``` > # A tibble: 5 x 3 > key_1 valor.x valor.y > <int> <chr> <chr> > 1 1 x1 <NA> > 2 2 x2 <NA> > 3 3 x3 x3 > 4 4 x4 x4 > 5 5 x5 x5 ``` Dicho **sufijo podemos especificárselo** en `suffix`, que nos permita distingar la variable de una tabla y de otra. ```r # Left tabla_1 %>% left_join(tabla_2, by = c("key_1" = "key_2"), suffix = c("_tab1", "_tab_2")) ``` ``` > # A tibble: 5 x 3 > key_1 valor_tab1 valor_tab_2 > <int> <chr> <chr> > 1 1 x1 <NA> > 2 2 x2 <NA> > 3 3 x3 x3 > 4 4 x4 x4 > 5 5 x5 x5 ``` ] --- # Inner, full y anti join * `inner_join()` vs `full_join()`: solo los registros comunes vs todos de ambas. ```r # Inner tabla_1 %>% inner_join(tabla_2, by = c("key_1" = "key_2"), suffix = c("_tab1", "_tab_2")) ``` ``` > # A tibble: 3 x 3 > key_1 valor_tab1 valor_tab_2 > <int> <chr> <chr> > 1 3 x3 x3 > 2 4 x4 x4 > 3 5 x5 x5 ``` ```r # full tabla_1 %>% full_join(tabla_2, by = c("key_1" = "key_2"), suffix = c("_tab1", "_tab_2")) ``` ``` > # A tibble: 7 x 3 > key_1 valor_tab1 valor_tab_2 > <int> <chr> <chr> > 1 1 x1 <NA> > 2 2 x2 <NA> > 3 3 x3 x3 > 4 4 x4 x4 > 5 5 x5 x5 > 6 6 <NA> x6 > 7 7 <NA> x7 ``` * `anti_join()`: los que están en la primera y no en la segunda. ```r # antijoin tabla_1 %>% anti_join(tabla_2, by = c("key_1" = "key_2"), suffix = c("_tabla1", "_tabla_2")) ``` ``` > # A tibble: 2 x 2 > key_1 valor > <int> <chr> > 1 1 x1 > 2 2 x2 ``` --- # Ejercicios .panelset[ .panel[.panel-name[Ejercicios] * 📝 **Ejercicio 1**: del paquete `{nycflights13}` cruza la tabla `flights` con `airlines`. Queremos mantener todos los registros de vuelo, añadiendo la información de las aerolíneas a la tabla de aviones. * 📝 **Ejercicio 2**: a la tabla obtenida del apartado anterior, cruza con los datos de los aviones en `planes`, pero incluyendo solo aquellos vuelos de los que tengamos información de sus aviones (y viceversa). * 📝 **Ejercicio 3**: repite el ejercicio anterior pero conservando ambas variables `year` (en una es el año del vuelo, en la otra es el año de construcción del avión). ] .panel[.panel-name[Sol. Ej. 1] ```r flights %>% left_join(airlines, by = "carrier") ``` ``` > # A tibble: 336,776 x 20 > year month day dep_time sched_dep_time dep_delay arr_time sched_arr_time > <int> <int> <int> <int> <int> <dbl> <int> <int> > 1 2013 1 1 517 515 2 830 819 > 2 2013 1 1 533 529 4 850 830 > 3 2013 1 1 542 540 2 923 850 > 4 2013 1 1 544 545 -1 1004 1022 > 5 2013 1 1 554 600 -6 812 837 > 6 2013 1 1 554 558 -4 740 728 > 7 2013 1 1 555 600 -5 913 854 > 8 2013 1 1 557 600 -3 709 723 > 9 2013 1 1 557 600 -3 838 846 > 10 2013 1 1 558 600 -2 753 745 > # ... with 336,766 more rows, and 12 more variables: arr_delay <dbl>, > # carrier <chr>, flight <int>, tailnum <chr>, origin <chr>, dest <chr>, > # air_time <dbl>, distance <dbl>, hour <dbl>, minute <dbl>, time_hour <dttm>, > # name <chr> ``` ] .panel[.panel-name[Sol. Ej. 2] ```r flights %>% left_join(airlines, by = "carrier") %>% inner_join(planes, by = "tailnum") ``` ``` > # A tibble: 284,170 x 28 > year.x month day dep_time sched_dep_time dep_delay arr_time sched_arr_time > <int> <int> <int> <int> <int> <dbl> <int> <int> > 1 2013 1 1 517 515 2 830 819 > 2 2013 1 1 533 529 4 850 830 > 3 2013 1 1 542 540 2 923 850 > 4 2013 1 1 544 545 -1 1004 1022 > 5 2013 1 1 554 600 -6 812 837 > 6 2013 1 1 554 558 -4 740 728 > 7 2013 1 1 555 600 -5 913 854 > 8 2013 1 1 557 600 -3 709 723 > 9 2013 1 1 557 600 -3 838 846 > 10 2013 1 1 558 600 -2 849 851 > # ... with 284,160 more rows, and 20 more variables: arr_delay <dbl>, > # carrier <chr>, flight <int>, tailnum <chr>, origin <chr>, dest <chr>, > # air_time <dbl>, distance <dbl>, hour <dbl>, minute <dbl>, time_hour <dttm>, > # name <chr>, year.y <int>, type <chr>, manufacturer <chr>, model <chr>, > # engines <int>, seats <int>, speed <int>, engine <chr> ``` ] .panel[.panel-name[Sol. Ej. 2] ```r flights %>% left_join(airlines, by = "carrier") %>% inner_join(planes, by = "tailnum", suffix = c("_vuelo", "_const")) ``` ``` > # A tibble: 284,170 x 28 > year_vuelo month day dep_time sched_dep_time dep_delay arr_time > <int> <int> <int> <int> <int> <dbl> <int> > 1 2013 1 1 517 515 2 830 > 2 2013 1 1 533 529 4 850 > 3 2013 1 1 542 540 2 923 > 4 2013 1 1 544 545 -1 1004 > 5 2013 1 1 554 600 -6 812 > 6 2013 1 1 554 558 -4 740 > 7 2013 1 1 555 600 -5 913 > 8 2013 1 1 557 600 -3 709 > 9 2013 1 1 557 600 -3 838 > 10 2013 1 1 558 600 -2 849 > # ... with 284,160 more rows, and 21 more variables: sched_arr_time <int>, > # arr_delay <dbl>, carrier <chr>, flight <int>, tailnum <chr>, origin <chr>, > # dest <chr>, air_time <dbl>, distance <dbl>, hour <dbl>, minute <dbl>, > # time_hour <dttm>, name <chr>, year_const <int>, type <chr>, > # manufacturer <chr>, model <chr>, engines <int>, seats <int>, speed <int>, > # engine <chr> ``` ] ] --- class: inverse center middle # Bloque III: Introducción al dataviz en R. .left[ ### 👉 [Dataviz: Introducción histórica.](#intro-historica) ### 👉 [Introducción a ggplot2](#intro-ggplot2) ### 👉 [La importancia de visualizar datos](#importancia-dataviz) ### 👉 [Profundizando en ggplot2](#importancia-dataviz) ] --- name: intro-historica class: center, middle # Dataviz: introducción histórica ## **La historia de los gráficos estadísticos** La aparición de gráficos estadísticos es relativamente reciente en la ciencia (hasta la Edad Media solo se acercaban a la idea los mapas) --- # Breve historia del dataviz ## Punto de partida: los mapas <img src = "https://images-na.ssl-images-amazon.com/images/I/713JiiOHrEL.jpg" alt = "atlas" align = "right" width = "230" style = "margin-top: 0.1vh;margin-right: 0.5rem;margin-left: 0.5rem;"> Podríamos considerar los **mapas** como las primeras **visualización de datos**, con la representación de nuestra realidad en **superficies bidimensionales**. Las propias palabras _chart_ y _cartography_ derivan del mismo origen latino, _charta_, así como otros términos matemáticos relacionados como contornos, niveles, curvas, escalas o dominios <sup>2</sup>, aunque el primer uso datado de coordenadas parece venir de los egipcios. <sup>3</sup> Parte de esta introducción está basada en: .footnote[[1] [«Gramática de las gráficas: pistas para mejorar las representaciones de datos» de Joaquín Sevilla](http://academica-e.unavarra.es/bitstream/handle/2454/15785/Gram%C3%A1tica.pdf) [2] [«Presentation Graphics» de Leland Wilkinson. International Encyclopedia of the Social & Behavioral Sciences](https://www.cs.uic.edu/~wilkinson/Publications/iesbs.pdf) [3] [«Quantitative Graphics in Statistics: A Brief History» de James R. Beniger y Dorothy L. Robyn. The American Statistician (1978)](https://www.jstor.org/stable/2683467)] --- # Breve historia de la estadística .pull-left[ * Del (neo)latín «statisticum collegium»: consejo de **Estado**. * Del alemán «statistik» (ciencia del **Estado**, intoducido por G. Achenwall). **Origen**: una herramienta para la **administración** eficiente del Estado, pero **sin intención de comunicar ni de convertir el dato en información**. #### Primeros usos: elaboración de censos Los **primeros usos** documentados de la estadística fueron la elaboración de **censos** por parte de **mesopotámicos, chinos y egipcios**, con tres fines: * Cobrar **impuestos** (un saludo, Willyrex). * Reparto de **tierras** y optimización de su uso. * **Reclutamiento de soldados**. ] .pull-right[ ## Estadística en la guerra Según Tucídides, conceptos estadísticos como la **moda** datan del **siglo V a.C.**: para asaltar la muralla de la ciudad de Platea, ponían a contar a varios soldados el número de ladrillos vistos en la muralla, quedándose con el **conteo más repetido (la moda, el más frecuente)**, permitiendo el cálculo de la altura de la muralla. <img src="./img/peloponeso.jpg" width="70%" style="display: block; margin: auto;" /> ] --- # ¿Qué han hecho los romanos por nosotros? .pull-left[ Precisamente por el tamaño de su Imperio, fueron los **romanos** quienes hicieron un uso más intenso de la estadística: * **Censos** (elaborados por la censura, que elaboraba no solo el censo sino la supervisión de la moralidad pública). * Primeras **tablas de natalidad/mortalidad** * Primeros **catastros** (registros oficiales de propiedades, primeros impuestos) <img src="./img/catastro.jpg" width="60%" style="display: block; margin: auto;" /> ] .pull-right[ <img src="https://www.publico.es/tremending/wp-content/uploads/2019/02/lifeofbrian3.jpg" width="95%" style="display: block; margin: auto auto auto 0;" /> ] --- # Breve historia de la estadística .pull-left[ ## **ÁRABES** Autores de los **primeros tratados de estadística**, como el manuscrito de **Al-Kindi (801-873)**, que usó la distribución de **frecuencias de palabras** para el desarrollo de métodos de cifrado y descifrado de **mensajes encriptados**. ] .pull-right[ ## **MÉXICO** Ya en el **año 1116, el rey Xólotl** implementó un **censo** que consistía en la **estimación de piedras**, tirando cada súbdito una a un montón (Nepohualco). ] .pull-left[ ## **INGLATERRA** Desde el siglo XII se realiza la **Prueba del Pyx**, considerado uno de los **primeros controles de calidad**: se extre una de las monedas acuñadas y se deposita en una caja, para un año después comprobar su calidad y pureza. ] .pull-right[ ## **ITALIA** En paralelo al **auge de los primeros «sistemas financieros» en Italia**, «La Nuova Crónica» de G. Villani fue considerado durante mucho tiempo el primer tratado de estadística (hasta el descubrimiento de los trabajos de Al-Kindi). ] --- # Navegación y astronomía Y es de aquella época medieval, en la que la navegación y la astronomía empezaban a tomar relevancia científica, cuando aparece la que se considera la primera gráfica (aunque no propiamente estadística) <sup>3</sup>, representando el **movimiento cíclico de los planetas** (entre los siglos X y XI) <div class="figure" style="text-align: center"> <img src="./img/dataviz_historico_1.png" alt="Gráfica extraída de Beniger y Robyn (1978)" width="60%" /> <p class="caption">Gráfica extraída de Beniger y Robyn (1978)</p> </div> [3] [«Quantitative Graphics in Statistics: A Brief History» de James R. Beniger y Dorothy L. Robyn. The American Statistician (1978)](https://www.jstor.org/stable/2683467) --- # Navegación y astronomía Con una motivación similar, en torno a 1360 el matemático **Nicole Oresme** diseñó el **primer gráfico de barras** (no estadístico), con la idea de **visualizar a la vez dos magnitudes físicas teóricas**. <div class="figure" style="text-align: center"> <img src="./img/dataviz_historico_2.jpeg" alt="Gráfica extraída de Friendly y Valero-Mora (2010), de «Tractatus De Latitudinibus Formarum»" width="30%" /> <p class="caption">Gráfica extraída de Friendly y Valero-Mora (2010), de «Tractatus De Latitudinibus Formarum»</p> </div> [5] [«The First (Known) Statistical Graph: Michael Florent van Langren and the 'Secret' of Longitude» de M. Friendly y P. M. Valero-Mora. The American Statistician (2010)](https://www.researchgate.net/publication/227369016_The_First_Known_Statistical_Graph_Michael_Florent_van_Langren_and_the_Secret_of_Longitude) --- # Primer gráfico estadístico La mayoría de expertos, como Tufte <sup>6,7</sup>, consideran **este gráfico** casi longitudinal como la **primera visualización de datos** de la historia, hecha por **van Langren** en 1644, representando la **distancia (en longitud) entre Toledo y Roma** (un poco mal medida ya que la distancia real es de 16.5º). <div class="figure" style="text-align: center"> <img src="./img/longitud_dataviz.jpg" alt="Gráfica original extraída de Friendly y Valero-Mora (2010)" width="45%" /> <p class="caption">Gráfica original extraída de Friendly y Valero-Mora (2010)</p> </div> <div class="figure" style="text-align: center"> <img src="./img/dataviz_historico_3.jpeg" alt="Adaptación extraída de Friendly y Valero-Mora (2010)" width="45%" /> <p class="caption">Adaptación extraída de Friendly y Valero-Mora (2010)</p> </div> [6] [«Visual explanations: images and quantities, evidence and narrative» de E. Tufte](https://archive.org/details/visualexplanatio00tuft) [7] [«PowerPoint is evil» de E. Tufte](https://www.wired.com/2003/09/ppt2/) --- # ¿Qué es una gráfica estadística? ¿Por qué ese gráfico se considera la primera visualización estadística de la historia? ¿Qué es lo que hace que una visualización sea una gráfica estadística? **¿Cuál es la frontera entre una ilustración y una gráfica (de datos)?** Esas mismas preguntas se hizo **Joaquín Sevilla** en su manual <sup>1</sup>, argumentando que deben cumplir **3 requisitos**: 1. Que se base en el esquema de composición de **eje métrico** (proceso de medida): **no cualquier dibujo que incluya números** lo podemos denominar «gráfica estadística». .pull-left[ <div class="figure" style="text-align: center"> <img src="./img/mapa_sevilla.jpg" alt="INFOGRAFÍA extraída del manual de Joaquín Sevilla" width="55%" /> <p class="caption">INFOGRAFÍA extraída del manual de Joaquín Sevilla</p> </div> ] .pull-right[ <div class="figure" style="text-align: center"> <img src="./img/grafica_dinero_sevilla.jpg" alt="GRÁFICA extraída del manual de Joaquín Sevilla" width="53%" /> <p class="caption">GRÁFICA extraída del manual de Joaquín Sevilla</p> </div> ] [1] [«Gramática de las gráficas: pistas para mejorar las representaciones de datos» de Joaquín Sevilla](http://academica-e.unavarra.es/bitstream/handle/2454/15785/Gram%C3%A1tica.pdf) --- # Vizfail .pull-left[ <div class="figure" style="text-align: center"> <img src="./img/persona_dataviz.jpg" alt="Ejemplo de metáfora visual mal ejecutada" width="99%" /> <p class="caption">Ejemplo de metáfora visual mal ejecutada</p> </div> ] .pull-right[ * La figura elegida (una persona caminando) no guarda relación alguna con lo que se pretende representar. * Los sectores señalados no tienen relación con el ítem a representar, lo que dificulta su interpretación * Los colores no dan información de ningún tipo. * La forma hace imposible la comparación entre áreas (salvo que leas el % adjunto). * La suma total supera el 100% ¿? * **Sin la fuente de los datos**. ] --- # ¿Qué es una gráfica estadística? Ese **proceso de medida** en el que una gráfica se debe apoyar lo ilustra muy bien Alberto Cairo <sup>8</sup> con esta **infografía**. * ¿Sabrías decir en 5 segundos en que región el empleo ha crecido más? ¿Y menos? * ¿Sabrías decir en 5 segundos si la variación ha sido mayor en Madrid, La Rioja o Canarias? <div class="figure" style="text-align: center"> <img src="./img/mapa_cairo_esp.jpg" alt="INFOGRAFÍA extraída de Alberto Cairo" width="65%" /> <p class="caption">INFOGRAFÍA extraída de Alberto Cairo</p> </div> [8] «The Functional Art: an introduction to information graphics and visualization» de Alberto Cairo. --- # ¿Qué es una gráfica estadística? <div class="figure" style="text-align: center"> <img src="./img/mapa_cairo_2.jpg" alt="GRÁFICA extraída de Alberto Cairo" width="90%" /> <p class="caption">GRÁFICA extraída de Alberto Cairo</p> </div> La principal diferencia entre otro tipo de comunicación visual y una **gráfica estadística** radica en proporcionar **herramientas de medida**. --- # ¿Qué es una gráfica estadística? 1. Que se base en el esquema de composición de **eje métrico** 2. Debe incluir **información cuantitativa** (debe visualizar datos) 3. La relación de representatividad debería **ser reversible**: los **datos deberían poder «recuperarse»** a partir de la gráfica (una gráfica estadística es un tipo particular de **aplicación** matemática). <div class="figure" style="text-align: center"> <img src="./img/sevilla_densidad.jpg" alt="Gráfica extraída del manual de Joaquín Sevilla" width="50%" /> <p class="caption">Gráfica extraída del manual de Joaquín Sevilla</p> </div> [1] [«Gramática de las gráficas: pistas para mejorar las representaciones de datos» de Joaquín Sevilla](http://academica-e.unavarra.es/bitstream/handle/2454/15785/Gram%C3%A1tica.pdf) --- # ¿Qué es una gráfica estadística? Hay muchas formas de hacer una gráfica estadística, y **no suele pasar** por hacer un gráfico de tartas. Los **diagramas de tartas o sectores** tiene un problema de reversibilidad: * Si hay **muchas variables**: salvo que conozcas el montante total y tengas un transportador de ángulos a mano, es muy complicado que obtengas información. * Si hay **pocas variables**: ¿aporta algo distinto (y/o mejor) que una tabla? <div class="figure" style="text-align: center"> <img src="./img/sectores_sevilla.jpg" alt="Gráfica extraída del manual de Joaquín Sevilla" width="40%" /> <p class="caption">Gráfica extraída del manual de Joaquín Sevilla</p> </div> --- # ¿Qué es una gráfica estadística? El principal problema de un **diagrama de sectores** es que la posible información está contenido en los **ángulos**, pero nuestra interpretación la realizamos a través de la **comparación de áreas** (nuestros ojos no miden bien ángulos), las cuales dependen no solo del ángulo sino del radio. Algo similar sucede con los mal llamados **gráficos tridimensionales** (son bidimensionales con perspectiva en realidad): los valores más cercanos aparecen sobredimensionados, siendo prácticamente imposible la reversibilidad. <div class="figure" style="text-align: center"> <img src="./img/sectores_3D_sevilla.jpg" alt="Gráfica extraída del manual de Joaquín Sevilla" width="70%" /> <p class="caption">Gráfica extraída del manual de Joaquín Sevilla</p> </div> --- # Vizfail Además, dado que un diagrama de sectores solo permite una **visualización relativa** de los datos, no son comparables con otros diagramas de tartas. <img src="https://pbs.twimg.com/media/BgDR4urIMAAnHmG?format=jpg&name=medium" width="78%" style="display: block; margin: auto;" /> Gráfica extraída de <https://twitter.com/Dave_Andrade/status/432576336872624129?t=FGjLtxE8V1BJR_QdXdtZgQ&s=19> --- # Historia de la estadística: navegación y astronomía .pull-left[ ### T. Brahe Uno de los primeros usos «modernos» de la estadística fue en la **navegación y la astronomía**, siendo Tycho Brahe de los primeros en utilizar la estadística para **reducir los errores** observacionales. ] .pull-right[ ### E. Wright Fue el primero en usar en 1599 lo que hoy llamamos **mediana** en su libro «Certaine errors in navigation», aplicada a la navegación. ] .pull-left[ ### G. Galileo Aunque la fama se la llevó **Gauss**, fue el primero en plantear una idea similar a la que hoy llamamos **método de mínimos cuadrados**: los valores más probables serían aquellos que minimizaran los errores. ] .pull-right[ ### C. F. Gauss y A. M. Legendre El **método de los mínimos cuadrados**, en el que basan modelos actuales como la regresión, fue desarrollado por **Legendre y Gauss** (el último lo aplicó a la detección más probable del planeta enano Ceres). ] --- # Historia de la estadística: demografía, epidemiología y fisiología .pull-left[ #### J. Graunt Autor de «Natural and Political Observations Made upon the Bills of Mortality» (1662), uno de los primeros trabajos en los que ya se hablaba de **exceso de mortalidad** a partir de las primeras tablas de natalidad y mortalidad, **estimando la población de Londres**. ] .pull-right[ #### G. Neumann Las **fakes news** ya existían en el siglo XVII: Gaspar Neumann también un precursor en el **análisis estadístico de tablas de mortalidad**, para desmentir bulos (ejemplo: desmontó la creencia de que en los años acabados en siete morían más personas). ] Son precisamente las tablas de Graunt las que usó **Christiaan Huygens** (pionero en teoría de probabilidad con su «De ratiociniis in ludo aleae» en 1656) para generar la **primera gráfica de densidad** de una distribución continua, visualizando la **esperanza de vida** (en función de la edad). --- # Primer gráfico de densidad Son precisamente las tablas de Graunt las que usó **Christiaan Huygens** (pionero en teoría de probabilidad con su «De ratiociniis in ludo aleae» en 1656) para generar la **primera gráfica de densidad** de una distribución continua, visualizando la **esperanza de vida** (en función de la edad). <div class="figure" style="text-align: center"> <img src="https://omeka.lehigh.edu/files/fullsize/65fc32c11a768f1d3263a99caca28dff.jpg" alt="Primera función de densidad, extraída de https://omeka.lehigh.edu/exhibits/show/data_visualization/vital_statistics/huygen" width="50%" /> <p class="caption">Primera función de densidad, extraída de https://omeka.lehigh.edu/exhibits/show/data_visualization/vital_statistics/huygen</p> </div> --- # El gran boom: los gráficos de Playfair La figura que cambió el dataviz fue, sin lugar a dudas, el economista y político **William Playfair (1759-1823)**. En 1786 publicó el **«Atlas político y comercial»** con 44 gráficas (43 series temporales y el **diagrama de barras más famoso**, aunque no el primero). .pull-left[ <div class="figure" style="text-align: center"> <img src="./img/playfair_1.jpg" alt="Gráficas de Playfair, extraídas de Funkhouser y Walker (1935)" width="85%" /> <p class="caption">Gráficas de Playfair, extraídas de Funkhouser y Walker (1935)</p> </div> ] .pull-right[ <div class="figure" style="text-align: center"> <img src="./img/playfair_2.jpg" alt="Gráficas de Playfair, extraídas de Funkhouser y Walker (1935)" width="35%" /> <p class="caption">Gráficas de Playfair, extraídas de Funkhouser y Walker (1935)</p> </div> ] [10] [«Atlas político y comercial» de William Playfair (1786)](https://www.amazon.es/Playfairs-Commercial-Political-Statistical-Breviary/dp/0521855543) [11] [«Playfair and his charts» de H. Gray Funkhouser and Helen M. Walker (1935)](https://www.jstor.org/stable/45366440) --- # El gran boom: los gráficos de Playfair Playfair no solo fue el primero en usar el dataviz para entender (y no solo describir) la realidad: fue el primero en usar **conceptos modernos** como el _grid_ o el color como un elemento no solo estético. .pull-left[ <div class="figure" style="text-align: center"> <img src="./img/playfair_3.jpg" alt="Gráficas de Playfair, extraídas de https://friendly.github.io/HistDataVis" width="99%" /> <p class="caption">Gráficas de Playfair, extraídas de https://friendly.github.io/HistDataVis</p> </div> ] .pull-right[ <div class="figure" style="text-align: center"> <img src="./img/playfair_4.png" alt="Gráficas de Playfair, extraídas de la wikipedia." width="99%" /> <p class="caption">Gráficas de Playfair, extraídas de la wikipedia.</p> </div> ] [10] [«Atlas político y comercial» de William Playfair (1786)](https://www.amazon.es/Playfairs-Commercial-Political-Statistical-Breviary/dp/0521855543) --- # Primer gráfico de barras Playfair es además el **autor del gráfico de barras más famoso** (aunque no fue el primero, pero sí el que sentó un precedente, quien lo hizo _mainstream_). .pull-left[ <div class="figure" style="text-align: center"> <img src="./img/playfair_5.jpg" alt="Gráficas de Playfair de importaciones (barras grises) y exportaciones (negras) de Escocia en 1781, extraídas de la wikipedia." width="95%" /> <p class="caption">Gráficas de Playfair de importaciones (barras grises) y exportaciones (negras) de Escocia en 1781, extraídas de la wikipedia.</p> </div> ] .pull-right[ <div class="figure" style="text-align: center"> <img src="./img/playfair_6.jpg" alt="Primer diagrama de barras (Philippe Buache y Guillaume de L’Isle), visualizando los niveles del Sena desde 1732 hasta 1766, extraída de https://friendly.github.io/HistDataVis" width="95%" /> <p class="caption">Primer diagrama de barras (Philippe Buache y Guillaume de L’Isle), visualizando los niveles del Sena desde 1732 hasta 1766, extraída de https://friendly.github.io/HistDataVis</p> </div> ] [10] [«Atlas político y comercial» de William Playfair (1786)](https://www.amazon.es/Playfairs-Commercial-Political-Statistical-Breviary/dp/0521855543) --- # El gran boom: los gráficos de Playfair Playfair además fue el primero en **combinar gráficos en la misma visualización** .pull-left[ <div class="figure" style="text-align: center"> <img src="./img/playfair_7.jpg" alt="Gráfica de Playfair, visualizando 3 series temporales: precios (barras)f a quarter of wheat (the histogram bars), salarios (línea) y time-line con reinados, extraída de https://friendly.github.io/HistDataVis." width="92%" /> <p class="caption">Gráfica de Playfair, visualizando 3 series temporales: precios (barras)f a quarter of wheat (the histogram bars), salarios (línea) y time-line con reinados, extraída de https://friendly.github.io/HistDataVis.</p> </div> ] .pull-right[ <div class="figure" style="text-align: center"> <img src="./img/playfair_8.jpg" alt="Gráfica de Playfair, visualizando un time-line histórico, extraída de https://friendly.github.io/HistDataVis." width="92%" /> <p class="caption">Gráfica de Playfair, visualizando un time-line histórico, extraída de https://friendly.github.io/HistDataVis.</p> </div> ] [12] [«A Letter on Our Agricultural Distresses, Their Causes and Remedies» de William Playfair (1821)](http://onlinebooks.library.upenn.edu/webbin/book/lookupid?key=ha009726110) [13] [«An Inquiry Into the Permanent Causes of the Decline and Fall of Powerful and Wealthy Nations» de William Playfair (1821)](https://www.amazon.com/Permanent-Powerful-Nations-Prosperity-Prolonged/dp/140691830X) --- # Gráficos combinados en mapas **Minard** fue además el primero en 1858 que se atrevió a **incorporar gráficas en mapas**, mostrando la proporción de consumo de carne (ternera, cerdo, cordero) proporcionado por el mercado central de París. <div class="figure" style="text-align: center"> <img src="./img/minard.png" alt="Mapa con diagramas de sectores de Minard, extraída de https://friendly.github.io/HistDataVis." width="43%" /> <p class="caption">Mapa con diagramas de sectores de Minard, extraída de https://friendly.github.io/HistDataVis.</p> </div> --- # Mapas figurativos de Minard Minard fue el autor del famoso «Carte figurative des pertes successives en hommes de l'Armée Française dans la campagne de Russie 1812-1813», según Tufte «el mejor gráfico estadístico jamás dibujado», publicado en 1869 sobre la **desastrosa campaña rusa de las tropas de Napoleón en 1812**, mostrando 3 variables en un gráfico bidimensional <div class="figure" style="text-align: center"> <img src="./img/minard_2.png" alt="Gráfico de Minard sobre el avance de las tropas de Napoleón hacia Rusia, extraída de https://friendly.github.io/HistDataVis." width="73%" /> <p class="caption">Gráfico de Minard sobre el avance de las tropas de Napoleón hacia Rusia, extraída de https://friendly.github.io/HistDataVis.</p> </div> --- # Primera función de distribución Años antes, en 1821, el **matemático J. B. J. Fourier** publicó el primer **gráfico de una distribución acumulada** (la población de Paris en 1817 por grupos de edad), aunque nacieron para ser leídas al revés de lo usual actualmente (de derecha a izquierda). <div class="figure" style="text-align: center"> <img src="./img/fourier_dataviz.jpg" alt="Primera función de distribución acumulada, extraída de Beniger y Robyn (1978)." width="57%" /> <p class="caption">Primera función de distribución acumulada, extraída de Beniger y Robyn (1978).</p> </div> --- # Primer diagrama de dispersión Según Sevilla <sup>1</sup>, se considera al astrónomo británico **John Frederick William Herschel** el primero que publicó un **diagrama de dispersión o scatterplot** en 1833, visualizando el movimiento de la estrella doble Virginis (tiempo en el eje horizontal, posición angular en el eje vertical) <div class="figure" style="text-align: center"> <img src="./img/herschel.jpg" alt="Primer scatterplot, extraído de https://friendly.github.io/HistDataVis." width="36%" /> <p class="caption">Primer scatterplot, extraído de https://friendly.github.io/HistDataVis.</p> </div> [15] [«On the investigation of the orbits of revolving double stars: being a supplement to a paper entitled micrometrical measures of 364 double stars», de John F. W. Herschel (1833)](https://adsabs.harvard.edu/full/1833MmRAS...5..171H) --- # Primera pirámide poblacional La **primera pirámide de población** (en realidad es un doble histograma de población, en función de dos variables, edad y sexo), fue publicada por **Francis Amasa Walker**, superintendente del censo de EE.UU., en 1874. <div class="figure" style="text-align: center"> <img src="./img/walker_piramide.jpg" alt="Primera pirámide de población, extraída de https://www.depauw.edu/learn/dew/wpaper/workingpapers/DePauw2016-02-Barreto-DemographyEconomics.pdf" width="55%" /> <p class="caption">Primera pirámide de población, extraída de https://www.depauw.edu/learn/dew/wpaper/workingpapers/DePauw2016-02-Barreto-DemographyEconomics.pdf</p> </div> --- # Primer gráfico de mosaico Walker también fue el autor del **primera gráfico de mosaico** en 1874, visualizando la población de Missouri por origen racial, según el censo de 1870 <div class="figure" style="text-align: center"> <img src="./img/walker_mosaico.jpg" alt="Primer gráfico de mosaico, extraído de https://friendly.github.io/HistDataVis." width="75%" /> <p class="caption">Primer gráfico de mosaico, extraído de https://friendly.github.io/HistDataVis.</p> </div> --- # El boom de la estadística: epidemiología y bioestadística .pull-left[ #### F. Galton Primo de Charles Darwin, inventor de los **silbatos para perretes**, de los mapas de predicción meteorológica y la persona que acuñó el concepto de **regresión** (y el de eugenesia :/). <img src="https://www.bogleheads.org/w/images/thumb/9/95/Screen_Shot_2012-01-03_at_7.36.29_AM.png/600px-Screen_Shot_2012-01-03_at_7.36.29_AM.png" width="93%" style="display: block; margin: auto;" /> ] .pull-right[ <img src="./img/galton_1.jpg" width="58%" style="display: block; margin: auto;" /> <img src="./img/galton_2.png" width="58%" style="display: block; margin: auto;" /> ] --- # El boom de la estadística: epidemiología y bioestadística .pull-left[ #### John Snow Se le considera uno de los pioneros de la **epidemiología moderna** y la **estadística espacial**: aunque los **diagramas de Voronoi** tardarían años en ser formalizados, John Snow aplicó el mismo concepto para mitigar la **epidemia de cólera en Londres**, con su **mapa con diagrama de barras**, localizando el foco en la conocida fuente de Broad Street. ] .pull-right[ <img src="https://media.revistagq.com/photos/5cc84a91c46d3a2b7435d7cf/2:3/w_1799,h_2699,c_limit/pelo%20jon%20snow.jpg" width="100%" style="display: block; margin: auto;" /> ] --- # El boom de la estadística: epidemiología y bioestadística .pull-left[ #### John Snow Se le considera uno de los pioneros de la **epidemiología moderna** y la **estadística espacial**: aunque los **diagramas de Voronoi** tardarían años en ser formalizados, John Snow aplicó el mismo concepto para mitigar la **epidemia de cólera en Londres**, con su **mapa con diagrama de barras**, localizando el foco en la conocida fuente de Broad Street. [📚 «El mapa fantasma», Steven Johnson, sobre la historia de John Snow](https://capitanswing.com/libros/el-mapa-fantasma/) ] .pull-right[ <div class="figure" style="text-align: center"> <img src="https://s1.eestatic.com/2016/04/22/reportajes/reportajes_119248513_3987143_854x640.jpg" alt="John Snow, el epidemiólogo" width="100%" /> <p class="caption">John Snow, el epidemiólogo</p> </div> ] --- # Nacimiento de la estadística espacial <div class="figure" style="text-align: center"> <img src="./img/snow_mapa.jpg" alt="Mapa de Londres, mostrando los casos de cólera del 19 de agosto al 30 de septiembre de 1854, extraído de https://friendly.github.io/HistDataVis." width="77%" /> <p class="caption">Mapa de Londres, mostrando los casos de cólera del 19 de agosto al 30 de septiembre de 1854, extraído de https://friendly.github.io/HistDataVis.</p> </div> --- # Cólera en Londres Esa **epidemia de cólera** en Londres fue una catástrofe en términos humanos pero supuso un verdadero auge de la **bioestadística y visualización de datos**. Unos años antes que Snow, **William Farr** ya usaba el dataviz para **monitorizar las muertes diarias** de cólera (abajo, cólera en azul, diarre en amarillo), en función del tiempo meteorológico (arriba) <img src="./img/farr_colera.png" width="48%" style="display: block; margin: auto;" /> [16] [«General Register Office, Report on the Mortality of Cholera in England, 1848–49» publicado en 1852](https://wellcomecollection.org/works/pajtrpez/items?canvas=9) --- # Primer diagrama de área polar Fue también Farr al que se le atribuye el primer **diagrama de área polar o radial**, cambiando por completo el **paradigma cartesiano** de la época: para visualizar la estacionalidad de la mortalidad en función de la temperatura, Farr decidió visualizarlo con coordenadas polares o esféricas. <img src="./img/farr_colera_2.png" width="48%" style="display: block; margin: auto;" /> [16] [«General Register Office, Report on the Mortality of Cholera in England, 1848–49» publicado en 1852](https://wellcomecollection.org/works/pajtrpez/items?canvas=9) --- # Florence Nigthingale: una revolución .pull-left[ * El 21 de octubre de 1854 **Florence Nigthingale** fue enviada para mejorar las **condiciones sanitarias** de los **soldados británicos en la guerra de Crimea**. * A su regreso se dedicó a demostrar que los **soldados fallecían por las condiciones sanitarias**: eran **muertes evitables**. Nigthingale es la creadora del famoso **diagrama de rosa**, permitiendo pintar tres variables a la vez y su estacionalidad. * El 8 de febrero de 1955, The Times la describió como la **«ángel guardián» de los hospitales**, y al finalizar la contienda, fue recibida como una heroína, conocida como **«The Lady with the Lamp»** tras un poema de H. W. Longfellow publicado en 1857. * Años después se convirtió en la **primera mujer en la Royal Statistical Society** y renunció a su puesto para crear las primeras escuelas de enfermería ] .pull-right[ <img src="https://www.researchgate.net/profile/Miguel-Guevara-4/publication/325622727/figure/fig1/AS:635977265582080@1528640204506/FIGURA-2-La-dama-con-la-lampara-1891-en-ingles-The-lady-with-the-lamp.png" width="90%" style="display: block; margin: auto;" /> ] --- # Los gráficos en espiral no son novedosos **Florence Nigthingale** es la creadora del famoso **diagrama de rosa**, permitiendo pintar tres variables a la vez y su estacionalidad: **tiempo** (cada **gajo** es un mes), **nº de muertes** (**área** del gajo) y **causa** de la muerte (**color** del gajo: azules enfermedades infecciosas, rojas por heridas, negras otras causas). <img src="./img/rosa_nightingale.jpg" width="75%" style="display: block; margin: auto;" /> --- # La espiral de la polémica .pull-left[ <div class="figure" style="text-align: center"> <img src="https://www.memo.com.ar/files/image/30/30306/61e162fc784b3_715_896!.jpg?s=8418d87978ab79e1a76da127f6b37bb2&d=1642160894" alt="Gráfica en espiral del New York Times" width="100%" /> <p class="caption">Gráfica en espiral del New York Times</p> </div> ] .pull-right[ **¿A favor? ¿En contra?** ✖️ No es novedosa (la novedad tampoco debería ser un argumento a favor per se) ✔️ Las **coordenadas polares** son un **acierto**: los datos estacionales como los datos epidemiológicos o las temperaturas son precisamente los casos en los que está más justificado representar los datos en coordenadas polares o en espiral. ✖️ **No respeta la reversibilidad**: es más una ilustración que una gráfica estadística. La forma de llevarlo a cabo **infraestima los datos** actuales y, por su **distorsión de la realidad**, no permite **medir** precisamente el patrón estacional que se propone visualizar. ✔️ **Metáfora visual** de la pandemia. ✖️ Opinión personal: buena idea, buena como ilustración, error como gráfica estadística en su ejecución. ] --- # La espiral de la polémica .pull-left[ <div class="figure" style="text-align: center"> <img src="https://www.memo.com.ar/files/image/30/30306/61e162fc784b3_715_896!.jpg?s=8418d87978ab79e1a76da127f6b37bb2&d=1642160894" alt="Gráfica en espiral del New York Times" width="60%" /> <p class="caption">Gráfica en espiral del New York Times</p> </div> ] .pull-right[ <div class="figure" style="text-align: center"> <img src="https://elartedeldato.com/blog/the-new-york-times-spiral-graph-en-r/featured-the_nyt_spiral_hu8f42fa4590b2c04399b29318317fd771_501398_720x0_resize_lanczos_3.png" alt="Mejora de la espiral realizada por Paula Casado https://elartedeldato.com/blog/the-new-york-times-spiral-graph-en-r/" width="100%" /> <p class="caption">Mejora de la espiral realizada por Paula Casado https://elartedeldato.com/blog/the-new-york-times-spiral-graph-en-r/</p> </div> ] ✖️ El **eje que recorre la espiral no aporta**, distrae de los datos a representar, **aumenta la distorsión producida** por la no linealidad de la espiral y duplica información, dificultando la comparación estacional. --- # La espiral de la polémica <div class="figure" style="text-align: center"> <img src="https://pbs.twimg.com/media/FIgzfW4XMAsWYMs?format=jpg&name=large" alt="Misma idea, distinta ejecución, de https://twitter.com/abmakulec/status/1479496579040034822" width="90%" /> <p class="caption">Misma idea, distinta ejecución, de https://twitter.com/abmakulec/status/1479496579040034822</p> </div> --- # La espiral de la polémica .pull-left[ <div class="figure" style="text-align: center"> <img src="https://pbs.twimg.com/media/FIg4FiIXIAITUq6?format=jpg&name=medium" alt="Mejora de https://twitter.com/abmakulec/status/1479496581439238152" width="100%" /> <p class="caption">Mejora de https://twitter.com/abmakulec/status/1479496581439238152</p> </div> ] .pull-right[ ✖️ La **inclusión de colores** codificando los datos hubiese mejorado el gráfico, máxime cuando no se añade ningún tipo de escala al gráfico (aunque mida la estacionalidad, para visualizar algo estacional es fundamental que precisamente los periodos puedan ser comparables para percibir ese patrón periódico) ] El **uso de colores** no es solo algo estético: es una variable más, y la paleta elegida así como su uso es fundamental (por ejemplo, la inclusión de colores nos permite centrarnos en los oscuros, y comparar entre tonalidades). --- # ¿Líneas rojas? * Entender el contexto y respetar el dato <div class="figure" style="text-align: center"> <img src="https://pbs.twimg.com/media/FIgZcF4X0AAzLnC?format=jpg&name=medium" alt="Infografía extraída de https://twitter.com/storywithdata" width="60%" /> <p class="caption">Infografía extraída de https://twitter.com/storywithdata</p> </div> (en lo de los diagramas de tartas discrepamos :P) --- # Contexto Una **buena idea** puede estar mejor o peor ejecutada, y la forma de llevarla a cabo es importante <div class="figure" style="text-align: center"> <img src="./img/semaforo.jpg" alt="Un semáforo no es una mala idea, la forma de instalarlo sí puede serlo. Imagen extraída del manual de Joaquín Sevilla" width="30%" /> <p class="caption">Un semáforo no es una mala idea, la forma de instalarlo sí puede serlo. Imagen extraída del manual de Joaquín Sevilla</p> </div> --- # Fundamentos de la visualización de datos estadísticos ## **Recomendaciones** [«The Functional Art: an introduction to information graphics and visualization» de Alberto Cairo](https://www.amazon.es/Functional-Art-Voices-That-Matter/dp/0321834739) [«Gramática de las gráficas: pistas para mejorar las representaciones de datos» de Joaquín Sevilla](https://academica-e.unavarra.es/bitstream/handle/2454/15785/Gram%C3%A1tica.pdf) [«A Brief History of Visualization» de Friendly et al. (2008)](https://www.researchgate.net/publication/226400313_A_Brief_History_of_Data_Visualization) [«Quantitative Graphics in Statistics: A Brief History» de James R. Beniger y Dorothy L. Robyn. The American Statistician (1978)](https://www.jstor.org/stable/2683467)] [«Presentation Graphics» de Leland Wilkinson. International Encyclopedia of the Social & Behavioral Sciences](https://www.cs.uic.edu/~wilkinson/Publications/iesbs.pdf) [«The Grammar of Graphics» de Leland Wilkinson](https://www.amazon.es/Grammar-Graphics-Statistics-Computing/dp/0387245448) [«The Minard System: The Graphical Works of Charles-Joseph Minard» de Sandra Rendgen](https://www.amazon.es/gp/product/1616896337/ref=sw_img_1?smid=A1AT7YVPFBWXBL&psc=1) [«The Visual Display of Quantitative Information» de E. W. Tufte](https://www.amazon.es/Visual-Display-Quantitative-Information/dp/0961392142) --- name: intro-ggplot2 class: center, middle # Dataviz e introducción a ggplot2 ## **La gramática de los gráficos** El paquete `{ggplot2}` se basa en la idea propuesta en «Grammar of graphics» del recién fallecido Wilkinson. --- ## ggplot2: grammar of graphics (gg) .pull-left[ <div class="figure" style="text-align: center"> <img src="https://dadosdelaplace.github.io/courses-ECI-2022/img/telling-dataviz" alt="Imagen extraída de Reddit" width="70%" /> <p class="caption">Imagen extraída de Reddit</p> </div> ] .pull-right[ Una de las **principales fortalezas** de `R` no solo es el manejo de datos con `{tidyverse}`, también la visualización con uno de sus paquetes: el paquete `{ggplot2}`. La **visualización de datos** debería ser una parte fundamental de todo análisis de datos. No es solo una cuestión estética. La visualización de datos es fundamental para convertir el dato en información. ] --- ## ggplot2: grammar of graphics (gg) .pull-left[ La idea de la filosofía detrás de `{ggplot2}` es entender los **gráficos como parte integrada del flujo de datos**, dotándoles de una **gramática**, basándose en la idea de [«The Grammar of Graphics» de Leland Wilkinson](https://www.amazon.es/Grammar-Graphics-Statistics-Computing/dp/0387245448). El objetivo es empezar con un lienzo en blanco e ir **añadiendo capas a tu gráfico**, como harías por ejemplo en Photoshop, con la diferencia de que nuestras capas podemos **ligarlas al conjunto de datos**. La ventaja de `{ggplot2}` es poder **mapear atributos estéticos** (color, forma, tamaño) de **objetos geométricos** (puntos, barras, líneas) en **función de los datos**, añadiendo transformaciones de los datos, resúmenes estadísticos y transformaciones de las coordenadas. La **documentación** del paquete puedes consultarla en <https://ggplot2-book.org/introduction.html> ] .pull-right[ <div class="figure" style="text-align: center"> <img src="https://dadosdelaplace.github.io/courses-ECI-2022/img/grammar.jpg" alt="Idea detrás de la «Grammar of graphics» de Wilkinson" width="100%" /> <p class="caption">Idea detrás de la «Grammar of graphics» de Wilkinson</p> </div> ] --- ## ggplot2: grammar of graphics (gg) .pull-left[ <div class="figure" style="text-align: center"> <img src="https://dadosdelaplace.github.io/courses-ECI-2022/img/grammar.jpg" alt="Idea detrás de la «Grammar of graphics» de Wilkinson" width="100%" /> <p class="caption">Idea detrás de la «Grammar of graphics» de Wilkinson</p> </div> ] .pull-right[ Un gráfico se podrá componer de las siguientes **capas** * **Datos** * **Mapeado de elementos estéticos (aesthetics)**: ejes, color, forma, tamaño, etc (en función de los datos) * **Elementos geométricos (geom)**: puntos, líneas, barras, polígonos, etc. * **Componer gráficas (facet)**: visualizar varias gráficas a la vez. * **Transformaciones estadísticas (stat)**: ordenar, resumir, agrupar, etc. * **Sistema de coordenadas (coord)**: coordenadas, grids, etc. * **Temas (theme)**: fuente, tamaño de letra, subtítulos, captions, leyenda, ejes, etc. ] --- # Primer intento: scatter plot Veamos un **primer intento** para entender la filosofía. Imagina que queremos dibujar un **scatter plot** o **diagrama de (dispersión) de puntos**. Para ello vamos a usar el **conjunto de datos** `gapminder`, del paquete homónimo: un fichero con **datos de esperanzas de vida, poblaciones y renta per cápita** de distintos países en distintos momentos temporales. ```r library(gapminder) gapminder ``` ``` > # A tibble: 1,704 x 6 > country continent year lifeExp pop gdpPercap > <fct> <fct> <int> <dbl> <int> <dbl> > 1 Afghanistan Asia 1952 28.8 8425333 779. > 2 Afghanistan Asia 1957 30.3 9240934 821. > 3 Afghanistan Asia 1962 32.0 10267083 853. > 4 Afghanistan Asia 1967 34.0 11537966 836. > 5 Afghanistan Asia 1972 36.1 13079460 740. > 6 Afghanistan Asia 1977 38.4 14880372 786. > 7 Afghanistan Asia 1982 39.9 12881816 978. > 8 Afghanistan Asia 1987 40.8 13867957 852. > 9 Afghanistan Asia 1992 41.7 16317921 649. > 10 Afghanistan Asia 1997 41.8 22227415 635. > # ... with 1,694 more rows ``` --- # Primer intento: scatter plot El fichero consta de 1704 registros y 6 variables: `country`, `continent`, `year`, `lifeExp` (esperanza de vida), `pop` (población) y `gdpPercap` (renta per cápita). ```r glimpse(gapminder) ``` ``` > Rows: 1,704 > Columns: 6 > $ country <fct> "Afghanistan", "Afghanistan", "Afghanistan", "Afghanistan", ~ > $ continent <fct> Asia, Asia, Asia, Asia, Asia, Asia, Asia, Asia, Asia, Asia, ~ > $ year <int> 1952, 1957, 1962, 1967, 1972, 1977, 1982, 1987, 1992, 1997, ~ > $ lifeExp <dbl> 28.801, 30.332, 31.997, 34.020, 36.088, 38.438, 39.854, 40.8~ > $ pop <int> 8425333, 9240934, 10267083, 11537966, 13079460, 14880372, 12~ > $ gdpPercap <dbl> 779.4453, 820.8530, 853.1007, 836.1971, 739.9811, 786.1134, ~ ``` Para empezar con algo sencillo **filtraremos solo los datos de 1997** -- ```r gapminder_1997 <- gapminder %>% filter(year == 1997) gapminder_1997 ``` ``` > # A tibble: 142 x 6 > country continent year lifeExp pop gdpPercap > <fct> <fct> <int> <dbl> <int> <dbl> > 1 Afghanistan Asia 1997 41.8 22227415 635. > 2 Albania Europe 1997 73.0 3428038 3193. > 3 Algeria Africa 1997 69.2 29072015 4797. > 4 Angola Africa 1997 41.0 9875024 2277. > 5 Argentina Americas 1997 73.3 36203463 10967. > 6 Australia Oceania 1997 78.8 18565243 26998. > 7 Austria Europe 1997 77.5 8069876 29096. > 8 Bahrain Asia 1997 73.9 598561 20292. > 9 Bangladesh Asia 1997 59.4 123315288 973. > 10 Belgium Europe 1997 77.5 10199787 27561. > # ... with 132 more rows ``` --- # Primer intento: scatter plot .pull-left[ Vamos a realizar un **diagrama de puntos**: * **Eje X**: renta per cápita (variable `gdpPercap`) * **Eje Y**: población (variable `pop`) **¿Qué necesitamos?** * **Datos**: el conjunto filtrado `gapminder_1997`. * **Mapeado**: indicarle dentro de `aes()` (aesthetics) las variables a pintar en cada coordenada. Todo lo que esté **dentro de aes() dependerá de los datos** (en este caso `aes(x = gdpPercap, y = pop)`). ```r *ggplot(gapminder_1997, aes(x = gdpPercap, y = pop)) ``` * **Elegir una geometría**: optaremos por **puntos** con `geom_point()`. ```r ggplot(gapminder_1997, aes(x = gdpPercap, y = pop)) + geom_point() #<< Geometría ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-338-1.png" width="100%" /> ] --- # Primer intento: scatter plot .pull-left[ Vamos a profundizar en ese mapeado, cambiando el rol de los ejes: * **Eje X**: población (variable `pop`) * **Eje Y**: renta per cápita (variable `gdpPercap`) ```r ggplot(gapminder_1997, * aes(y = gdpPercap, x = pop)) + geom_point() ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-340-1.png" width="100%" /> ] --- # Primer intento: scatter plot .pull-left[ La idea podemos repetirla enfrentando ahora la **esperanza de vida** frente a **la renta per cápita**. * **Eje X**: esperanza de vida (variable `lifeExp`) * **Eje Y**: renta per cápita (variable `gdpPercap`) ```r ggplot(gapminder_1997, * aes(y = gdpPercap, x = lifeExp)) + geom_point() ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-342-1.png" width="100%" /> ] --- # Colores, tamaños y formas (constantes) .pull-left[ Para **cambiar el color de los puntos**, indicaremos dentro de `geom_point()` el color de la geometría con `color = ...` (en este caso, el color del punto). Empezaremos por un **color fijo**, por ejemplo `"red"` (existen otros como `"blue"`, `"black"`, `"yellow"`, etc) ```r # Color con palabra reservada ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp)) + * geom_point(color = "red") ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-344-1.png" width="100%" /> ] --- # Colores, tamaños y formas (constantes) .pull-left[ Los colores también podemos asignárselos por su **código hexadecimal**, consultando en la página <https://htmlcolorcodes.com/es/>, eligiendo el color que queramos. El código hexadecimal siempre comenzará con `#` ```r # Color en hexadecimal # https://htmlcolorcodes.com/es/ ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp)) + * geom_point(color = "#2EA2D8") ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-346-1.png" width="100%" /> ] --- # Colores, tamaños y formas (constantes) .pull-left[ De la misma manera podemos **indicarle el tamaño de la geometría** (en este caso el **tamaño de los punto**) con `size = ...` (cuanto mayor sea el número, mayor será el tamaño de la geometría). ```r # Color y tamaño ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp)) + * geom_point(color = "#A02B85", size = 5) ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-348-1.png" width="100%" /> ] --- # Colores, tamaños y formas (constantes) .pull-left[ También podemos jugar con la **transparencia del color** con `alpha = ...`: si `alpha = 1`, el color será totalmente opaco (por defecto); si `alpha = 0` será totalmente transparente. ```r # Color, tamaño y transparencia ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp)) + geom_point(color = "#A02B85", size = 9, * alpha = 0.4) ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-350-1.png" width="100%" /> ] --- # Mapear aesthetics .pull-left[ Hasta ahora los **parámetros estéticos** se los hemos pasado fijos y **constantes**. Pero la verdadera potencia y versatilidad de `ggplot` es entender todos esos parámetros como entendemos el mapeado coordenadas: podemos **mapear los atributos estéticos** en `aes()` para que dependan de variables de los datos Por ejemplo, vamos a **asignar un color a cada dato en función de su continente**. ```r # Tamaño fijo # Color por continentes ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp, * color = continent)) + geom_point(size = 5) ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-352-1.png" width="100%" /> ] --- # Mapear aesthetics .pull-left[ Podemos combinarlo con lo que hemos hecho anteriormente: * **color** en función del **continente**. * **tamaño** en función de la **población** * **transparencia** fija del 70% ```r ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp, * color = continent, size = pop)) + geom_point(alpha = 0.7) ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-354-1.png" width="100%" /> ] --- # Mapear aesthetics .pull-left[ En lugar de jugar con el color, también podríamos añadir las variables en función de la **forma de la geometría** (en este caso la forma de los «puntos») con `shape = ...`. ```r ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp, shape = continent, size = pop)) + geom_point(alpha = 0.7) ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-356-1.png" width="100%" /> ] --- # Gráfico multidimensional en 2D .pull-left[ Reflexionemos sobre el gráfico anterior: * **color** en función del **continente**. * **tamaño** en función de la **población** * **transparencia** fija del 70% Usando los datos hemos conseguido **dibujar en un gráfico bidimensional 4 variables** (`lifeExp` y `gdpPercap` en los ejes `\((X, Y)\)`), `continent` como color y `pop` como tamaño de la geometría) con muy pocas líneas de código. ```r ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp, color = continent, size = pop)) + geom_point(alpha = 0.7) ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-358-1.png" width="100%" /> ] --- # Escala de los ejes .pull-left[ A veces nos puede ser más conveniente **representar alguna de las variables** en otras escalas, por ejemplo en **escala logarítmica** (importante indicarlo en el gráfico), lo que podemos hacer facilmente con `scale_x_log10()` y/o `scale_y_log10()`. ```r ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp, color = continent, size = pop)) + geom_point(alpha = 0.7) + # Eje Y con escala logarítmica * scale_y_log10() ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-360-1.png" width="100%" /> ] --- # Paletas de colores .pull-left[ Si no indicamos nada, `R` selecciona automáticamente una **paleta de colores**, pero podemos indicarle alguna paleta concreta de varias maneras. La primera y más inmediata es indicarle los **colores manualmente**: con `scale_color_manual` le podemos indicar un **vector de colores**. ```r pal <- c("#A02B85", "#2DE86B", "#4FB2CA", "#E8DA2D", "#E84C2D") ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp, color = continent, size = pop)) + geom_point(alpha = 0.7) + scale_y_log10() + # Escala manual de colores * scale_color_manual(values = pal) ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-362-1.png" width="100%" /> ] --- # Paletas de colores .pull-left[ Otra opción es elegir alguna de las **paletas de colores disponibles** en el paquete `{ggthemes}`: * `scale_color_economist()`: paleta de colores basada en los colores de The Economist. ```r library(ggthemes) # scale_color_economist() ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp, color = continent, size = pop)) + geom_point(alpha = 0.8) + scale_y_log10() + scale_color_economist() ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-364-1.png" width="100%" /> ] --- # Paletas de colores .pull-left[ Otra opción es elegir alguna de las **paletas de colores disponibles** en el paquete `{ggthemes}`: * `scale_color_excel()`: paleta de colores basada en los colores del Excel. ```r library(ggthemes) ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp, color = continent, size = pop)) + geom_point(alpha = 0.8) + scale_y_log10() + scale_color_excel() ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-366-1.png" width="100%" /> ] --- # Paletas de colores .pull-left[ Otra opción es elegir alguna de las **paletas de colores disponibles** en el paquete `{ggthemes}`: * `scale_color_tableau()`: paleta de colores basada en los colores de Tableau. ```r library(ggthemes) ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp, color = continent, size = pop)) + geom_point(alpha = 0.8) + scale_y_log10() + scale_color_tableau() ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-368-1.png" width="100%" /> ] --- # Paletas de colores .pull-left[ También existen **múltiples paquetes** que nos proporcionan **paletas de colores** basados en: * **películas**: paquete `{harrypotter}` (repositorio de Github `aljrico/harrypotter`) usando `scale_color_hp_d()`. ```r library(devtools) install_github(repo = "https://github.com/aljrico/harrypotter") library(harrypotter) ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp, color = continent, size = pop)) + geom_point(alpha = 0.9) + scale_y_log10() + scale_color_hp_d(option = "ravenclaw") ``` ] .pull-right[ <div class="figure" style="text-align: center"> <img src="https://raw.githubusercontent.com/aljrico/harrypotter/master/readme_raw_files/palettes/ravenclaw.png" alt="Paleta basada en la casa Ravenclaw" width="21%" /> <p class="caption">Paleta basada en la casa Ravenclaw</p> </div> <img src="index_files/figure-html/unnamed-chunk-371-1.png" width="100%" /> ] --- # Paletas de colores .pull-left[ También existen **múltiples paquetes** que nos proporcionan **paletas de colores** basados en: * **cuadros**(paquete `{MetBrewer}` descargado desde el repositorio de Github `BlakeRMills/MetBrewer`). ```r devtools::install_github("BlakeRMills/MetBrewer") library(MetBrewer) MetBrewer::met.brewer("Renoir") ``` <img src="index_files/figure-html/unnamed-chunk-373-1.png" width="60%" /> ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-374-1.png" width="100%" /> ] --- # Paletas de colores .pull-left[ También existen **múltiples paquetes** que nos proporcionan **paletas de colores** basados en: * **cuadros**(paquete `{MetBrewer}` descargado desde el repositorio de Github `BlakeRMills/MetBrewer`). ```r devtools::install_github("BlakeRMills/MetBrewer") library(MetBrewer) MetBrewer::met.brewer("Klimt") ``` <img src="index_files/figure-html/unnamed-chunk-376-1.png" width="60%" /> ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-377-1.png" width="100%" /> ] --- # Geometrías (geom) .pull-left[ Hemos jugado un poco con las formas, tamaños y colores, pero siempre ha sido un diagrama de dispersión con puntos. Al igual que hemos usado `geom_point()`, podríamos usar otras geometrías como **líneas** con `geom_line()`. ```r ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp)) + * geom_line(alpha = 0.8) + scale_y_log10() + scale_color_tableau() ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-379-1.png" width="100%" /> ] --- # Geometrías (geom) .pull-left[ Asignado los colores a la variable `continent`, automáticamente obtenemos cada curva separada por continente. ```r # Separando por continente ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp, color = continent)) + geom_line(alpha = 0.8) + scale_y_log10() + scale_color_tableau() ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-381-1.png" width="100%" /> ] --- # Geometrías (geom) .pull-left[ La **filosofía es siempre la misma**: dado que cada elemento lo podemos tratar de forma individual, pasar de un gráfico a otro es relativamente sencillo, sin más que cambiar `geom_point()` por `geom_line()`. De la misma manera podemos dibujar un diagrama de dispersión con **formas hexagonales** con `geom_hex()`. Dado que ahora nuestra geometría **tiene volumen** tendremos dos parámetros: `color` para el contorno y `fill` para el **relleno** (fíjate que también cambiamos `scale_color_tableau()` por `scale_fill_tableau()`) ```r ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp, fill = continent, size = pop)) + * geom_hex(alpha = 0.8) + scale_y_log10() + * scale_fill_tableau() ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-383-1.png" width="100%" /> ] --- # Geometrías (geom) .pull-left[ Tenemos varias funciones de este tipo, como `geom_tile()`, que nos visualiza los datos con «mosaicos» (como baldosas), o `geom_text()`, con la podemos hacer que en lugar de una forma geométrica aparezcan **textos que tengamos en alguna variable**, que la pasaremos en `aes()` por el **parámetro label** (en este caso, la variable de la que tomará los nombres será `country`). ```r ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp, color = continent, size = pop, label = country)) + * geom_text(alpha = 0.8) + scale_y_log10() + scale_color_tableau() ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-385-1.png" width="100%" /> ] --- # Componer (facet) .pull-left[ Hasta ahora hemos pintado una sola gráfica, **codificando información en colores y formas**. Pero también podemos **dividir/desagregar los gráficos (facetar) por variables**, pintando por ejemplo un **gráfico por continente**, mostrando todos los gráficos a la vez pero por separado, con `facet_wrap()`. ```r ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp)) + geom_point(alpha = 0.9) + scale_y_log10() + * facet_wrap(~ continent) ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-387-1.png" width="100%" /> ] --- # Componer (facet) .pull-left[ También le podemos pasar **argumentos opcionales** para indicarle el **número de columnas o de filas** que queremos. ```r ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp)) + geom_point(alpha = 0.9) + scale_y_log10() + * facet_wrap(~ continent, nrow = 3) ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-389-1.png" width="100%" /> ] --- # Componer (facet) .pull-left[ De esta manera podríamos incluso **visualizar el fichero de datos originales incluye hasta 5 variables** en un gráfico bidimensional: * las variables `pop` y `lifeExp` en los **ejes**. * la variable `gdpPercap` en el **tamaño**. * la variable `continent` en el **color**. * la variable `year` en la composición de `facet_wrap()`. ```r library(MetBrewer) ggplot(gapminder %>% filter(year > 1962), aes(y = lifeExp, x = pop, size = gdpPercap, color = continent)) + geom_point(alpha = 0.6) + scale_x_log10() + scale_colour_manual(values = met.brewer("Klimt")) + facet_wrap(~ year) ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-391-1.png" width="100%" /> ] --- # Componer (facet) .pull-left[ Con `facet_grid()` podemos incluso **organizar una cuadrícula en base a dos variables**, por ejemplo que haya una **fila por año** (vamos a usar la tabla original en los **años 1952, 1972, 1982 y 2002**) y una **columna por continente**. ```r ggplot(gapminder %>% filter(year %in% c(1952, 1972, 1982, 2002)), aes(y = gdpPercap, x = lifeExp)) + geom_point(alpha = 0.9) + scale_y_log10() + facet_grid(year ~ continent) ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-393-1.png" width="100%" /> ] --- # Coordenadas y tema .pull-left[ Los gráficos pueden además **personalizarse añadiendo**, por ejemplo, *títulos y subtítulos** de la gráfica con `labs()`, asignando textos a `title, subtitle y caption`. ```r ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp, color = continent, size = pop)) + geom_point(alpha = 0.8) + scale_y_log10() + scale_color_tableau() + labs(title = "EJEMPLO DE SCATTERPLOT CON GGPLOT2", subtitle = "Esperanza vida vs renta per cápita (1997)", caption = "J. Perez Liébana | Datos: gapminder") ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-395-1.png" width="100%" /> ] --- # Coordenadas y tema .pull-left[ También podemos **personalizar algunos aspectos extras**, como el **título que vamos a dar a los ejes** o el **título de las leyendas**. ```r ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp, color = continent, size = pop)) + geom_point(alpha = 0.8) + # Eje Y con escala logarítmica scale_y_log10() + scale_color_tableau() + * labs(x = "Esperanza de vida", * y = "Renta per cápita", * color = "Continente", * size = "Población", title = "EJEMPLO DE SCATTERPLOT CON GGPLOT2", subtitle = "Esperanza vida vs renta per cápita (1997)", caption = "J. Perez Liébana | Datos: gapminder") ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-397-1.png" width="100%" /> ] --- # Coordenadas y tema .pull-left[ También podemos **ocultar algún nombre de las leyendas** (o ambos) si ya es explícito de lo que se está hablando. Por ejemplo, vamos a indicarle que no queremos el nombre de la leyenda en continentes, haciendo `color = NULL` (la variable que codifica los continentes a `NULL`). ```r ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp, color = continent, size = pop)) + geom_point(alpha = 0.8) + scale_y_log10() + scale_color_tableau() + labs(x = "Esperanza de vida", y = "Renta per cápita", * color = NULL, size = "Población", title = "EJEMPLO DE SCATTERPLOT CON GGPLOT2", subtitle = "Esperanza vida vs renta per cápita (1997)", caption = "J. Perez Liébana | Datos: gapminder") ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-399-1.png" width="100%" /> ] --- # Coordenadas y tema .pull-left[ Incluso podemos **ocultar la leyenda en sí de alguna de alguna de las variables** con `guides(size = "none")` (en este caso, `size = "none"` nos elimina la leyenda que codifica el tamaño de los puntos). ```r ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp, color = continent, size = pop)) + geom_point(alpha = 0.8) + scale_y_log10() + scale_color_tableau() + * guides(size = "none") + labs(x = "Esperanza de vida", y = "Renta per cápita", color = NULL, size = "Población", title = "EJEMPLO DE SCATTERPLOT CON GGPLOT2", subtitle = "Esperanza vida vs renta per cápita (1997)", caption = "J. Perez Liébana | Datos: gapminder") ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-401-1.png" width="100%" /> ] --- # Ejercicio .panelset[ .panel[.panel-name[Ejercicios] * 📝 **Ejercicio 1**: del conjunto `starwars` filtra solo los registros que no tenga ausente las columnas `mass, height, eye_color`. Dibuja un diagrama de puntos enfrentando `x = height` en el eje X e `y = mass` en el eje Y. * 📝 **Ejercicio 2**: modifica el código anterior para asignar el tamaño en función de `mass`. * 📝 **Ejercicio 3**: modifica el código del gráfico anterior para asignar el color en función de su color de ojos guardado en `eye_color`. Antes procesa la variable para quedarte con colores reales: si hay dos colores, quédate con el primero; el color `"hazel"` pásalo a `"brown"`; los colores `"unknown"` pásalo a gris. * 📝 **Ejercicio 4**: repite el gráfico anterior localizando ese dato con un peso extremadamente elevado (outlier), elimínalo y vuelve a repetir la visualización. * 📝 **Ejercicio 5**: repite el gráfico anterior eliminando la leyenda del tamaño del punto y cambia el título de la leyenda del color de ojos a castellano. Añade además transparencia alpha = 0.6 a los puntos. * 📝 **Ejercicio 6**: repite el gráfico modificando títulos de ejes (a castellano) con título, subtítulo y caption. * 📝 **Ejercicio 7**: explicita los cortes en los ejes con `scale_x_continuous()` y `scale_y_continuous()`: el eje X de 60 a 240 (de 30 en 30 cada marca), el eje Y de 20 a 160 (de 20 en 2). ] .panel[.panel-name[Sol. 1] .pull-left[ ```r # Eliminamos NA starwars_filtro <- starwars %>% drop_na(c(mass, height, eye_color)) # Visualizamos ggplot(starwars_filtro, aes(x = height, y = mass)) + geom_point() ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-403-1.png" width="100%" /> ] ] .panel[.panel-name[Sol. 2] .pull-left[ ```r ggplot(starwars_filtro, aes(x = height, y = mass, size = mass)) + geom_point() ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-405-1.png" width="100%" /> ] ] .panel[.panel-name[Sol. 3a] ```r # Transformar colores starwars_filtro <- starwars_filtro %>% mutate(eye_color = case_when(eye_color == "blue-gray" ~ "blue", eye_color == "hazel" ~ "brown", eye_color == "unknown" ~ "gray", eye_color == "green, yellow" ~ "green", TRUE ~ eye_color)) ``` ] .panel[.panel-name[Sol. 3b] .pull-left[ ```r # Visualizamos ggplot(starwars_filtro, aes(x = height, y = mass, size = mass, color = eye_color)) + geom_point() + scale_color_manual(values = c("black", "blue", "brown", "gray", "green", "orange", "red", "white", "yellow")) ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-408-1.png" width="100%" /> ] ] .panel[.panel-name[Sol. 4a] ```r # Localizamos el valor y su nombre starwars_filtro %>% slice_max(mass, n = 5) %>% pull(name) ``` ``` > [1] "Jabba Desilijic Tiure" "Grievous" "IG-88" > [4] "Darth Vader" "Tarfful" ``` ```r # Filtramos starwars_filtro <- starwars_filtro %>% filter(name != "Jabba Desilijic Tiure") ``` ] .panel[.panel-name[Sol. 4b] .pull-left[ ```r ggplot(starwars_filtro, aes(x = height, y = mass, size = mass, color = eye_color)) + geom_point() + scale_color_manual(values = c("black", "blue", "brown", "gray", "green", "orange", "red", "white", "yellow")) ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-411-1.png" width="100%" /> ] ] .panel[.panel-name[Sol. 5] .pull-left[ ```r ggplot(starwars_filtro, aes(x = height, y = mass, size = mass, color = eye_color)) + geom_point(alpha = 0.6) + guides(size = "none") + scale_color_manual(values = c("black", "blue", "brown", "gray", "green", "orange", "red", "white", "yellow")) + labs(color = "color de ojos") ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-413-1.png" width="100%" /> ] ] .panel[.panel-name[Sol. 6] .pull-left[ ```r ggplot(starwars_filtro, aes(x = height, y = mass, size = mass, color = eye_color)) + geom_point(alpha = 0.6) + guides(size = "none") + scale_color_manual(values = c("black", "blue", "brown", "gray", "green", "orange", "red", "white", "yellow")) + labs(color = "color de ojos", x = "altura (cm)", y = "peso (kg)", title = "STARWARS", subtitle = "Diagrama de puntos altura vs peso", caption = "J. Perez Liébana | Datos: starwars") ``` ] .pull-left[ <img src="index_files/figure-html/unnamed-chunk-415-1.png" width="100%" /> ] ] .panel[.panel-name[Sol. 7] .pull-left[ ```r ggplot(starwars_filtro, aes(x = height, y = mass, size = mass, color = eye_color)) + geom_point(alpha = 0.6) + guides(size = "none") + scale_color_manual(values = c("black", "blue", "brown", "gray", "green", "orange", "red", "white", "yellow")) + scale_y_continuous(breaks = seq(20, 160, by = 20)) + scale_x_continuous(breaks = seq(60, 240, by = 30)) + labs(color = "color de ojos", x = "altura (cm)", y = "peso (kg)", title = "STARWARS", subtitle = "Diagrama de puntos altura vs peso", caption = "J. Perez Liébana | Datos: starwars") ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-417-1.png" width="100%" /> ] ] ] --- name: stats class: center, middle # Segundo intento: diagrama de barras Vamos a visualizar en un **diagrama de barras** la **variable cualitativa** `species`, calculando primero el número de personajes de cada especie con `count()` (filtrando primero los ausentes). ```r starwars %>% filter(!is.na(species)) %>% count(species) ``` ``` > # A tibble: 37 x 2 > species n > <chr> <int> > 1 Aleena 1 > 2 Besalisk 1 > 3 Cerean 1 > 4 Chagrian 1 > 5 Clawdite 1 > 6 Droid 6 > 7 Dug 1 > 8 Ewok 1 > 9 Geonosian 1 > 10 Gungan 3 > # ... with 27 more rows ``` --- # Diagrama de barras .pull-left[ Para visualizar un **diagrama de barras** usaremos `geom_col()` en lugar de `geom_point()`, con `y = species` y `x = n` (las frecuencias) ```r ggplot(starwars %>% filter(!is.na(species)) %>% count(species), aes(y = species, x = n)) + * geom_col() ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-420-1.png" width="100%" /> ] --- # Diagrama de barras .pull-left[ Podemos usar las **frecuencias** `\(n\)` también para codificar el color de las barras, añadiendo un **degradado de colores continuo**. ```r ggplot(starwars %>% filter(!is.na(species)) %>% count(species), aes(y = species, x = n, fill = n)) + geom_col() + * scale_fill_continuous_tableau() + labs(fill = "Frecuencia absoluta", x = "Número de personajes", y = "Especies") ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-422-1.png" width="100%" /> ] --- # Factores Al tener **muchas categorías** en `species` con muy pocos elementos, tenemos un **gráfico poco claro**, así que vamos a preprocesar antes la variable. En `R` las **variables cualitativas** se conoce como **factores**, y las posibles modalidades como **niveles**. El paquete `{forcats}` de `{tidyverse}` contienen multitud de herramientas para su manejo. Veamos un **ejemplo sencillo** definiendo una variable estado que tome los valores `"sano"`, `"leve"` y `"grave"`: ```r estado <- c("grave", "leve", "sano", "sano", "sano", "grave", "grave", "leve", "grave", "sano", "sano") estado ``` ``` > [1] "grave" "leve" "sano" "sano" "sano" "grave" "grave" "leve" "grave" > [10] "sano" "sano" ``` La variable `estado` actualmente es de tipo `character`. ```r class(estado) ``` ``` > [1] "character" ``` --- # Factores **¿Cómo indicarle que es cualitativa (factor?)** Haciendo uso de la función `as_factor` en `{forcats}`. ```r library(tidyverse) estado_fct <- as_factor(estado) estado_fct ``` ``` > [1] grave leve sano sano sano grave grave leve grave sano sano > Levels: grave leve sano ``` ```r class(estado_fct) ``` ``` > [1] "factor" ``` No solo ha cambiado la clase de la variable sino que ahora, debajo del valor guardado, nos aparece la frase `Levels: grave leve sano`: son las **modalidades o niveles de nuestra cualitativa**. Imagina que ese día en el hospital no tuviésemos a nadie en estado grave: aunque ese día nuestra variable no tome dicho valor, el estado grave es un nivel permitido, así que aunque lo eliminemos, el nivel permanece. ```r estado_fct[estado_fct %in% c("sano", "leve")] ``` ``` > [1] leve sano sano sano leve sano sano > Levels: grave leve sano ``` --- # Factores Si queremos indicarle que **elimine un nivel no usado** en ese momento podemos hacerlo con `fct_drop()` ```r fct_drop(estado_fct[estado_fct %in% c("sano", "leve")]) ``` ``` > [1] leve sano sano sano leve sano sano > Levels: leve sano ``` Al igual que podemos eliminar niveles podemos **ampliar los niveles** existentes (aunque no existan datos de ese nivel en ese momento) con `fct_expand()` ```r fct_expand(estado_fct, c("UCI", "fallecido")) ``` ``` > [1] grave leve sano sano sano grave grave leve grave sano sano > Levels: grave leve sano UCI fallecido ``` Para variables de tipo factor podemos contar los elementos de cada nivel de una manera sencilla con `fct_count()` (similar al `count()`). ```r fct_count(estado_fct) ``` ``` > # A tibble: 3 x 2 > f n > <fct> <int> > 1 grave 4 > 2 leve 2 > 3 sano 5 ``` --- # Factores Si te fijas el **orden de los niveles** es por **orden de aparición** en la variable, pero podemos **ordenarlos por frecuencia** con `fct_infreq()` ```r fct_infreq(estado_fct) ``` ``` > [1] grave leve sano sano sano grave grave leve grave sano sano > Levels: sano grave leve ``` A veces queremos **agrupar niveles**, por ejemplo, **no permitiendo niveles que no sucedan un mínimo de veces**. Con `fct_lump_min(estado_fct, min = 5)` le indicaremos que para que **exista el nivel debe de suceder al menos 3 veces** (las observaciones que no lo cumplan irán al nivel indicado en `other_level`; podemos hacer algo equivalente en función de su frecuencia relativa con `fct_lump_prop()`). ```r fct_lump_min(estado_fct, min = 5, other_level = "otros") ``` ``` > [1] otros otros sano sano sano otros otros otros otros sano sano > Levels: sano otros ``` --- # Diagrama de barras .pull-left[ Vamos a repetir el diagrama de barras anterior pero indicándole que nos convierta `species` a **factor**, y que nos **agrupe aquellas niveles que tengan menos de 2 personajes**. ```r ggplot(starwars %>% filter(!is.na(species)) %>% mutate(species = fct_lump_min(species, min = 3, other_level = "Otras especies")) %>% count(species), aes(y = species, x = n, fill = n)) + geom_col() + scale_fill_continuous_tableau() + labs(fill = "Frecuencia absoluta", x = "Número de personajes", y = "Especies") ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-433-1.png" width="100%" /> ] --- # Mosaicos o treemaps .pull-left[ Otra opción habitual son los **mosaicos o treemaps**, representando en una **cuadrícula cada una de las categorías**, cuya **área sea proprocional a las veces que aparece**. Lo haremos con `{treemapify}`: los parámetros dentro de `aes()` serán `area` (asociado a la frecuencia absoluta), `fill` (color del relleno) y `label` (nombre). Usaremos una de las paletas de colores de cuadros vistas, en este caso `MetBrewer::met.brewer("Renoir")`. ```r library(treemapify) library(MetBrewer) ggplot(starwars %>% drop_na(species) %>% mutate(species = fct_lump_min(species, min = 2, other_level = "otras")) %>% count(species), aes(area = n, fill = species, label = species)) + * geom_treemap() + scale_fill_manual(values = met.brewer("Renoir")) + labs(fill = "Especies") ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-435-1.png" width="100%" /> ] --- # Mosaicos o treemaps .pull-left[ Con `geom_treemap_text()` podemos además escribir el **nombre de los niveles**, pudiendo **eliminar la leyenda**. ```r library(treemapify) library(MetBrewer) ggplot(starwars %>% drop_na(species) %>% mutate(species = fct_lump_min(species, min = 2, other_level = "otras")) %>% count(species), aes(area = n, fill = species, label = species)) + geom_treemap() + geom_treemap_text(colour = "white", place = "centre", size = 17) + scale_fill_manual(values = met.brewer("Renoir")) + labs(fill = "Especies") + guides(fill = "none") ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-437-1.png" width="100%" /> ] --- # Ejercicio .panelset[ .panel[.panel-name[Ejercicios] * 📝 **Ejercicio 1**: visualiza un **diagrama de barras vertical** con `gapminder`, al que le pasaremos la variable `gdpPercap` a representar en el eje Y (la altura de las barras) y `year` en el eje X (tendremos una barra por año, con la altura del total mundial para ese año). * 📝 **Ejercicio 2**: calcula la media de renta per cápita agrupada por continente y año, y guárdalo en `gapminder_por_continente`. * 📝 **Ejercicio 3**: con `gapminder_por_continente` realiza un diagrama de barras, de forma que el color del relleno `fill` dependa del continente. * 📝 **Ejercicio 4**: repite el gráfico anterior con `position = "dodge2"` dentro de la función `geom_col()`. * 📝 **Ejercicio 5**: repite el gráfico anterio con `position = "fill"`,` que hará cada barra de igual longitud, permitiéndonos visualizar los datos en relativo. * 📝 **Ejercicio 6**: añade `coord_flip()` para un **diagrama de barras horizontales**. Dale además un título, subtítulo y caption, y nombre en leyenda. ] .panel[.panel-name[Sol. 1] .pull-left[ ```r ggplot(gapminder, aes(y = gdpPercap, x = year)) + geom_col() ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-439-1.png" width="100%" /> ] ] .panel[.panel-name[Sol. 2] ```r gapminder_por_continente <- gapminder %>% group_by(year, continent) %>% summarise(sum_gdpPercap = mean(gdpPercap)) gapminder_por_continente ``` ``` > # A tibble: 60 x 3 > # Groups: year [12] > year continent sum_gdpPercap > <int> <fct> <dbl> > 1 1952 Africa 1253. > 2 1952 Americas 4079. > 3 1952 Asia 5195. > 4 1952 Europe 5661. > 5 1952 Oceania 10298. > 6 1957 Africa 1385. > 7 1957 Americas 4616. > 8 1957 Asia 5788. > 9 1957 Europe 6963. > 10 1957 Oceania 11599. > # ... with 50 more rows ``` ] .panel[.panel-name[Sol. 3] .pull-left[ ```r ggplot(gapminder_por_continente, aes(y = sum_gdpPercap, x = year, fill = continent)) + geom_col() + scale_fill_tableau() ``` Si te fijas, por defecto nos ha construido las **barras apiladas** ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-442-1.png" width="100%" /> ] ] .panel[.panel-name[Sol. 4] .pull-left[ ```r ggplot(gapminder_por_continente, aes(y = sum_gdpPercap, x = year, fill = continent)) + geom_col(position = "dodge2") + scale_fill_tableau() ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-444-1.png" width="100%" /> ] ] .panel[.panel-name[Sol. 5] .pull-left[ ```r ggplot(gapminder_por_continente, aes(y = sum_gdpPercap, x = year, fill = continent)) + geom_col(position = "fill") + scale_fill_tableau() ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-446-1.png" width="100%" /> ] ] .panel[.panel-name[Sol. 6] .pull-left[ ```r ggplot(gapminder_por_continente, aes(y = sum_gdpPercap, x = year, fill = continent)) + geom_col() + * coord_flip() + scale_fill_tableau() + labs(x = "Renta per cápita", y = "Año", color = "Continente", title = "EJEMPLO DE DIAGRAMA DE BARRAS", subtitle = "Barras horizontales (agrupadas por continente y año)", caption = "J. Perez Liébana | Datos: gapminder") ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-448-1.png" width="100%" /> ] ] ] --- # CASO PRÁCTICO ## Visualización de datos continuos Hasta ahora hemos visualizado variables cuantitaivas discretas o cualitativas: ¿cómo visualizar datos continuos? --- # Ejemplo: percepción de la probabilidad Vamos a analizar un **conjunto de datos** interesante, que contiene las respuestas a las preguntas... **«¿Qué probabilidad (%) asignarías al término ...?** Dicha pregunta se realizó para distintos términos como «almost no chance», «about even», «probable» o «almost certainly», con el objetivo de comprender **cómo la gente percibe el vocabulario** relativo a la probabilidad. Los datos y gráficos están basados en el trabajo de [Zoni Nation](https://github.com/zonination/perceptions) y [data-to-viz.com](https://www.data-to-viz.com/story/OneNumOneCatSeveralObs.html) ```r datos <- read_csv("https://raw.githubusercontent.com/zonination/perceptions/master/probly.csv") datos ``` ``` > # A tibble: 46 x 17 > `Almost Certainly` `Highly Likely` `Very Good Chan~` Probable Likely Probably > <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> > 1 95 80 85 75 66 75 > 2 95 75 75 51 75 51 > 3 95 85 85 70 75 70 > 4 95 85 85 70 75 70 > 5 98 95 80 70 70 75 > 6 95 99 85 90 75 75 > 7 85 95 65 80 40 45 > 8 97 95 75 70 70 80 > 9 95 95 80 70 65 80 > 10 90 85 90 70 75 70 > # ... with 36 more rows, and 11 more variables: `We Believe` <dbl>, > # `Better Than Even` <dbl>, `About Even` <dbl>, `We Doubt` <dbl>, > # Improbable <dbl>, Unlikely <dbl>, `Probably Not` <dbl>, > # `Little Chance` <dbl>, `Almost No Chance` <dbl>, `Highly Unlikely` <dbl>, > # `Chances Are Slight` <dbl> ``` --- # Convirtiendo a tidy data .panelset[ .panel[.panel-name[Ejercicio] 1. Vamos a visualizar con un diagrama de barras la **media de las probabilidades** que la gente ha asignado a cada término. Pero antes debemos de **transformar el dataset a tidydata** con `pivot_longer()`, para que tenga el formato adecuado para `ggplot2()` 2. Tras la transformación a un formato adecuado para `ggplot2()`, vamos a **seleccionar los términos** `Almost No Chance`, `Chances Are Slight`, `Improbable`, `About Even`, `Probable` y `Almost Certainly`. 3. Además vamos a **reordenar los niveles de factor** de la variable `termino` con `fct_reorder()` (del paquete `{forcats}`) en función de sus probabilidades. ] .panel[.panel-name[Paso 1] ```r datos_pivot <- datos %>% pivot_longer(cols = everything(), names_to = "termino", values_to = "prob") datos_pivot ``` ``` > # A tibble: 782 x 2 > termino prob > <chr> <dbl> > 1 Almost Certainly 95 > 2 Highly Likely 80 > 3 Very Good Chance 85 > 4 Probable 75 > 5 Likely 66 > 6 Probably 75 > 7 We Believe 66 > 8 Better Than Even 55 > 9 About Even 50 > 10 We Doubt 40 > # ... with 772 more rows ``` ] .panel[.panel-name[Paso 2] ```r datos_final <- datos_pivot %>% filter(termino %in% c("Almost No Chance", "Chances Are Slight", "Improbable", "About Even", "Probable", "Almost Certainly")) datos_final ``` ``` > # A tibble: 276 x 2 > termino prob > <chr> <dbl> > 1 Almost Certainly 95 > 2 Probable 75 > 3 About Even 50 > 4 Improbable 20 > 5 Almost No Chance 5 > 6 Chances Are Slight 25 > 7 Almost Certainly 95 > 8 Probable 51 > 9 About Even 50 > 10 Improbable 49 > # ... with 266 more rows ``` ] .panel[.panel-name[Paso 3] ```r datos_final <- datos_final %>% mutate(termino = fct_reorder(termino, prob)) datos_final ``` ``` > # A tibble: 276 x 2 > termino prob > <fct> <dbl> > 1 Almost Certainly 95 > 2 Probable 75 > 3 About Even 50 > 4 Improbable 20 > 5 Almost No Chance 5 > 6 Chances Are Slight 25 > 7 Almost Certainly 95 > 8 Probable 51 > 9 About Even 50 > 10 Improbable 49 > # ... with 266 more rows ``` ] ] --- # Diagrama de barras de las medias .panelset[ .panel[.panel-name[Ejercicio] 1. Tras preprocesar los datos, calcula las medias de las probabilidades agrupadas por la variable `termino` (con `group_by()` y `summarise()`) 2. Usamos `geom_col()` para visualizar dichas medias como un diagrama de barras. ] .panel[.panel-name[Paso 1] ```r datos_resumen <- datos_final %>% group_by(termino) %>% summarise(media = mean(prob)) datos_resumen ``` ``` > # A tibble: 6 x 2 > termino media > <fct> <dbl> > 1 Almost No Chance 5.63 > 2 Chances Are Slight 14.1 > 3 Improbable 18.0 > 4 About Even 49.6 > 5 Probable 71.5 > 6 Almost Certainly 92.6 ``` ] .panel[.panel-name[Paso 2] .pull-left[ ```r ggplot(datos_resumen, aes(x = termino, y = media, fill = termino)) + geom_col() ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-455-1.png" width="100%" /> ] ] ] --- # Paleta de colores .pull-left[ Con `scale_fill_brewer()` vamos a darle una **paleta de colores divergente** al relleno, de rojo (poca probabilidad) a azul (mucha probabilidad) con `scale_fill_brewer(palette = "RdBu")`. Además definiremos un tema para los siguientes gráficos. ```r library(showtext) font_add_google(family = "Roboto", name = "Roboto") showtext_auto() theme_set(theme_minimal(base_family = "Roboto")) theme_update( plot.background = element_rect(fill = "white", color = "white"), plot.title = element_text(color = "black", face = "bold", size = 27)) ggplot(datos_resumen, aes(x = termino, y = media, fill = termino)) + geom_col(alpha = 0.8) + scale_fill_brewer(palette = "RdBu") + labs(fill = "Términos", y = "Probabilidad (%)", title = "Percepción de la probabilidad") ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-457-1.png" width="100%" /> ] ] --- # Visualizando puntos: geom_quasirandom .panelset[ .panel[.panel-name[Ejercicio] 1. Dado el conjunto sin resumir, el conjunto `datos_final`, define una cabecera de `ggplot()` con * `termino` en el eje x * `prob` en el eje y * `termino` en el color 2. La función `geom_quasirandom()` del paquete `{ggbeeswarm}` nos permite visualizar los datos en puntos, desperdigados de forma «aleatoria» a lo ancho. A la cabecera anterior añade `geom_quasirandom()` con: * tamaño igual a 3.5 * anchura igual a 0.5 (lo que se desperdigan respecto al centro los datos) * transparencia del 70% 3. Añade con `scale_color_brewer(palette = "RdBu")` una paleta de colores divergente. 4. Añade el título de la leyenda (de `color`), título y títulos de ejes, con `labs(...)`. ] .panel[.panel-name[Paso 1] ```r ggplot(datos_final, aes(x = termino, y = prob, color = termino)) ``` <img src="index_files/figure-html/unnamed-chunk-458-1.png" width="100%" /> ] .panel[.panel-name[Paso 2] .pull-left[ ```r library(ggbeeswarm) ggplot(datos_final, aes(x = termino, y = prob, color = termino)) + geom_quasirandom(size = 3.5, width = 0.5, alpha = 0.7) ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-460-1.png" width="100%" /> ] ] .panel[.panel-name[Paso 3] .pull-left[ ```r ggplot(datos_final, aes(x = termino, y = prob, color = termino)) + geom_quasirandom(size = 3.5, width = 0.5, alpha = 0.7) + scale_color_brewer(palette = "RdBu") ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-462-1.png" width="100%" /> ] ] .panel[.panel-name[Paso 4] .pull-left[ ```r ggplot(datos_final, aes(x = termino, y = prob, color = termino)) + geom_quasirandom(size = 3.5, width = 0.5, alpha = 0.7) + scale_color_brewer(palette = "RdBu") + labs(color = "Términos", x = "Términos", y = "Probabilidad (%)", title = "Percepción de la probabilidad") ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-464-1.png" width="100%" /> ] ] ] --- # Visualizando puntos: geom_quasirandom .panelset[ .panel[.panel-name[Ejercicio] Repite el gráfico con el conjunto de `starwars`: 1. Filtra los ausentes en `height` y `species`, y recategoriza la variable `species` en humanos o no humanos. 2. Tras el preprocesado visualiza con `geom_quasirandom()` la estatura (variable continua) de cada uno de los dos grupos. 3. Además indica con `scale_x_discrete()` las etiquetas de nuestras categorías (que por defecto aparecen en inglés, `FALSE` vs `TRUE`) 4. Elimina el título de las leyendas del color y el relleno. Añade además títulos para el eje X e Y, y título de la gráfica. ] .panel[.panel-name[Paso 1] ```r starwars_altura <- starwars %>% drop_na(height, species) %>% mutate(human = as_factor(species == "Human")) starwars_altura ``` ``` > # A tibble: 78 x 15 > name height mass hair_color skin_color eye_color birth_year sex gender > <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr> > 1 Luke Sk~ 172 77 blond fair blue 19 male mascu~ > 2 C-3PO 167 75 <NA> gold yellow 112 none mascu~ > 3 R2-D2 96 32 <NA> white, bl~ red 33 none mascu~ > 4 Darth V~ 202 136 none white yellow 41.9 male mascu~ > 5 Leia Or~ 150 49 brown light brown 19 fema~ femin~ > 6 Owen La~ 178 120 brown, gr~ light blue 52 male mascu~ > 7 Beru Wh~ 165 75 brown light blue 47 fema~ femin~ > 8 R5-D4 97 32 <NA> white, red red NA none mascu~ > 9 Biggs D~ 183 84 black light brown 24 male mascu~ > 10 Obi-Wan~ 182 77 auburn, w~ fair blue-gray 57 male mascu~ > # ... with 68 more rows, and 6 more variables: homeworld <chr>, species <chr>, > # films <list>, vehicles <list>, starships <list>, human <fct> ``` ] .panel[.panel-name[Paso 2] .pull-left[ ```r library(ggbeeswarm) ggplot(starwars_altura, aes(x = human, y = height, fill = human, color = human)) + geom_quasirandom(size = 4.5, width = 0.5, alpha = 0.5) ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-467-1.png" width="100%" /> ] ] .panel[.panel-name[Paso 3] .pull-left[ ```r library(ggbeeswarm) ggplot(starwars_altura, aes(x = human, y = height, fill = human, color = human)) + geom_quasirandom(size = 4.5, width = 0.5, alpha = 0.5) + scale_x_discrete(labels = c("NO", "SÍ")) ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-469-1.png" width="100%" /> ] ] .panel[.panel-name[Paso 4] .pull-left[ ```r library(ggbeeswarm) ggplot(starwars_altura, aes(x = human, y = height, fill = human, color = human)) + geom_quasirandom(size = 4.5, width = 0.5, alpha = 0.5) + scale_x_discrete(labels = c("NO", "SÍ")) + guides(color = "none", fill = "none") + labs(x = "¿Son humanos?", y = "Altura (cm)", title = "ALTURA DE LOS PERSONAJES DE STARWARS") ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-471-1.png" width="100%" /> ] ] ] --- # Gráficos de cajas y bigotes Otra opción para visualizar datos continuos son con los famosos **gráficos de cajas y bigotes** con `geom_boxplot()` .pull-left[ ```r ggplot(datos_final, aes(x = termino, y = prob, fill = termino)) + geom_boxplot(alpha = 0.8) + scale_fill_brewer(palette = "RdBu") + labs(fill = "Términos", y = "Probabilidad (%)", title = "Percepción de la probabilidad") ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-473-1.png" width="100%" /> ] --- # Gráficos de cajas y bigotes .pull-left[ <img src="index_files/figure-html/unnamed-chunk-474-1.png" width="100%" /> ] .pull-right[ Un **diagrama de caja y bigotes o box plot** es un gráfico que resume algunos datos estadísticos en relación a la **mediana** y **medidas de posición** (percentiles) * Los **lados** inferior y superior de la caja representan el **primer y tercer cuartil**: la **altura de la caja** es igual al **rango intercuartílico** (dentro están el 50% de los datos centrales en torno a la mediana=. * La **línea gruesa** que divide la caja marca la **mediana**. * Las **líneas (bigotes)** que salen de las cajas llegan hasta el primer/último dato que no supere 1.5 veces el rango intercuartílico (diferencia entre tercer y primer cuartil). Los **puntos alejados** representan los **datos atípicos o outliers**. ] --- # Gráficos de cajas y bigotes .pull-left[ Podemos añadir, además del gráfico resumido, los propios datos con `geom_jitter()`, que nos añadirá los puntos como una especie de «gotelé aleatorio» (la altura es la de los datos pero la anchura la disemina) ```r ggplot(datos_final, aes(x = termino, y = prob, color = termino, fill = termino)) + geom_boxplot(alpha = 0.8) + geom_jitter(alpha = 0.25, size = 1.5) + scale_fill_brewer(palette = "RdBu") + scale_color_brewer(palette = "RdBu") + guides(color = "none", fill = "none") labs(fill = "Términos", y = "Probabilidad (%)", title = "Percepción de la probabilidad") ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-476-1.png" width="100%" /> ``` > $fill > [1] "Términos" > > $y > [1] "Probabilidad (%)" > > $title > [1] "Percepción de la probabilidad" > > attr(,"class") > [1] "labels" ``` ] --- # Gráficos de cajas y bigotes .pull-left[ Con `coord_flip()` podemos **invertir los ejes** ```r ggplot(datos_final, aes(x = termino, y = prob, color = termino, fill = termino)) + geom_boxplot(alpha = 0.8) + geom_jitter(alpha = 0.25, size = 1.5) + coord_flip() + scale_fill_brewer(palette = "RdBu") + scale_color_brewer(palette = "RdBu") + guides(color = "none", fill = "none") + labs(fill = "Términos", y = "Probabilidad (%)", title = "Percepción de la probabilidad") ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-478-1.png" width="100%" /> ] ] --- # Histograma, densidad y violín .pull-left[ Una opción también muy habitual son los **histogramas** (que no diagrama de barras). El histograma **segmenta en tramos el conjunto de posibles valores** y representa la frecuencia de cada uno de esos segmentos. Para pintarlos podemos hacerlo con `geom_histogram()` (por ejemplo, para las probabilidades de `termino == "Probable"`) ```r ggplot(datos_pivot %>% filter(termino == "Probable"), aes(x = prob)) + * geom_histogram(alpha = 0.4) + labs(fill = "Términos", y = "Probabilidad (%)", title = "Percepción de probabilidad") ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-480-1.png" width="100%" /> ] --- # Histograma, densidad y violín .panelset[ .panel[.panel-name[Ejercicio] 1. Sin filtrer los datos, codifica el color y el relleno en función de la variable `termino`, componiendo con `facet_wrap()` en función de dicha variable. 2. Añade `scale_fill_viridis_d()` y `scale_fill_viridis_d()` para las escalas de colores y rellenos. Elimina el nombre de las leyendas de `color` y `fill`. 3. Modifica la variable `termino` con `fct_reorder` para reordenar los términos en función de sus probabilidades. 4. Por defecto nos pinta 30 barras pero podemos indicarle que pinte más o menos, por ejemplo 10 barras con `bins = 10` dentro de `geom_histogram()`. ] .panel[.panel-name[Paso 1] .pull-left[ ```r ggplot(datos_pivot, aes(x = prob, color = termino, fill = termino)) + geom_histogram(alpha = 0.4) + * facet_wrap(~ termino, scale = "free_y") + labs(fill = "Términos", y = "Probabilidad (%)", title = "Percepción de probabilidad") ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-482-1.png" width="100%" /> ] ] .panel[.panel-name[Paso 2] .pull-left[ ```r ggplot(datos_pivot, aes(x = prob, color = termino, fill = termino)) + geom_histogram(alpha = 0.4) + * scale_fill_viridis_d() + * scale_color_viridis_d() + facet_wrap(~ termino, scale = "free_y") + guides(color = "none", * fill = "none") + labs(fill = "Términos", y = "Probabilidad (%)", title = "Percepción de probabilidad") ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-484-1.png" width="100%" /> ] ] .panel[.panel-name[Paso 3] .pull-left[ ```r datos_pivot <- datos_pivot %>% mutate(termino = * fct_reorder(termino, prob)) ggplot(datos_pivot, aes(x = prob, color = termino, fill = termino)) + geom_histogram(alpha = 0.4) + scale_fill_viridis_d() + scale_color_viridis_d() + facet_wrap(~ termino, scale = "free_y") + guides(color = "none", fill = "none") + labs(fill = "Términos", y = "Probabilidad (%)", title = "Percepción de probabilidad") ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-486-1.png" width="100%" /> ] ] .panel[.panel-name[Paso 4] .pull-left[ ```r ggplot(datos_pivot, aes(x = prob, color = termino, fill = termino)) + geom_histogram(alpha = 0.4, * bins = 10) + scale_fill_viridis_d() + scale_color_viridis_d() + facet_wrap(~ termino, scale = "free_y") + guides(color = "none", fill = "none") + labs(fill = "Términos", y = "Probabilidad (%)", title = "Percepción de probabilidad") ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-488-1.png" width="100%" /> ] ] ] --- # Histograma, densidad y violín .pull-left[ Los **histogramas** en realidad son una **aproximación discreta** de los **gráficos de densidad** (asumiendo que los intervalos se pudieran ir haciendo tan pequeños como queramos, generado una curva continua). Las densidades mejoran la robustez al histograma que puede variar mucho en función de los tramos de agregación elegidos. Basta cambiar `geom_histogram()` por `geom_density()` ```r ggplot(datos_final, aes(x = prob, color = termino, fill = termino)) + * geom_density(alpha = 0.4) + scale_fill_brewer(palette = "RdBu") + scale_color_brewer(palette = "RdBu") + guides(color = "none") + labs(fill = "Términos", y = "Probabilidad (%)", title = "Percepción de probabilidad") ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-490-1.png" width="100%" /> ] --- # Histograma, densidad y violín .pull-left[ A veces puede ser interesante **superponer las densidades**, lo cual lo podemos hacer con `geom_density_ridges()`, del paquete `{ggridges}`. ```r library(ggridges) ggplot(datos_pivot %>% mutate(termino = fct_reorder(termino, prob)), aes(y = termino, x = prob, fill = termino)) + * geom_density_ridges(alpha = 0.4) + scale_fill_viridis_d() + scale_color_viridis_d() + guides(color = "none", fill = "none") + labs(fill = "Términos", y = "Probabilidad (%)", title = "Percepción de la probabilidad") ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-492-1.png" width="100%" /> ] --- class: center, middle # CASO PRÁCTICO ## VISUALIZANDO DATOS DE NETFLIX Vamos a profundizar en la personalización de nuestro gráfico --- # Datos En esta ocasión vamos a usar los **datos de Netflix** proporcionados por [El Arte del Dato](https://elartedeldato.com/blog/como-anadir-una-anotacion-en-ggplot/), página en la que se basará esta visualización: **visualizaremos** el **número de películas y series de instituto** que se han estrenado en **Netflix en cada año**. Los datos provienen originalmente de [Kaggle](https://www.kaggle.com/shivamb/netflix-shows), ```r netflix <- read_csv('https://raw.githubusercontent.com/elartedeldato/datasets/main/netflix_titles.csv') netflix ``` ``` > # A tibble: 7,787 x 12 > show_id type title director cast country date_added release_year rating > <chr> <chr> <chr> <chr> <chr> <chr> <chr> <dbl> <chr> > 1 s1 TV Show 3% <NA> João~ Brazil August 14~ 2020 TV-MA > 2 s2 Movie 7:19 Jorge Mic~ Demi~ Mexico December ~ 2016 TV-MA > 3 s3 Movie 23:59 Gilbert C~ Tedd~ Singap~ December ~ 2011 R > 4 s4 Movie 9 Shane Ack~ Elij~ United~ November ~ 2009 PG-13 > 5 s5 Movie 21 Robert Lu~ Jim ~ United~ January 1~ 2008 PG-13 > 6 s6 TV Show 46 Serdar Ak~ Erda~ Turkey July 1, 2~ 2016 TV-MA > 7 s7 Movie 122 Yasir Al ~ Amin~ Egypt June 1, 2~ 2019 TV-MA > 8 s8 Movie 187 Kevin Rey~ Samu~ United~ November ~ 1997 R > 9 s9 Movie 706 Shravan K~ Divy~ India April 1, ~ 2019 TV-14 > 10 s10 Movie 1920 Vikram Bh~ Rajn~ India December ~ 2008 TV-MA > # ... with 7,777 more rows, and 3 more variables: duration <chr>, > # listed_in <chr>, description <chr> ``` --- # Proyecto Cuando se empieza a programar para un trabajo concreto de `R` es recomendable crearnos lo que se conoce como un **proyecto de trabajo**: en lugar de ir abriendo ventanas sueltas para programar, podemos agruparlos en proyectos, de forma que podamos acceder a ellos de forma **ordenada** <img src = "https://dadosdelaplace.github.io/courses-ECI-2022/img/crear_proyecto1.jpg" alt = "course-ECI" align = "left" width = "220" style = "margin-top: 5vh;margin-right: 0.5rem;margin-left: 0.5rem;"> <img src = "https://dadosdelaplace.github.io/courses-ECI-2022/img/crear_proyecto2.jpg" alt = "course-ECI" align = "middle" width = "220" style = "margin-top: 5vh;margin-right: 0.5rem;margin-left: 0.5rem;"> <img src = "https://dadosdelaplace.github.io/courses-ECI-2022/img/crear_proyecto3.jpg" alt = "course-ECI" align = "right" width = "250" style = "margin-top: 5vh;margin-right: 0.5rem;margin-left: 0.5rem;"> <img src = "https://dadosdelaplace.github.io/courses-ECI-2022/img/crear_proyecto4.jpg" alt = "course-ECI" align = "right" width = "250" style = "margin-top: 5vh;margin-right: 0.5rem;margin-left: 0.5rem;"> --- # Preprocesamiento Para visualizar vamos a filtrar las **películas y series de instituto**, usando la función `str_detect()` (del paquete `{stringr}`), que nos devolverá `TRUE` si detecta en la variable `description` (pasándola a mayúsculas por si acaso) el patrón de texto `"HIGH SCHOOL"`. ```r netflix_hs <- netflix %>% filter(str_detect(toupper(description), "HIGH SCHOOL")) netflix_hs ``` ``` > # A tibble: 150 x 12 > show_id type title director cast country date_added release_year rating > <chr> <chr> <chr> <chr> <chr> <chr> <chr> <dbl> <chr> > 1 s8 Movie 187 Kevin R~ Samu~ United~ November ~ 1997 R > 2 s32 Movie #FriendB~ Rako Pr~ Adip~ Indone~ May 21, 2~ 2018 TV-G > 3 s34 Movie #reality~ Fernand~ Nest~ United~ September~ 2017 TV-14 > 4 s47 Movie 1 Chance~ Adam De~ Lexi~ United~ July 1, 2~ 2014 TV-PG > 5 s48 Movie 1 Mile t~ Leif Ti~ Bill~ United~ July 7, 2~ 2017 TV-14 > 6 s56 Movie 100 Thin~ <NA> Isab~ United~ November ~ 2014 TV-Y > 7 s58 Movie 100% Hal~ Jastis ~ Anis~ Indone~ January 7~ 2020 TV-14 > 8 s148 Movie A Babysi~ Rachel ~ Tama~ United~ October 1~ 2020 TV-PG > 9 s251 Movie A Walk t~ Adam Sh~ Mand~ United~ July 1, 2~ 2002 PG > 10 s297 Movie Across T~ Julien ~ Sara~ Canada April 1, ~ 2015 TV-MA > # ... with 140 more rows, and 3 more variables: duration <chr>, > # listed_in <chr>, description <chr> ``` --- # Preprocesamiento 📝 Del conjunto original **elimina** aquellos registros de los que no tengamos su año de estreno, aquellos registros cuyo campo `date_added` esté ausente. 📝 Tras ello comprueba que efectivamente no queda ninguno ausente -- ```r netflix_filtro <- netflix_hs %>% filter(!is.na(date_added)) netflix_filtro %>% filter(is.na(date_added)) ``` ``` > # A tibble: 0 x 12 > # ... with 12 variables: show_id <chr>, type <chr>, title <chr>, > # director <chr>, cast <chr>, country <chr>, date_added <chr>, > # release_year <dbl>, rating <chr>, duration <chr>, listed_in <chr>, > # description <chr> ``` --- # Preprocesammiento Tras dicho filtro vamos a añadir el **año en el que se estrenó**, con la función `year()` de `{lubridate}`, que nos devuelve el año de una fecha concreta. Esa fecha concreta la vamos a construir con `mdy()`. ```r library(lubridate) mdy("August 26, 2016") ``` ``` > [1] "2016-08-26" ``` ```r mdy("January 13, 2015") ``` ``` > [1] "2015-01-13" ``` --- # Preprocesammiento 📝 Añade una nueva columna a nuestros datos que contenga el año de estreno en Netflix (convirtiendo primero a fecha y luego extrayendo el año). -- ```r netflix_final <- netflix_filtro %>% mutate(year = year(mdy(date_added))) ``` --- # Análisis preliminar Tras la depuración 📝 Agrupa los datos por año (que tenemos en la nueva variable `year` creada) 📝 Cuenta el número de elementos en cada año. -- ```r netflix_resumen <- netflix_final %>% * group_by(year) %>% count() %>% * ungroup() netflix_resumen ``` ``` > # A tibble: 9 x 2 > year n > <dbl> <int> > 1 2011 2 > 2 2013 1 > 3 2015 1 > 4 2016 6 > 5 2017 18 > 6 2018 28 > 7 2019 42 > 8 2020 46 > 9 2021 6 ``` --- # Diagrama de barras .panelset[ .panel[.panel-name[Ejercicio] 📝 Con estos datos ya estamos condiciones de poder hacer nuestro diagrama de barras. Realiza un diagrama de barras con el año en el eje X, el número de películas en cada una en el eje Y. ] .panel[.panel-name[Solución] .pull-left[ ```r ggplot(netflix_resumen, aes(x = year, y = n)) + * geom_col() ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-500-1.png" width="100%" /> ] ] ] --- # Diagrama de barras: color .panelset[ .panel[.panel-name[Ejercicio] 📝 Repite el gráfico indicándole el color del RELLENO de las barras sea rojo (`"red"`) ] .panel[.panel-name[Solución] .pull-left[ ```r ggplot(netflix_resumen, aes(x = year, y = n)) + * geom_col(fill = "red") ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-502-1.png" width="100%" /> ] ] ] --- # Modificando la escala de los ejes .pull-left[ Si te fijas solo nos ha mostrado algunos años en el eje X, así que le vamos a indicar la **escala concreta** que queremos en dicho eje con `scale_x_continuous()`, usando el argumento `breaks` en el que le indicaremos los valores donde queremos que «corte» el eje X (los corte serán los años guardados en `netflix_resumen$year`) ```r ggplot(netflix_resumen, aes(x = year, y = n)) + geom_col(fill = "red") + scale_x_continuous(breaks = * netflix_resumen$year) ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-504-1.png" width="100%" /> ] --- # Fechas en los ejes Cuando uno de los **ejes representa una fecha** podemos indicárselo con `scale_x_date()`, asigándole en `date_breaks` el lapso temporal que queremos en las marcas (por ejemplo, `date_breaks = '1 month'`). Como ejemplo, vamos a visualizar el número de películas y series generales estrenadas en Netflix desde el 1 de julio de 2020. 📝 Antes filtra tu conjunto original `netflix` para quedarte solo con los registros posteriroes al 1 de julio de 2020, agrupalos por fecha -- ```r netflix_julio_2022 <- netflix %>% mutate(date_added = mdy(date_added)) %>% * filter(date_added > as.Date("2020-07-01")) ``` --- # Fechas en los ejes .panelset[ .panel[.panel-name[Ejercicio] 📝 Visualiza en un diagrama de barras las películas agrupadas por fecha de `netflix_julio_2022`, con `geom_col()`, y añade `scale_x_date(date_breaks = '1 month')` ] .panel[.panel-name[Solución] .pull-left[ ```r ggplot(netflix_julio_2022 %>% group_by(date_added) %>% count(), aes(x = date_added, y = n)) + geom_col(fill = "red") + * scale_x_date(date_breaks = '1 month') ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-507-1.png" width="100%" /> ] ] ] --- # Personalizando tema .panelset[ .panel[.panel-name[Ejercicio] 📝 Lo primero que vamos a hacer para personalizar es **añadir título, subtítulo y caption**. ] .panel[.panel-name[Solución] .pull-left[ ```r ggplot(netflix_resumen, aes(x = year, y = n)) + geom_col(fill = "red") + scale_x_continuous(breaks = netflix_resumen$year) + * labs(title = "NETFLIX", subtitle = "Películas y series de instituto", caption = "Basada en El Arte del Dato (https://elartedeldato.com) | Datos: Kaggle") ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-509-1.png" width="100%" /> ] ] ] --- # Fuente propia En este caso, al ser datos de Netflix, la propia palabara de es una marca por sí misma, y quizás nos interese **usar alguna fuente de Google** para cambiar la fuente por defecto. En este caso vamos a usar la fuente de Netflix, la fuente `Bebas Neue`, y para poder usarla usaremos `font_add_google()` ```r library(sysfonts) library(showtext) font_add_google(family = "Bebas Neue", name = "Bebas Neue") showtext_auto() ``` --- # Fuente propia Tras ello vamos a **personalizar totalmente nuestro tema**. Lo primero que haremos será «resetear» el tema que podamos tener por defecto con `theme_minimal()`. Tras dicho reseteo, le indicaremos con `theme()` * `legend.position = "none"`: sin leyenda. * `plot.title = element_text(family = "Bebas Neue", color = "red", size = 50)`: le indicaremos la fuente, el color y el tamaño de nuestro título. ```r gg <- ggplot(netflix_resumen, aes(x = year, y = n)) + geom_col(fill = "red") + scale_x_continuous(breaks = netflix_resumen$year) + theme_minimal() + theme(legend.position = "none", plot.title = element_text(family = "Bebas Neue", color = "red", size = 80)) + labs(title = "NETFLIX", subtitle = "Películas y series de instituto", caption = "Basada en El Arte del Dato (https://elartedeldato.com) | Datos: Kaggle") gg ``` --- # Fuente propia <img src="index_files/figure-html/unnamed-chunk-512-1.png" width="100%" /> --- # Color de fondo .pull-left[ Tras cambiar la fuente del título vamos a indicarle que el fondo del gráfico sea todo negro. ```r gg <- gg + theme(panel.background = element_rect(fill = "black"), plot.background = element_rect(fill = "black", color = "black")) gg ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-514-1.png" width="100%" /> ] --- # Grid .pull-left[ También vamos a personalizar el grid horizontal (el que marca las alturas del eje y), indicándole color y tamaño. ```r gg <- gg + theme(panel.grid.major.y = element_line(size = 0.1, color = "white"), panel.grid.minor.y = element_blank(), panel.grid.major.x = element_blank(), panel.grid.minor.x = element_blank()) gg ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-516-1.png" width="100%" /> ] --- # Fuentes de textos .pull-left[ Vamos a personalizar también la **fuente del subtítulo y caption** y los textos de los ejes. ```r font_add_google(family = "Permanent Marker", name = "Permanent Marker") showtext_auto() gg <- gg + theme(plot.subtitle = element_text(family = "Permanent Marker", size = 21, color = "white"), plot.caption = element_text(family = "Permanent Marker", color = "white", size = 19), axis.text = element_text(size = 15, family = "Permanent Marker", color = "white")) gg ``` ] .pull-right[ <img src="index_files/figure-html/unnamed-chunk-518-1.png" width="100%" /> ] --- # Márgenes Vamos a darle un poco de aire **añadiendo márgenes** ```r gg <- gg + theme(plot.margin = margin(t = 4, r = 4, b = 4, l = 8, "pt")) gg ``` --- # Gráfico casi final <img src="index_files/figure-html/unnamed-chunk-520-1.png" width="50%" style="display: block; margin: auto;" /> --- # Anotaciones Por último con `annotate()` podemos añadir anotaciones al gráfico, por ejemplo, escribiendo el mes de enero en la última barra para remarcar que solo llega hasta enero de 2021, con una fina curva como «flecha». ```r gg <- gg + annotate("text", label = "(hasta enero)", x = 2021, y = 11, hjust = 0.3, vjust = 0, family = "Permanent Marker", size = 5, color='white', angle = 20) + annotate("curve", x = 2021, y = 9, xend = 2021, yend = 5, color = "white") gg ``` --- # Tachán <img src="index_files/figure-html/unnamed-chunk-522-1.png" width="50%" style="display: block; margin: auto;" /> --- class: inverse center middle # Bloque IV: Introducción a Rmarkdown  --- # Qué es R Markdown? 1. ["Un marco de creación para la ciencia de datos."](https://rmarkdown.rstudio.com/lesson-1.html) (✔️) 1. [Un formato de documento (`.Rmd`).](https://bookdown.org/yihui/rmarkdown/) (✔️) 1. [Un paquete R llamado `rmarkdown`.](https://rmarkdown.rstudio.com/docs/) (✔️) 1. ["Un formato de archivo para hacer documentos dinámicos con R.."](https://rmarkdown.rstudio.com/articles_intro.html) (✔️) 1. ["Una herramienta para integrar prosa, código y resultados."](https://r4ds.had.co.nz/communicate-intro.html) (✔️) 1. ["Un documento computacional."](http://radar.oreilly.com/2011/07/wolframs-computational-documen.html) (✔️) 1. Hechicería. (🧙♂️) --- background-image: url(img/rmarkdown_wizards.png) background-size: contain --- class: inverse, middle, center # Por qué R-Markdown? --- class: bottom, center background-image: url(https://media.giphy.com/media/11fDMHAzihB8D6/source.gif) # `Crisis de reproducibilidad?` --- class: middle, center # Cambia tu modelo mental .pull-left[ ### Source ↔ output <img src="img/word.png" width="50%" /> ] .pull-right[ ### Source → output <img src="img/rmd-file.png" width="50%" /> ] --- class: middle, center # Cambia tu modelo mental .pull-left[ ### Source ↔ output <img src="img/haba-elise.jpg" width="50%" /> ] .pull-right[ ### Source → output <img src="img/doll.png" width="50%" /> ] --- class: middle, inverse, center # Misma ~~Muñeca~~ Fuente -- # Diferente ~~Vestido~~ Salida --- class: middle, center # `html_document` <img src="img/doll.png" width="40%" /> --- class: middle, center # Qué hay adentro?  --- name: card0 background-image: url(img/card0.png) background-size: contain --- name: card1 background-image: url(img/card1.png) background-size: contain --- # Guarda las opciones de la salida en el YAML .pull-left[ ```yaml --- author: Your name here title: Your title here output: html_document --- ``` ```yaml --- author: Your name here title: Your title here output: html_document: toc: true toc_float: true theme: flatly --- ``` ] .pull-right[ <img src="img/orchestra.jpg" width="75%" style="display: block; margin: auto;" /> ] --- background-image: url(img/Single-rmd.png) background-size: contain --- background-image: url(img/Single-rmd1.png) background-size: contain --- background-image: url(img/Single-rmd2.png) background-size: contain --- background-image: url(img/Single-rmd3.png) background-size: contain --- class: middle center background-image: url(img/Single-knit1.png) background-size: contain -- <img src="img/Single-knit2.png" style=" position: absolute; width: 91%; top: -53px; right: 5px; "> -- <img src="img/Single-knit3.png" style=" position: absolute; width: 97%; top: -45px; right: -51px; "> --- # Formatos de salida .center[ <a href="https://rmarkdown.rstudio.com/docs/reference/index.html#section-output-formats" target="_blank"><img src="img/rmdbase-formats.png" width="40%" /></a> ] https://rmarkdown.rstudio.com/docs/reference/index.html#section-output-formats --- class: middle, center # Formatos de extensión de salida <img src="https://raw.githubusercontent.com/rstudio/hex-stickers/master/PNG/flexdashboard.png" width="32%" /><img src="https://raw.githubusercontent.com/rstudio/hex-stickers/master/PNG/bookdown.png" width="32%" /><img src="https://raw.githubusercontent.com/rstudio/hex-stickers/master/PNG/xaringan.png" width="32%" /> --- # <center>Headers</center> -- .pull-left[ ```markdown # HEADER 1 ## HEADER 2 ### HEADER 3 #### HEADER 4 ##### HEADER 5 ###### HEADER 6 ``` ] -- .pull-right[ # HEADER 1 ## HEADER 2 ### HEADER 3 #### HEADER 4 ##### HEADER 5 ###### HEADER 6 ] ---  --- # <center>Text</center> -- .pull-left[ ```markdown Childhood **vaccines** are one of the _great triumphs_ of modern medicine. ``` ] -- .pull-right[ Childhood **vaccines** are one of the _great triumphs_ of modern medicine. ] --- class: top # <center>Lists</center> -- .pull-left[ ```markdown Indeed, parents whose children are vaccinated no longer have to worry about their child's death or disability from: - whooping cough, - polio, - diphtheria, - hepatitis, or - a host of other infections. Vaccines are the most cost-effective health care interventions there are. We have three new, extremely effective vaccines to roll out in developing countries: 1. pneumonia 1. rotavirus 1. meningitis A ``` ] -- .pull-right[ Indeed, parents whose children are vaccinated no longer have to worry about their child's death or disability from: - whooping cough, - polio, - diphtheria, - hepatitis, or - a host of other infections. Vaccines are the most cost-effective health care interventions there are. We have three new, extremely effective vaccines to roll out in developing countries: 1. pneumonia 1. rotavirus 1. meningitis A ] --- # <center>Images</center> -- ```markdown Edificio E56 - Universidad del Valle  ``` -- Edificio E56 - Universidad del Valle  --- # <center>Links</center> -- ```markdown [Photo](http://industrial.univalle.edu.co/images/la_escuela/presentacion/ESCUELA_20181001.jpg) Edificio E56 - Universidad del Valle  ``` -- [Photo](http://industrial.univalle.edu.co/images/la_escuela/presentacion/ESCUELA_20181001.jpg) Edificio E56 - Universidad del Valle  --- # Chunk Una vez que controlamos como poner títulos, subtítulos y cabeceras y texto, podemos incluir análisis y resultados de R en el informe o tablero. Para ello debemos escribir el codigo de R entre una cabecera y pie que permite interpretar la sintáxis de `R` incluyendo un trozo o `chunk`: .pull-left[ ```` ```{r} x <- 3 + 4 ``` ```` Este es un ejemplo de **chunk** ] .pull-right[ <img src="img/chunk.png" width="50%" /> ] --- # Opciones de los chunk La salida de chunk se puede personalizar con las opciones de a traves de argumentos establecidos.En la Guía de referencia de R Markdown se encuentra una lista completa de las opciones de los chunk. Los argumentos más comunes son los siguientes: * **include = FALSE** evita que el código y los resultados aparezcan en el archivo terminado. R Markdown todavía ejecuta el código en el fragmento, y los resultados pueden ser utilizados por otros fragmentos. echo = FALSE impide el código, pero no aparecen los resultados en el archivo terminado. Esta es una forma útil de incrustar figuras. * **message = FALSE** evita que los mensajes generados por el código aparezcan en el archivo terminado. * **warning = FALSE** evita que las advertencias generadas por el código aparezcan en el final. * **fig.cap = "..."** agrega un título a los resultados gráficos. --- # message / warning  --- # message / warning .pull-left[ ```` ```{r message=FALSE, warning=FALSE} library(tidyverse) ``` ```` ] .pull-right[ ```r library(tidyverse) ``` ] --- class: center, middle # Websites! --- class: middle .left-column[ # 🔗 ] .right-column[ <a href="https://rmarkdown.rstudio.com/" target="_blank"><img src="img/rmarkdown-site.png" width="80%" /></a> ] .footnote[https://rmarkdown.rstudio.com/] --- class: middle .left-column[ # 🔗 ] .right-column[ <a href="https://rmarkdown.rstudio.com/docs/" target="_blank"><img src="img/rmarkdowndocs-site.png" width="80%" /></a> ] .footnote[https://rmarkdown.rstudio.com/docs/] --- class: middle .left-column[ # 🎯 ] .right-column[ <a href="https://community.rstudio.com/c/R-Markdown" target="_blank"><img src="img/rmarkdowncommunity-site.png" width="80%" /></a> ] .footnote[https://community.rstudio.com/c/R-Markdown] --- class: middle .left-column[ # 🤖 ] .right-column[ <a href="https://rmarkdown.rstudio.com/lesson-6.html" target="_blank"><img src="https://raw.githubusercontent.com/rstudio/rmarkdown/gh-pages/lesson-images/params-1-hawaii.png" width="80%" /></a> ] .footnote[https://bookdown.org/yihui/rmarkdown/parameterized-reports.html,<br> https://rmarkdown.rstudio.com/lesson-6.html] --- class: inverse, middle, center # Thank you!